Análisis de datos en R
¡Bienvenidos a R!
Dejad vuestras hojas de cálculo a un lado
¡Buenas!
Correo: javalv09@ucm.es. Despacho: 722 (3ª planta).

Javier Álvarez Liébana, de Carabanchel (Bajo).
Licenciado en Matemáticas (UCM). Doctorado en estadística (UGR).
Encargado de la visualización y análisis de datos covid del Principado de Asturias (2021-2022).
Miembro de la Sociedad Española de Estadística e IO y la Real Sociedad Matemática Española.
Objetivos
.png)
Quitarnos el miedo a los errores en programación → a programar se aprende programando
Entender los conceptos básicos de R desde cero → aprender a abstraer ideas y algoritmos
Utilidad de programar → flujos de trabajo reproducibles, transparentes y mantenibles
Introducción al análisis y preprocesamiento de datos →
{tidyverse}Adquirir habilidades en la visualización de datos →
{ggplot2}Introducción al modelo de técnicas Machine Learning →
{tidymodels}
Evaluación
- Asistencia. La asistencia se valorará muy positivamente así como la participación en clase.
- Evaluación. A lo largo del curso se han planteado 5 entregas individuales, así como una entrega final grupal (entre 2 y 4 personas) donde se deberá presentar el análisis realizado de un caso real.
- Nota mínima. Para no ir al examen final se deberá obtener al menos un 4/10 en cada entrega o una media ponderada superior al 6/10. Se deberá obtener además en la entrega grupal una nota superior a 6-6.5-7/10 (según 2-3-4 personas)
- Examen final. Cualquier alumno podrá presentarse a un examen final, siendo la valoración del mismo el 100% de su nota (perdiendo la evaluación continua).
Planificación: clase 1
Materiales
- Diapositivas: las diapositivas que usaremos en el aula a lo largo del curso, estructuradas por clases, estarán disponibles y actualizadas en https://javieralvarezliebana.es/R-ayuntamiento/diapos
En el menú de las diapositivas (abajo a la izquierda) tienes una opción para descargarlas en pdf en Tools (consejo: no lo hagas hasta el final del curso ya que irán modificándose)
Material: scripts de cada tema y materiales extras
Resúmenes de paquetes: chuletas de los paquetes en formato .pdf
Clase 1: primeros pasos
Instalando R y RStudio. Primeros pasos. Scripts y proyectos
Requisitos
Para el curso los únicos requisitos serán:
Conexión a internet (para la descarga de algunos datos y paquetes).
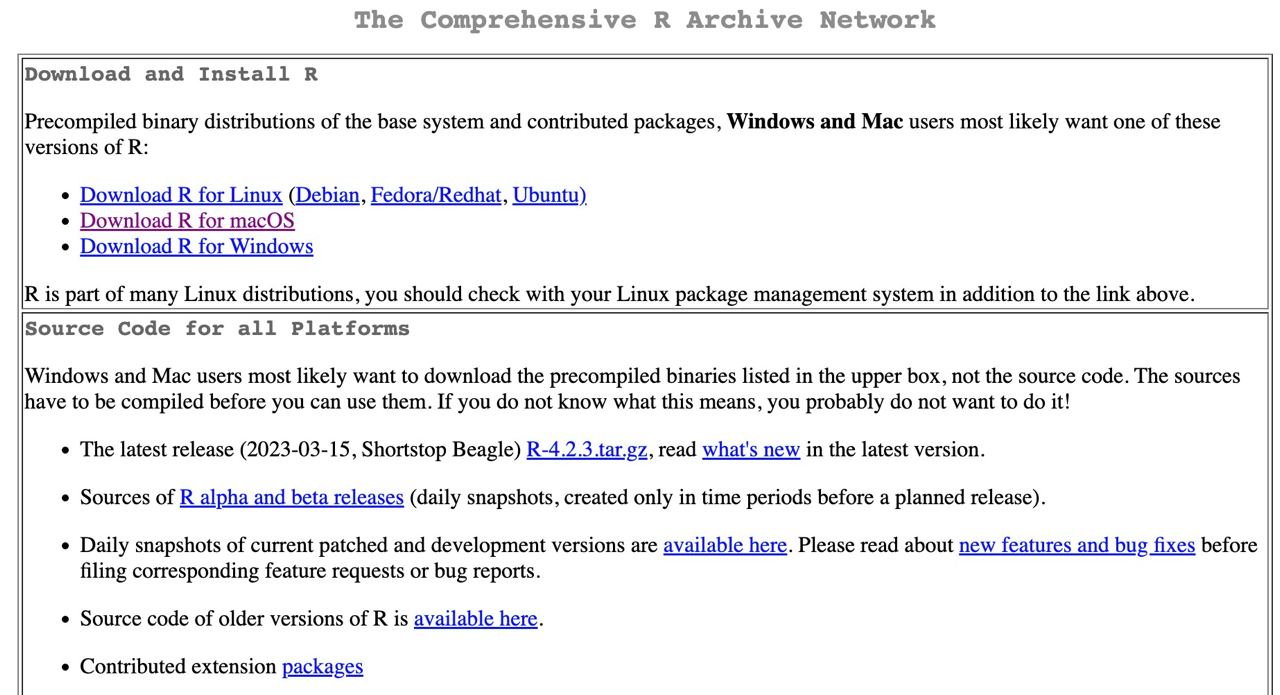
Instalar R: será nuestro lenguaje. La descarga la haremos (gratuitamente) desde https://cran.r-project.org/
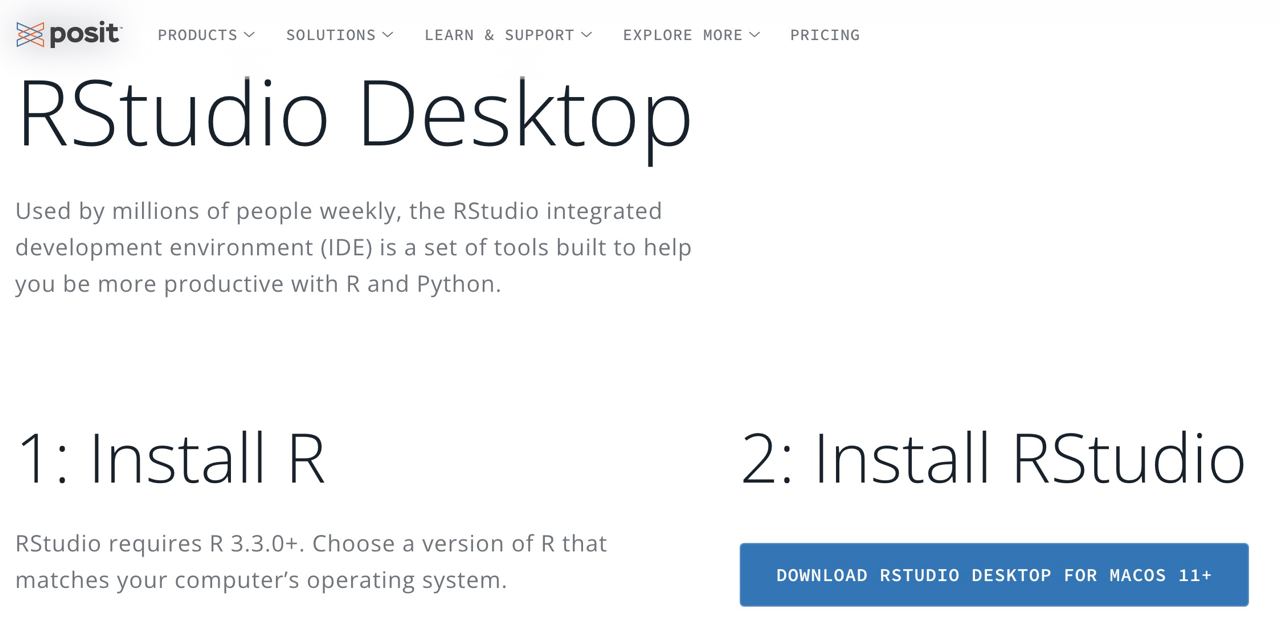
- Instalar RStudio desde https://posit.co/download/rstudio-desktop/

Programaremos como escribimos (castellano, por ejemplo) → R es lenguaje
- Necesitaremos una gramática (
R)
- Y un entorno, por ejemplo un Word (
RStudio), para escribirlo
Instalación de R
El lenguaje R será nuestra gramática y ortografía (nuestras reglas de juego)
Paso 1: entra en https://cran.r-project.org/ y selecciona tu sistema operativo.
Paso 2: para Mac basta con que hacer click en el archivo .pkg, y abrirlo una vez descargado. Para sistemas Windows, debemos clickar en install R for the first time y después en Download R for Windows. Una vez descargado, abrirlo como cualquier archivo de instalación.
Paso 3: abrir el ejecutable de instalación.
Advertencia
Siempre que tengas que descargar algo de CRAN (ya sea el propio R o un paquete), asegúrate de tener conexión a internet.
Primera operación
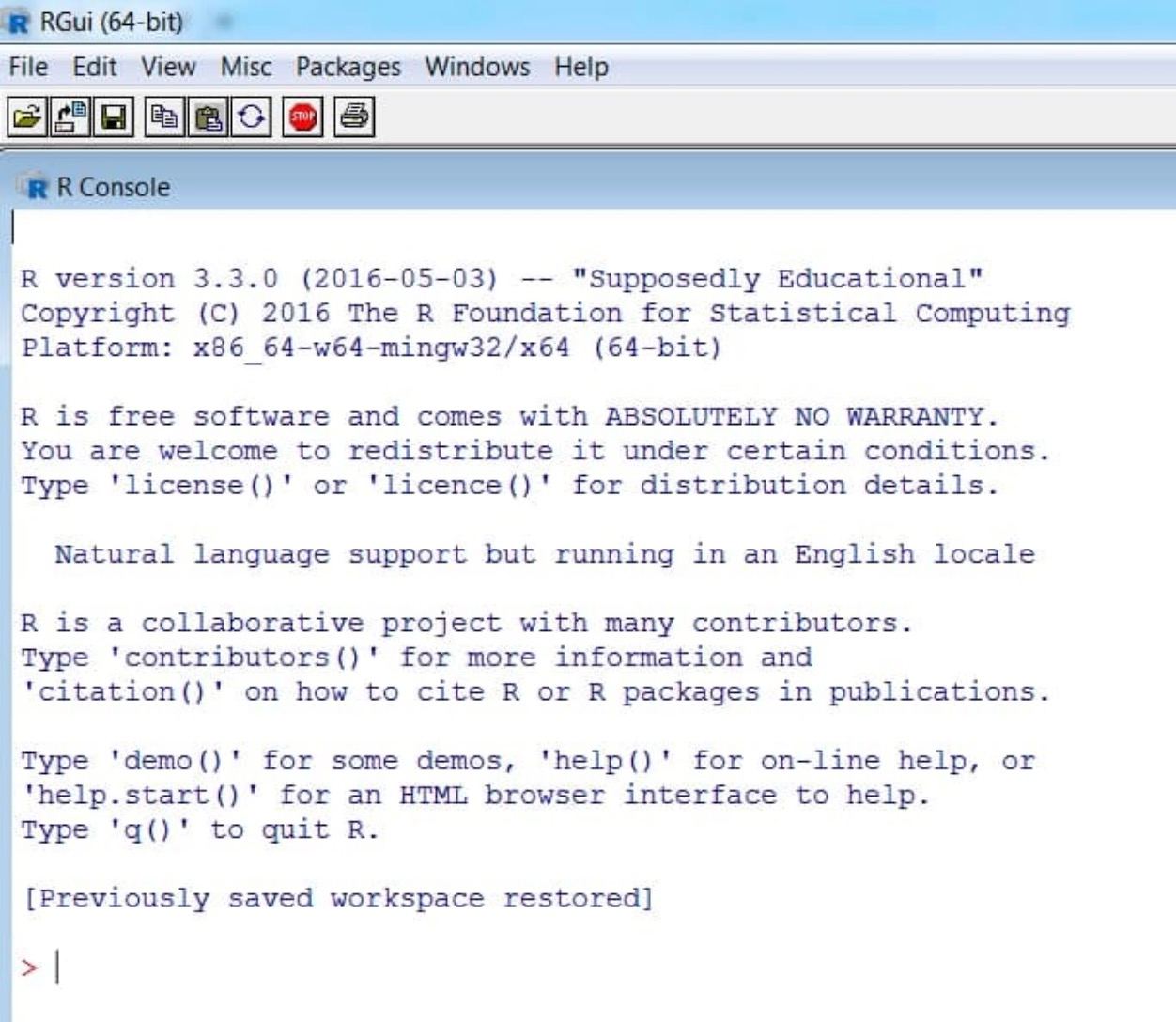
Para comprobar que se ha instalado correctamente, tras abrir R, deberías ver una pantalla blanca similar a esta.
Esa «pantalla blanca» se llama consola y podemos hacer un primer uso de ella como una calculadora.
Idea: a una variable llamada a le asignaremos el valor 1 (escribiremos el código de abajo en la consola y daremos «enter»)
Primera operación
Para comprobar que se ha instalado correctamente, tras abrir R, deberías ver una pantalla blanca similar a esta.
Esa «pantalla blanca» se llama consola y podemos hacer un primer uso de ella como una calculadora.

Idea: definiremos otra variable llamada b y le asignaremos el valor 2
Fíjate que…
En R usaremos <- como una flecha: la variable a la izquierda de dicha flecha le asignamos el valor que hay a la derecha (por ejemplo, a <- 1)
Primera operación
Para comprobar que se ha instalado correctamente, tras abrir R, deberías ver una pantalla blanca similar a esta.
Esa «pantalla blanca» se llama consola y podemos hacer un primer uso de ella como una calculadora.

Idea: haremos la suma a + b y nos devolverá su resultado
Instalación de R Studio
RStudio será el Word que usaremos para escribir (lo que se conoce como un IDE: entorno integrado de desarrollo).
Paso 1: entra la web oficial de RStudio (ahora llamado Posit) y selecciona la descarga gratuita.
Paso 2: selecciona el ejecutable que te aparezca acorde a tu sistema operativo.
Paso 3: tras descargar el ejecutable, hay que abrirlo como otro cualquier otro y dejar que termine la instalación.
Organización de RStudio
Al abrir RStudio seguramente tengas tres ventanas:
- Consola: es el nombre para llamar a la ventana grande que te ocupa buena parte de tu pantalla. Prueba a escribir el mismo código que antes (la suma de las variables) en ella. La consola será donde ejecutaremos órdenes y mostraremos resultados.
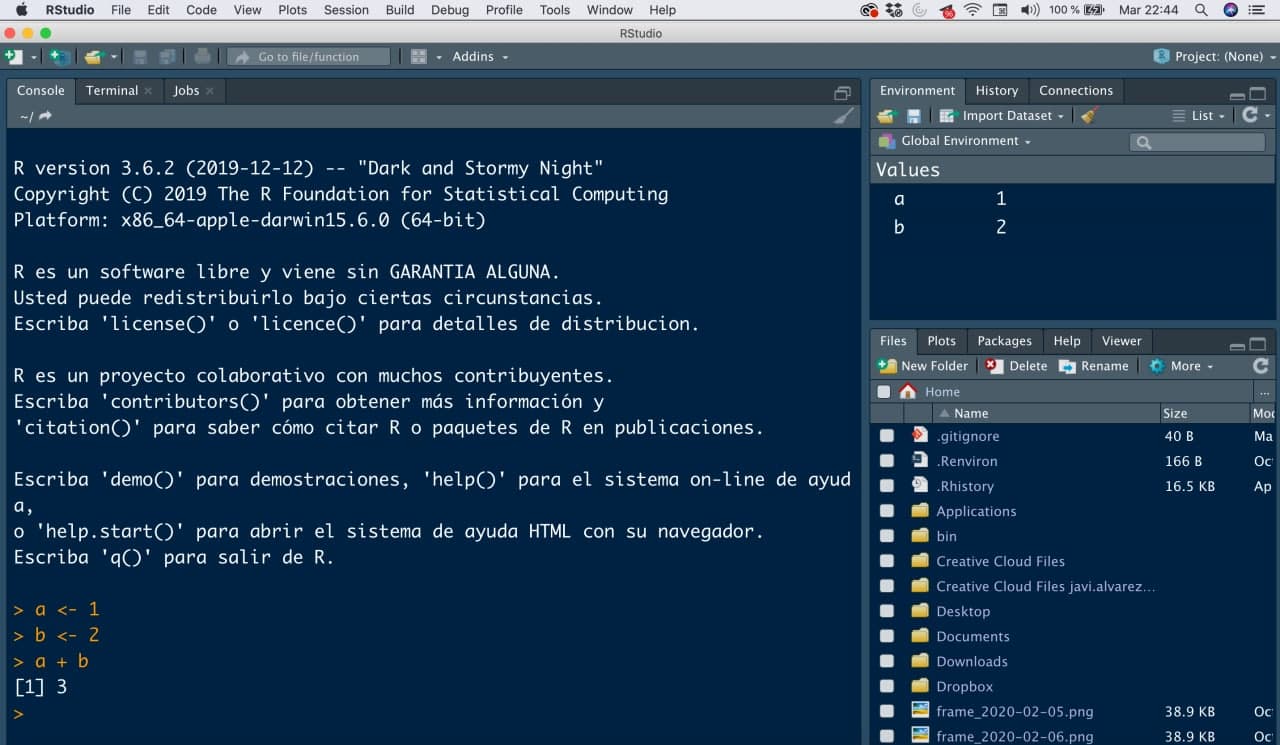
Organización de RStudio
Al abrir RStudio seguramente tengas tres ventanas:
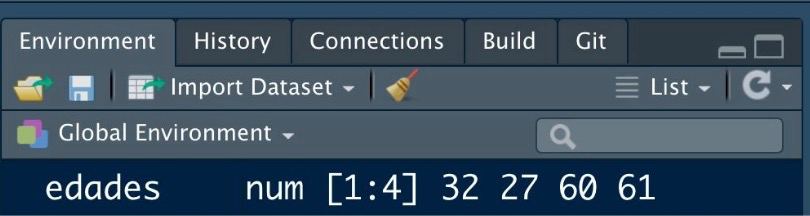
- Environment: la pantalla pequeña (puedes ajustar los márgenes con el ratón a tu gusto) que tenemos en la parte superior derecha. Nos mostrará las variables que tenemos definidas.
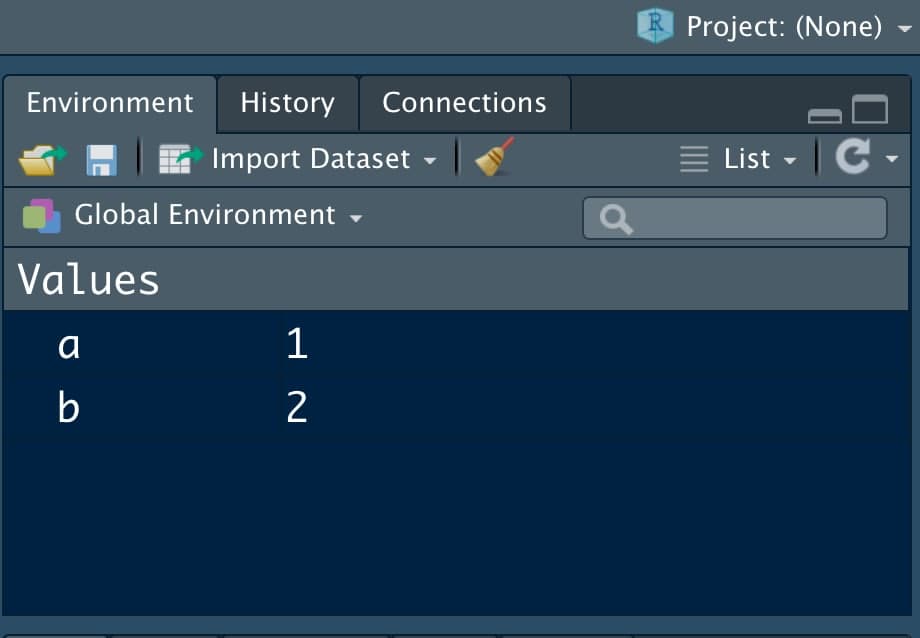
Organización de RStudio
Al abrir RStudio seguramente tengas tres ventanas:
- Panel multiusos: la ventana que tenemos en la parte inferior derecha no servirá para buscar ayuda de funciones, además de para visualizar gráficos.
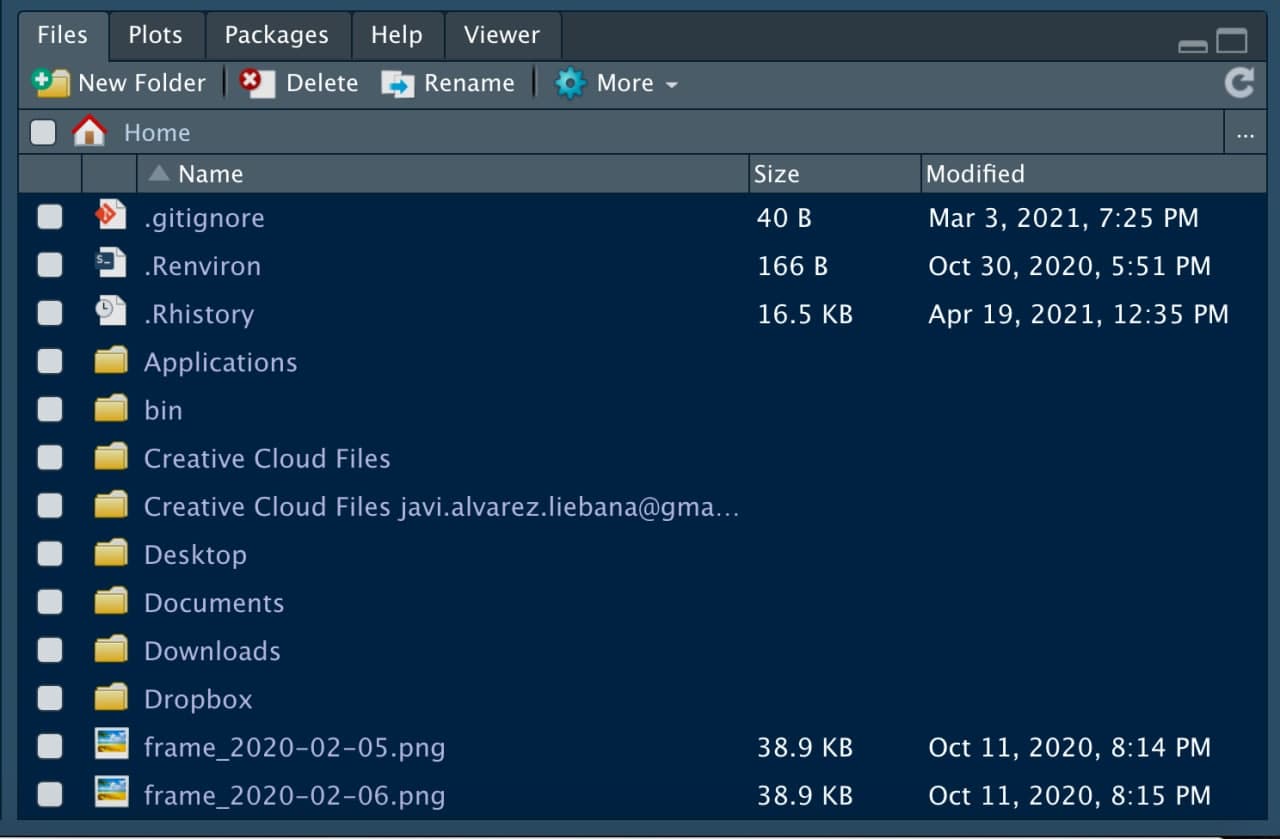
¿Qué es R? ¿Por qué R?

¿Qué es R? ¿Por qué R?
R es el lenguaje estadístico por excelencia, creado por y para estadísticos/as, con 5 ventajas fundamentales frente a Excel:
Lenguaje de programación: la obviedad → análisisreplicables
Gratuito: la filosofía de la comunidad de
Res el compartir código bajo copyleft →uso ético de dinero públicoSoftware libre: no solo es gratis sino que permite acceder libremente a código ajeno, incluso al propio código fuente →flexibilidad y transparencia
Lenguaje modular: hemos instalado lo mínimo, pero existen códigos de otras personas que podemos reusar (casi 20 000 paquetes) →ahorro de tiempo
Lenguaje de alto nivel: facilita la programación (como Python) →menor curva de aprendizaje
¿Qué es R? ¿Por qué R?
¿Por qué programar?
Automatizar → te permitirá automatizar tareas recurrentes.
Replicabilidad → podrás replicar tu análisis siempre de la misma manera.
Flexibilidad → podrás adaptar el software a tus necesidades.
Transparencia → ser auditado por la comunidad.

Idea fundamental: paquetes
Una de las ideas claves de R es el uso de paquetes: códigos que otras personas han implementado para resolver un problema
Idea fundamental: paquetes
Una vez instalado, hay dos manera de usar un paquete (traerlo de la estantería)
- Paquete entero: con
library(), usando el nombre del paquete sin comillas, cargamos en la sesión todo el libro
Te vas equivocar
Durante tu aprendizaje va a ser muy habitual que las cosas no salgan a la primera → te vas equivocar. No solo será importante asumirlo sino que es importante leer los mensajes de error para aprender de ellos.
- Mensajes de error: precedidos de «Error in…» y serán aquellos fallos que impidan la ejecución
Scripts (documentos .R)
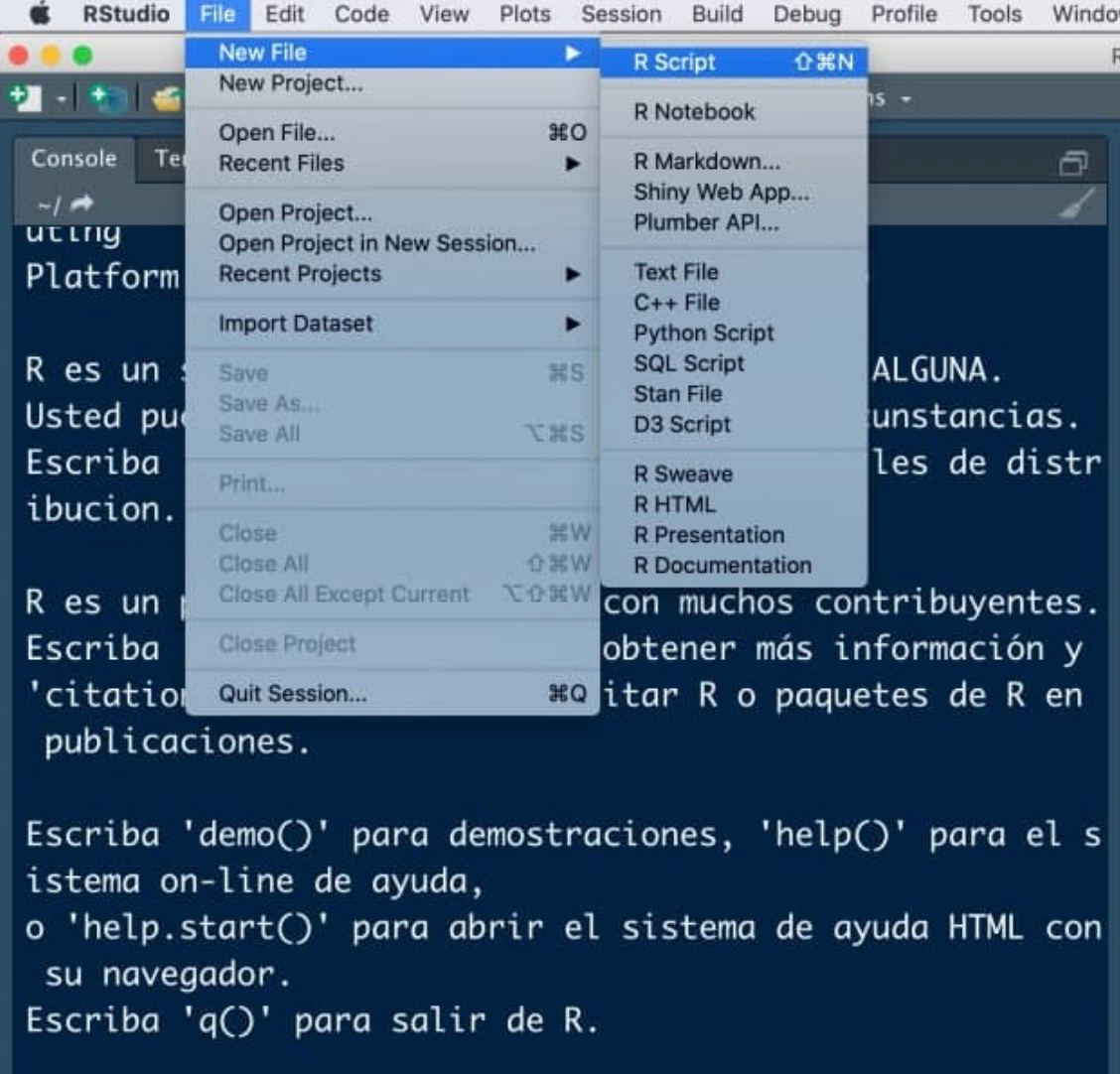
Un script será el documento en el que programamos, nuestro archivo .doc (aquí con extensión .R) donde escribiremos las órdenes. Para abrir nuestro primero script, haz click en el menú en File < New File < R Script.
Cuidado
Es importante no abusar de la consola: todo lo que no escribas en un script, cuando cierres, lo habrás perdido.
Ejecutando el primer script
Ahora tenemos una cuarta ventana: la ventana donde escribiremos nuestros códigos. ¿Cómo ejecutarlo?
- Escribimos el código a ejecutar.
- Guardamos el archivo .R haciendo click en
Save current document.
- El código no se ejecuta salvo que se lo indiquemos. Tenemos tres opciones:
- Copiar y pegar en consola.
- Seleccionar líneas y
Ctrl+Enter - Activar Source on save a la derecha de guardar: no solo guarda sino que ejecuta el código completo.
💻 Tu turno
Ejecuta tu primer script: crea un script de cero, programa lo indicado debajo y ejecútalo (de las 3 maneras posibles)
📝 Añade debajo otra línea para definir una variable b con el valor 5. Tras ello múltiplica ambas variables
📝 Modifica el código inferior para definir dos variables c y d, con valores 3 y -1. Tras ello divide las variables.
📝 Asigna un valor positivo a x y calcula su raíz cuadrada; asigna otro negativo y y calcula su valor absoluto con la función abs().
Toma nota
Comandos como sqrt(), abs() o max() son lo que llamamos funciones: líneas de código que hemos «encapsulado» bajo un nombre, y dado unos argumentos de entrada, ejecuta las órdenes (una especie de atajo).
Sé organizado: proyectos
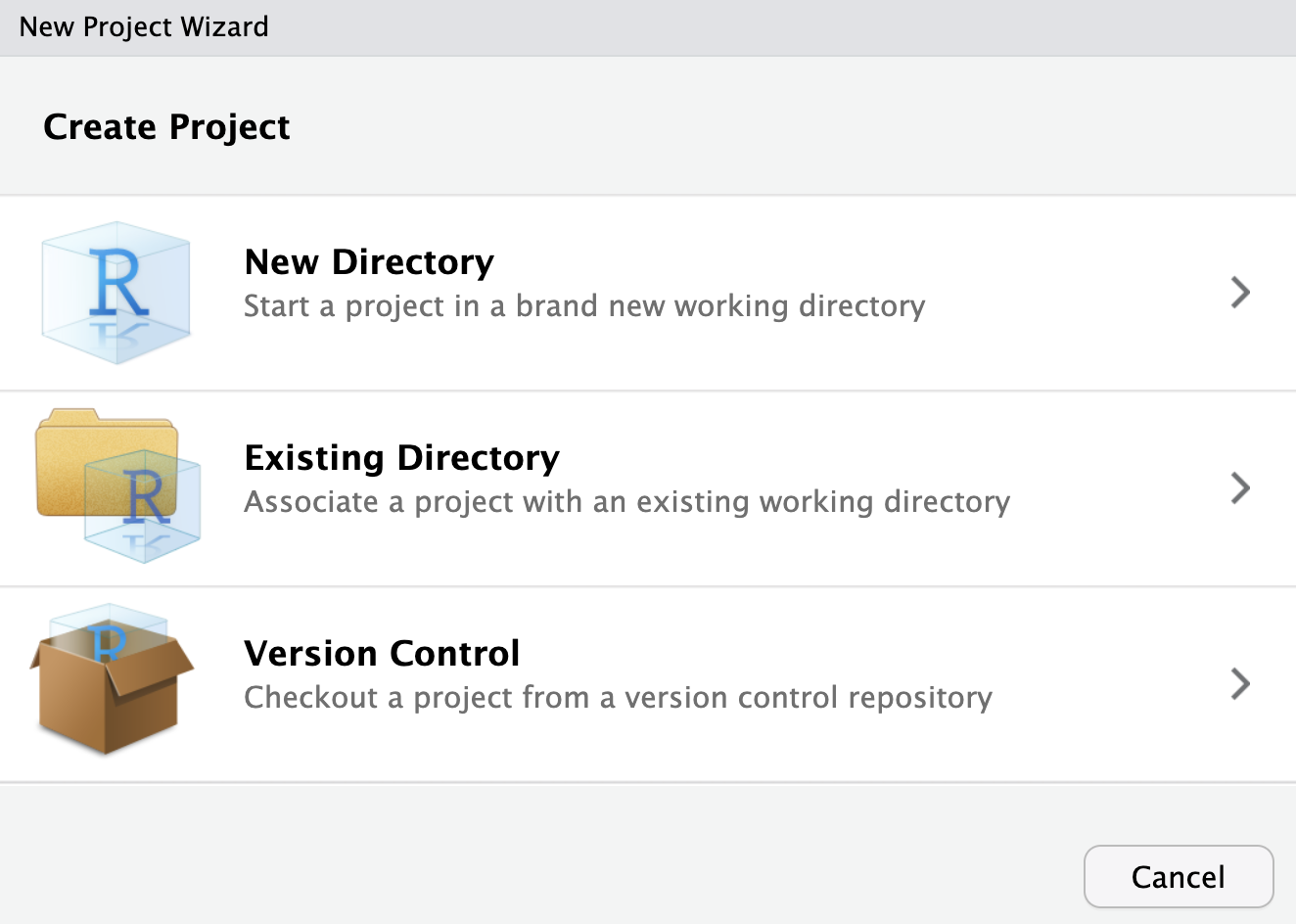
De la misma manera que en el ordenador solemos trabajar de manera ordenada por carpetas, en RStudio podemos hacer lo mismo para trabajar de manera eficaz creando proyectos.
Un proyecto será una «carpeta» dentro de RStudio, de manera que nuestro directorio raíz automáticamente será la propia carpeta de proyecto (pudiendo pasar de un proyecto a otro con el menu superior derecho).
Podemos crear uno en una carpeta nueva o en una carpeta ya existente.
Filosofía: de la CELDA a la TABLA
¿Qué tipo de dato podemos tener en cada celda de una tabla?
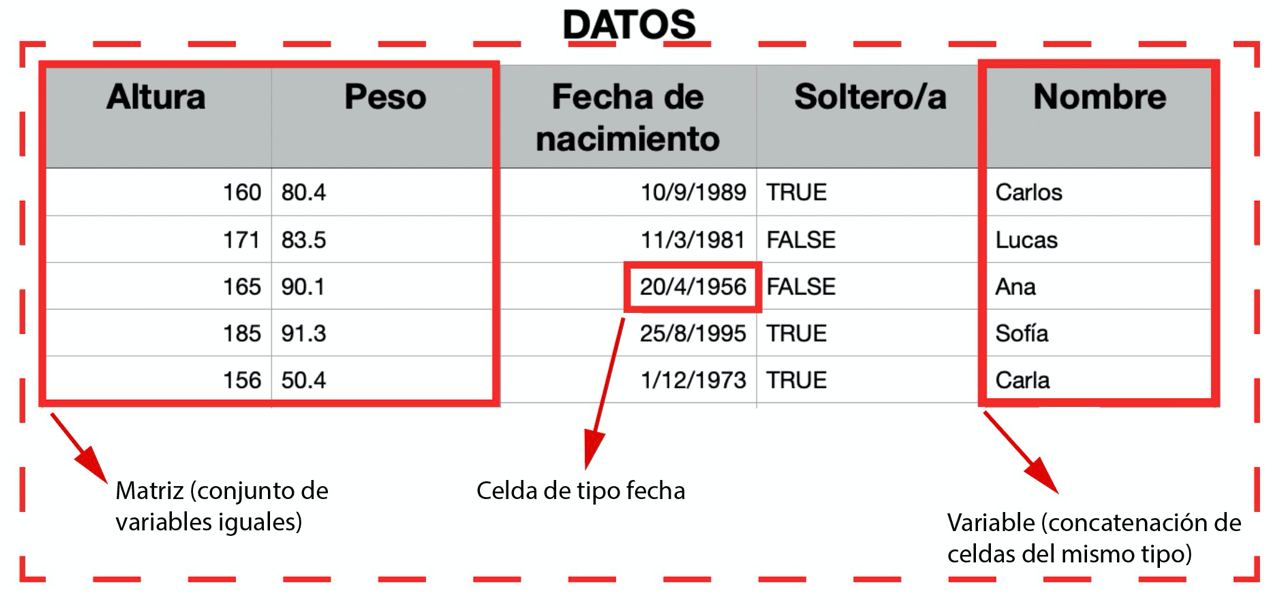
- Celda: dato individual de un tipo concreto.
- Variable: concatenación de valores del mismo tipo (vectores).
- Matriz: concatenación de variables del mismo tipo y longitud.
- Tabla: concatenación de variables de distinto tipo pero igual longitud
- Lista: concatenación de variables de distinto tipo y distinta longitud
Clase 1: primeros datos y vectores
¿Qué tipos de celdas (datos) existen? Concatenando celdas: vectores
De la CELDA a la TABLA
¿Qué tipo de dato podemos tener en cada celda de una tabla?

- Celda: dato individual de un tipo concreto.
- Variable: concatenación de valores del mismo tipo (vectores).
- Matriz: concatenación de variables del mismo tipo y longitud.
- Tabla: concatenación de variables de distinto tipo pero igual longitud
- Lista: concatenación de variables de distinto tipo y distinta longitud
Celdas: tipos de datos
¿Existen variables más allá de los números?
Piensa por ejemplo en los datos guardados de una persona:
- A la pregunta «¿estás matriculado en la Facultad?» la respuesta será lo que llamamos una variable lógica (
TRUEsi está matriculado oFALSEen otro caso).
- Su fecha de nacimiento será precisamente eso, una fecha.
Variables numéricas
El dato más sencillo (ya lo hemos usado) serán las variables numéricas
Variables numéricas
Con las variables numéricas podemos realizar las operaciones aritméticas de una calculadora: sumar (+)…
Variables de texto
Imagina que además de la edad de una persona queremos guardar su nombre: ahora la variable será de tipo character
Las cadenas de texto son un tipo con el que obviamente no podremos hacer operaciones aritméticas (sí otras operaciones como pegar o localizar patrones).
Recuerda que…
Las variables de tipo texto (character o string) van SIEMPRE entre comillas: no es lo mismo TRUE (valor lógico, binario) que "TRUE" (texto).
Primera función: paste
En R llamaremos función a un trozo de código encapsulado bajo un nombre, y que depende de unos argumentos de entrada. Nuestra primera función será paste(): dadas dos cadenas de texto nos permite pegarlas.
Primera función: paste
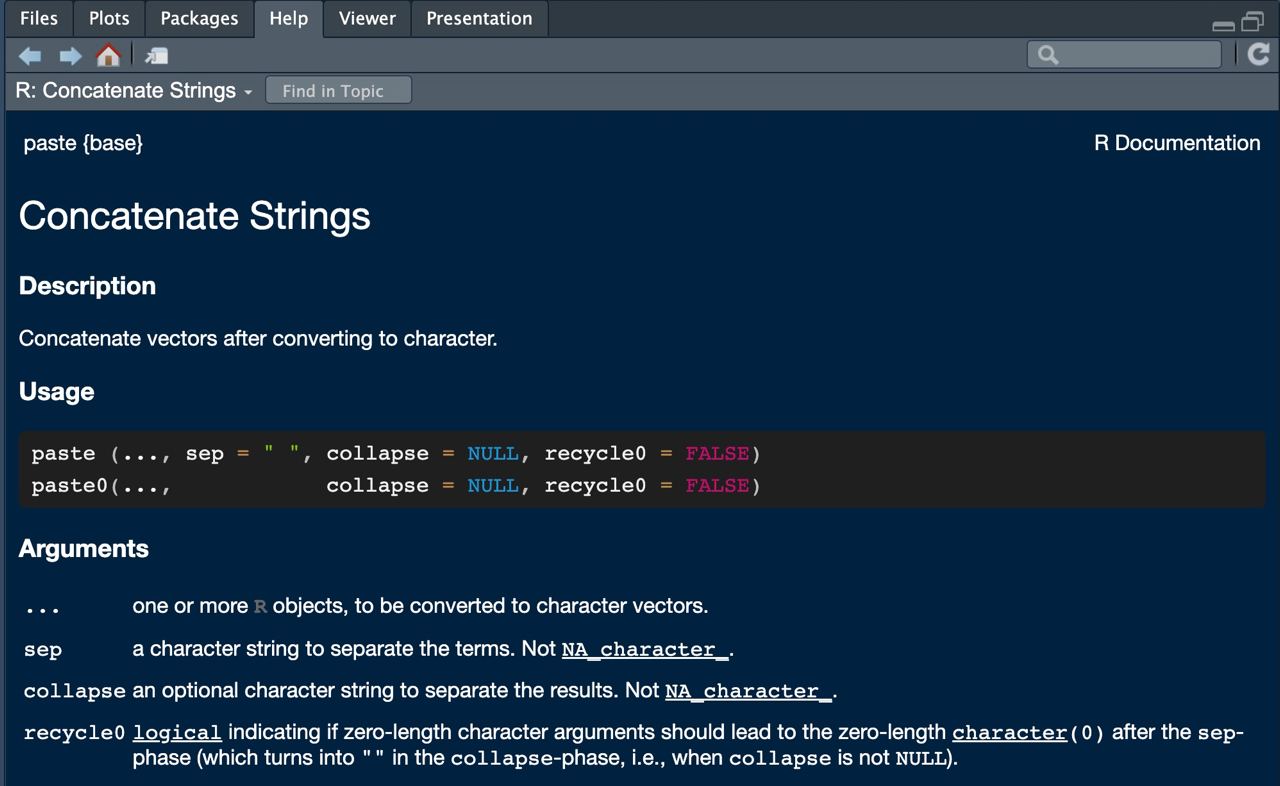
¿Cómo saber qué argumentos necesita una función? Escribiendo en consola ? paste te aparecerá una ayuda en el panel multiusos.
En dicha ayuda podrás ver en su cabecera que argumentos ya tiene asignados por defecto la función
Funciones: argumentos por defecto
Es muy importante entender el concepto de argumento por defecto de una función en R: es un valor que la función usa pero a veces podemos no ver porque ya tiene un valor asignado.
[1] "Javier Álvarez"[1] "Javier Álvarez"Toma nota
El operador = lo reservaremos para asignar argumentos dentro de funciones. Para todas las demás asignaciones usaremos <-
Primer paquete: glue
Una forma más intuitiva de trabajar con textos es usar el paquete {glue}: lo primero que haremos será «comprar el libro» (si nunca lo hemos hecho). Tras ello cargamos el paquete
Con la función glue() de dicho paquete podemos usar variables dentro de cadenas de texto. Por ejemplo, «la edad es de … años», donde la edad está guardada en una variable.
Variables lógicas
Otro tipo fundamental serán las variables lógicas o binarias (dos valores):
TRUE: verdadero guardado internamente como un 1.FALSE: falso guardado internamente como un 0.
Variables lógicas
Como veremos en breve, las variables lógicas en realidad puede tomar un tercer valor: NA o dato ausente, representando las siglas de not available, y será muy habitual encontrarlo dentro de una base de datos.
Condiciones lógicas
Los valores lógicos suelen ser resultado de evaluar condiciones lógicas. Por ejemplo, imaginemos que queremos comprobar si una persona se llama Javi.
Con el operador lógico == preguntamos sí lo que tenemos guardado a la izquierda es igual que lo que tenemos a la derecha: es una pregunta
Fíjate que…
No es lo mismo <- (asignación) que == (estamos preguntando, es una comparación lógica).
Condiciones lógicas
Además de las comparaciones «igual a» frente «distinto», también comparaciones de orden como <, <=, > o >=.
¿Tiene la persona menos de 32 años?
Variables de fecha
Un tipo de datos muy especial: los datos de tipo fecha.
Parece una simple cadena de texto pero debería representar un instante en el tiempo. ¿Qué debería suceder si sumamos un 1 a una fecha?
Las fechas NO pueden ser texto: debemos convertir la cadena de texto a fecha.
Variables de fecha
Una vez instalado, de todos los paquetes (libros) que tenemos, le indicaremos que nos cargue ese concretamente.
Para convertir a tipo fecha usaremos la función as_date() del paquete {lubridate}
Variables de fecha
En dicho paquete tenemos funciones muy útiles para manejar fechas:
- Con
today()podemos obtener directamente la fecha actual.
Resúmenes de paquetes

Amplia contenido
Tienes un resumen en pdf de los paquetes más importantes en la carpeta correspondiente en el campus
💻 Tu turno
Intenta realizar los siguientes ejercicios sin mirar las soluciones
📝 Define una variable que guarde tu edad (llamada edad) y otra con tu nombre (llamada nombre)
📝 Comprueba si NO tienes 60 años o si te llamas “Ornitorrinco” (debes obtener variables lógicas)
📝 Define otra variable llamada hermanos que responda la pregunta «¿tienes hermanos?» y otra variable que almacene tu fecha de nacimiento (llamada fecha_nacimiento).
📝 Define otra variable con tus apellidos (llamada apellidos) y usa glue() para tener, en una sola variable llamada nombre_completo, tu nombre y apellidos separando nombre y apellido por una coma
Vectores: concatenar
Cuando trabajamos con datos normalmente tendremos columnas que representan variables: llamaremos vectores a una concatenación de celdas (valores) del mismo tipo (lo que sería una columna de una tabla).
La forma más sencilla es con el comando c() (c de concatenar), y basta con introducir sus elementos entre paréntesis y separados por comas
Tip
Un número individual x <- 1 (o bien x <- c(1)) es en realidad un vector de longitud uno –> todo lo que sepamos hacer con un número podemos hacerlo con un vector de ellos.
Vectores: concatenar
Secuencias numéricas
El vector más famoso será el de tipo numérico, y en concreto, las conocidas como secuencias numéricas (por ejemplo, los días del mes), usadas para, entre otras cosas, indexar bucles.
El comando seq(inicio, fin) nos permite crear una secuencia numérica desde un elemento inicial hasta uno final, avanzando de uno en uno.
Secuencias numéricas
Un atajo es el comando 1:n, que nos devuelve lo mismo que seq(1, n)
Si el elemento inicial es mayor que el final, entenderá que la secuencia es en orden decreciente.
Secuencias numéricas
Otras veces nos interesará definir una secuencia con una longitud concreta
[1] 1.000000 9.166667 17.333333 25.500000 33.666667 41.833333 50.000000Incluso puede nos interese generar un vector de n elementos repetidos
Vectores de caracteres
Un vector es una concatenación de elementos del mismo tipo, pero no tienen porque ser necesariamente números. Vamos a crear una frase de ejemplo.
Vectores de caracteres
¿Qué sucederá si concatenamos elementos de diferente tipo?
Fíjate que como todos tienen que ser del mismo tipo, lo que hace R es convertir todo a texto, violando la integridad del dato
Operaciones con vectores
Con los vectores numéricos podemos hacer las mismas operaciones aritméticas que con los números → un número es un vector (de longitud uno)
¿Qué sucederá si sumamos o restamos un valor a un vector?
Operaciones con vectores
Los vectores también pueden interactuar entre ellos, así que podemos definir, por ejemplo, sumas de vectores (elemento a elemento)
Dado que la operación (por ejemplo, una suma) se realiza elemento a elemento, ¿qué sucederá si sumamos dos vectores de distinta longitud?
Operaciones con vectores
Una operación muy habitual es preguntar a los datos mediante el uso de condiciones lógicas. Por ejemplo, si definimos un vector de temperaturas…
¿Qué días hizo menos de 22 grados?
Nos devolverá un vector lógico, en función de si cada elemento cumple o no la condición pedida (de igual longitud que el vector preguntado)
Operaciones con vectores
Las condiciones lógicas pueden ser combinadas de dos maneras:
- Intersección: todas las condiciones concatenadas se deben cumplir (conjunción y con
&) para devolver unTRUE
- Unión: basta con que al menos una se cumpla (conjunción o con
|)
Operaciones con vectores
Otra operación muy habitual es la de acceder a elementos. La forma más sencilla es usar el operador [i] (acceder al elemento i-ésimo)
Dado que un número no es más que un vector de longitud uno, esta operación también la podemos aplicar usando un vector de índices a seleccionar
Tip
Para acceder al último, sin preocuparnos de cuál es, podemos pasarle como índice la propia longitud x[length(x)]
Operaciones con vectores
Otras veces no querremos seleccionar sino eliminar algunos elementos. Deberemos repetir la misma operación pero con el signo - delante: el operador [-i] no selecciona el elemento i-ésimo del vector sino que lo «des-selecciona»
En muchas ocasiones los queremos seleccionar o eliminar en base a condiciones lógicas, en función de los valores, así que pasaremos como índice la propia condición (recuerda, x < 2 nos devuelve un vector lógico)
💻 Tu turno
Intenta realizar los siguientes ejercicios sin mirar las soluciones
📝 Define el vector x como la concatenación de los 5 primeros números impares. Calcula la longitud del vector
📝 Accede al tercer elemento. Accede al último elemento (sin importar la longitud, un código que pueda ejecutarse siempre). Elimina el primer elemento.
📝 Obtén de dicho vector los elementos de x mayores que 4. Calcula el vector 1/x y guárdalo en una variable.
📝 Crea un vector que represente los nombres de 5 personas, de los cuales uno es desconocido.
📝 Encuentra del vector x de ejercicios anteriores los elementos mayores (estrictos) que 1 Y ADEMÁS menores (estrictos) que 7. Encuentra una forma de averiguar si todos los elementos son o no positivos.
📝 Dado el vector x <- c(1, -5, 8, NA, 10, -3, 9), extrae los elementos que ocupan los lugares 1, 2, 5, 6. Elimina del vector el segundo elemento. Tras eliminarlo determina su suma y su media
🐣 Caso práctico
En el paquete {datasets} tenemos diversos conjuntos de datos y uno de ellos es airquality. Debajo te he extraído 3 variables de dicho dataset
- ¿Qué representan los datos? ¿Cómo averiguarlo?
Haciendo uso de ? ... podemos consultar en el panel de ayuda lo que significa el objeto.
🐣 Caso práctico
- ¿Cuántos registros tenemos de mayo? ¿Y de abril? Construye una nueva variable
datecon la fecha de cada registro (combinando año, mes y día)
[1] 31[1] 31[1] 0🐣 Caso práctico
- ¿Cuál fue la media de temperatura del mes de agosto? Extrae los días en los que la temperatura superó los 30 grados y calcula la cantidad de días en los que lo hizo.
[1] 83.96774[1] 28.87097 [1] "1973-06-08" "1973-06-09" "1973-06-10" "1973-06-11" "1973-06-12"
[6] "1973-07-07" "1973-07-08" "1973-07-09" "1973-07-10" "1973-07-14"
[11] "1973-07-19" "1973-07-28" "1973-08-06" "1973-08-07" "1973-08-08"
[16] "1973-08-09" "1973-08-10" "1973-08-27" "1973-08-28" "1973-08-29"
[21] "1973-08-30" "1973-08-31" "1973-09-01" "1973-09-02" "1973-09-03"
[26] "1973-09-04" "1973-09-05"[1] 27[1] 27Operaciones con vectores
También podemos hacer uso de operaciones estadísticas como por ejemplo sum() que, dado un vector, nos devuelve la suma de todos sus elementos.
¿Qué sucede cuando falta un dato (ausente)?
Operaciones con vectores
Como hemos comentado que los valores lógicos son guardados internamente como 0 y 1, podremos usarlos en operaciones aritméticas.
Por ejemplo, si queremos averiguar el número de elementos que cumplen una condición (por ejemplo, menores que 3), los que lo hagan tendrán asignado un 1 (TRUE) y los que no un 0 (FALSE) , por lo que basta con sumar dicho vector lógico para obtener el número de elementos que cumplen
Operaciones con vectores
Otra operación habitual que puede sernos útil es la suma acumulada con cumsum() que, dado un vector, nos devuelve un vector a su vez con el primero, el primero más el segundo, el primero más el segundo más el tercero…y así sucesivamente.
¿Qué sucede cuando falta un dato (ausente)?
Operaciones con vectores
Otra operación habitual que puede sernos útil es la diferencia (con retardo) con diff() que, dado un vector, nos devuelve un vector con el segundo menos el primero, el tercero menos el segundo, el cuarto menos el tercero…y así sucesivamente.
Operaciones con vectores
Otras operaciones habituales son la media, mediana, percentiles, etc.
- Media: medida de centralidad que consiste en sumar todos los elementos y dividirlos entre la cantidad de elementos sumados. La más conocida pero la menos robusta: dado un conjunto, si se introducen valores atípicos o outliers (valores muy grandes o muy pequeños), la media se perturba con mucha facilidad.
Operaciones con vectores
Otras operaciones habituales son la media, mediana, percentiles, etc.
- Mediana: medida de centralidad que consiste en ordenar los elementos y quedarse con el que ocupa la mitad.
Clase 1: primeras bases de datos
Variables de tipo texto. Primeras bases de datos
Última operación: ordenar
Por último, una acción habitual es saber ordenar valores:
sort(): devuelve el vector ordenado. Por defecto de menor a mayor pero condecreasing = TRUEpodemos cambiarlo
[1] 7 20 23 25 33 41 65 77 81[1] 81 77 65 41 33 25 23 20 7💻 Tu turno
Intenta realizar los siguientes ejercicios sin mirar las soluciones
📝 Define el vector x como la concatenación de los 4 primeros números pares, y calcula su suma.
📝 Obtén los elementos de x menores estrictamente que 5. Calcula el número de elementos de x menores estrictamente que 5.
📝 Calcula el vector 1/x y obtén la versión ordenada (de menor a mayor) de las dos formas posibles
📝 Encuentra del vector x los elementos mayores (estrictos) que 1 y menores (estrictos) que 6. Encuentra una forma de averiguar si todos los elementos son o no negativos.
Clase 2: bases de datos en R
Primeras bases de datos. Tidydata
Clase 2: matrices
Matrices
Primera base de datos: matrices
Cuando analizamos datos solemos tener varias variables de cada individuo: necesitamos una «tabla» que las recopile. La opción más inmediata son las matrices: concatenación de variables del mismo tipo e igual longitud.
Imagina que tenemos estaturas y pesos de 4 personas. ¿Cómo crear un dataset con las dos variables?
- Con
cbind()concatenamos vectores en forma de columnas
Primer intento: matrices
- También podemos construir la matriz por filas con la función
rbind()(aunque lo recomendable es tener cada variable en columna e individuo en fila).
[,1] [,2] [,3] [,4]
estaturas 150 160 170 180
pesos 63 70 85 95- Podemos visualizar la matriz con la función
View().
Primer intento: matrices
- También podemos «darle vuelta» (matriz transpuesta) con
t().
Primer intento: matrices
- También podemos definir una matriz a partir de un vector numérico, reorganizando los valores en forma de matriz (sabiendo que los elementos se van colocando por columnas).
Primer intento: matrices
Con las matrices sucede como con los vectores: cuando aplicamos una operación aritmética lo hacemos elemento a elemento
[,1] [,2] [,3] [,4] [,5]
[1,] 0.2 0.8 1.4 2.0 2.6
[2,] 0.4 1.0 1.6 2.2 2.8
[3,] 0.6 1.2 1.8 2.4 3.0Primer intento: matrices
También podemos realizar operaciones por columnas/filas sin recurrir a bucles con la función apply(), y le indicaremos como argumentos
- la matriz
- el sentido de la operación (MARGIN = 1 por filas, MARGIN = 2 por columnas)
- la función a aplicar
💻 Tu turno (matrices)
Intenta realizar los siguientes ejercicios sin mirar las soluciones
📝 Modifica el código para definir una matriz x de ceros de 3 filas y 7 columnas de unos.
📝 A la matriz anterior, suma un 1 a cada número de la matriz y divide el resultado entre 5. Tras ello calcula su transpuesta y obtén sus dimensiones
📝 Define la matriz x <- matrix(1:12, nrow = 4). Obtén la primera fila, la tercera columna, y el elemento (4, 1).
📝 Con la matriz anterior definida como x <- matrix(1:12, nrow = 4), calcula la media de todos los elementos, la media de cada fila y la media de cada columna. Calcula la suma de de cada fila y de cada columna
Clase 2: datos tabulados
Data.frames y tibbles
Segundo intento: data.frame
Las matrices tienen el mismo problema que los vectores: si juntamos datos de distinto tipo, se perturba la integridad del dato ya que los convierte
Segundo intento: data.frame
Para poder trabajar con variables de distinto tipo tenemos lo que se conoce como data.frame: concatenación de variables de igual longitud pero pueden ser de tipo distinto.
Segundo intento: data.frame
Dado que un data.frame es ya una «base de datos» las variables no son meros vectores matemáticos: tienen un significado y podemos (debemos) ponerles nombres
Segundo intento: data.frame
¡TENEMOS NUESTRO PRIMER CONJUNTO DE DATOS! Puedes visualizarlo escribiendo su nombre en consola o con View(tabla)
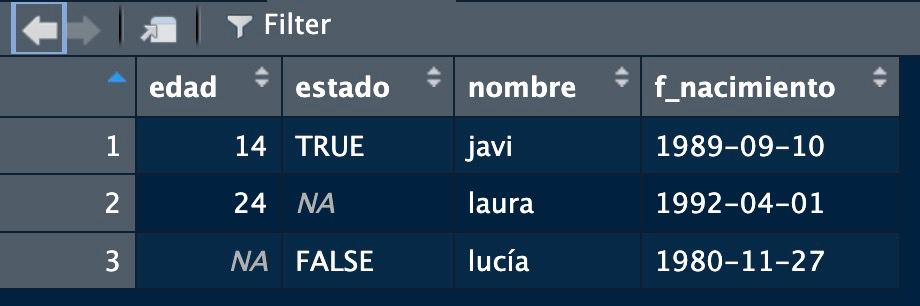
Segundo intento: data.frame
Si queremos acceder a sus elementos, podemos como en las matrices (aunque no es recomendable): ahora tenemos dos índices (filas y columnas, dejando libre la que no usemos)
edad estado nombre f_nacimiento
2 24 NA laura 1992-04-01[1] "javi" "laura" "lucía"[1] 24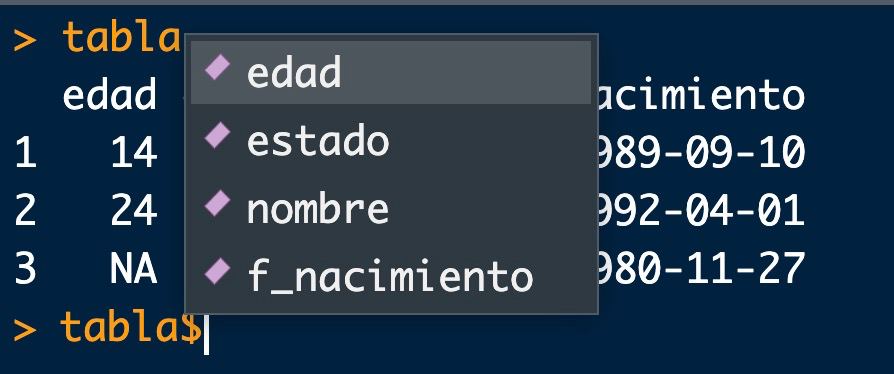
También tiene ventajas de una «base» de datos : podemos aceder a las variables por su nombre (recomendable ya que las variables pueden cambiar de posición), poniendo el nombre de la tabla seguido del símbolo $ (con el tabulador, nos aparecerá un menú de columnas a elegir)
Segundo intento: data.frame
names(): nos muestra los nombres de las variables
Segundo intento: data.frame
Si tenemos uno ya creado y queremos añadir una columna es tan simple como usar la función data.frame() que ya hemos visto para concatenar la columna. Vamos añadir por ejemplo una nueva variable, el número de hermanos de cada individuo.
Intento final: tibble
Las tablas en formato data.frame tienen algunas limitaciones
La principal es que no permite la recursividad: imagina que definimos una base de datos con estaturas y pesos, y queremos una tercera variable con el IMC
Error in data.frame(estatura = c(1.7, 1.8, 1.6), peso = c(80, 75, 70), : object 'peso' not foundIntento final: tibble
tabla <- tibble("estatura" = c(1.7, 1.8, 1.6), "peso" = c(80, 75, 70),
"IMC" = peso / (estatura^2))
tabla# A tibble: 3 × 3
estatura peso IMC
<dbl> <dbl> <dbl>
1 1.7 80 27.7
2 1.8 75 23.1
3 1.6 70 27.3Las tablas en formato tibble nos permitirá una gestión más ágil, eficiente y coherente de los datos, con 4 ventajas principales:
- Metainformación: si te fijas en la cabecera, nos dice ya automáticamente el número de filas y columnas, y el tipo de cada variable
- Recursividad: permite definir las variables secuencialmente (como hemos visto)
Intento final: tibble
- Consistencia: si accedes a una columna que no existe avisa con un warning
- Por filas: crear por filas (copiar y pegar de una tabla) con
tribble()
Tip
El paquete {datapasta} nos permite copiar y pegar tablas de páginas web y documentos sencillos
💻 Tu turno (tibble)
Intenta realizar los siguientes ejercicios sin mirar las soluciones
📝 Carga del paquete {datasets} el conjunto de datos airquality (variables de la calidad del aire de Nueva York desde mayo hasta septiembre de 1973). ¿Es el conjunto de datos airquality de tipo tibble? En caso negativo, conviértelo a tibble (busca en la documentación del paquete en https://tibble.tidyverse.org/index.html).
📝 Una vez convertido a tibble obtén el nombre de las variables y las dimensiones del conjunto de datos. ¿Cuántas variables hay? ¿Cuántos días se han medido?
📝 Selecciona aquellos datos que no sean ni de julio ni de agosto.
📝 Modifica el siguiente código para quedarte solo con las variable de ozono y temperatura.
Resumen hasta ahora
- Cada celda puede ser de un tipo diverso: números, texto, fechas, valores lógicos, etc
- Un vector es una concatenación de celdas (las futuras columnas de nuestras tablas) –> En
Rpor defecto las operaciones se hacen elemento a elemento
- Una matriz nos permite concatenar variables del MISMO tipo y MISMA longitud –> objeto bidimensional (dos índices)
- Un data.frame nos permite concatenar variables de DISTINTO tipo y MISMA longitud –> usaremos tibble como una opción mejorada de base de datos
🐣 Caso práctico
Del paquete Biostatistics usaremos el conunto de datos pinniped
- ¿Qué representan los datos? ¿Qué tipo de dato es? En caso de que no lo sea, convierte la base de datos a un
tibble(renombra conpinniped_tb)
[1] "data.frame"🐣 Caso práctico
- Incorpora una variable extra llamada phoca que sea de tipo lógico y que nos diga si una especie es de la categoría
Phocao no.
- ¿A qué sexo le pesa más el cerebro: a las hembras o a los machos? ¿A quienes les pesa más el cuerpo: a los monógamos o a los polígamos?
Código
[1] TRUECódigo
[1] FALSE🐣 Caso práctico
- Incopora una nueva variable que represente la diferencia entre el peso del cerebro entre machos y hembras (machos - hembras) para cada especie.
# A tibble: 33 × 8
Species Male_brain_g Female_brain_g Male_mass_Kg Female_mass_Kg Mate_type
<chr> <dbl> <dbl> <dbl> <dbl> <chr>
1 Monachus s… 370 NA 173 272. mono
2 Monachus m… 480 480 260 275 mono
3 Mirounga a… 700 640 2275 488 poly
4 Mirounga l… 1431. 899. 3510 566. poly
5 Leptonycho… 535 638. 450 447 poly
6 Ommatophoc… 425 530 154. 164 mono
7 Lobodon ca… 578. 539. 220. 224 mono
8 Hydrurga l… 765 660 324 367 mono
9 Cystophora… 480 430 343. 222. mono
10 Erignathus… NA 460 312. 326 mono
# ℹ 23 more rows
# ℹ 2 more variables: phoca <lgl>, dif_m_f <dbl>Clase 2: tidydata
Nuestra base de datos: tibble. Tidydata: un multiverso de datos limpios
Previously, in Breaking Bad…
Nuestro formato final de base de datos será el objeto de tipo tibble, un data.frame mejorado
library(tibble)
tibble("estatura" = c(1.7, 1.8, 1.6), "peso" = c(80, 75, 70), "IMC" = peso / (estatura^2))# A tibble: 3 × 3
estatura peso IMC
<dbl> <dbl> <dbl>
1 1.7 80 27.7
2 1.8 75 23.1
3 1.6 70 27.3- Metainformación: en la cabecera nos dice ya automáticamente el número de filas y columnas, y el tipo de cada variable
- Recursividad: permite definir las variables secuencialmente (como hemos visto)
- Consistencia: si accedes a una columna que no existe avisa con un warning
- Por filas: permite crear por filas con
tribble()
Previously, in Breaking Bad…
Para definir un tibble() nosotro mismos tenemos dos opciones:
- Concatenando vectores que ya tengamos definidos, haciendo uso de la función
tibble()del paquete{tibble}(ya incluido en{tidyverse})
Previously, in Breaking Bad…
- Directamente en tibble proporcionando manualmente valores y nombres de variables
R base vs Tidyverse
Si conoces algún otro lenguaje de programación (o tienes gente cercana que programa) te extrañará que aún no hayamos hablado de conceptos habituales como
Bucles for: repetir un código un número fijo de iteraciones.
Bucles while: repetir un código hasta que se cumpla una condición
Estructuras if-else: estructuras de control para decidir por donde camina el código en función del valor de las variables.
Y aunque conocer dichas estructuras puede sernos en algún momento interesante, en la mayoría de ocasiones vamos a poder evitarlas (en especial los bucles)
¿Qué es tidyverse?

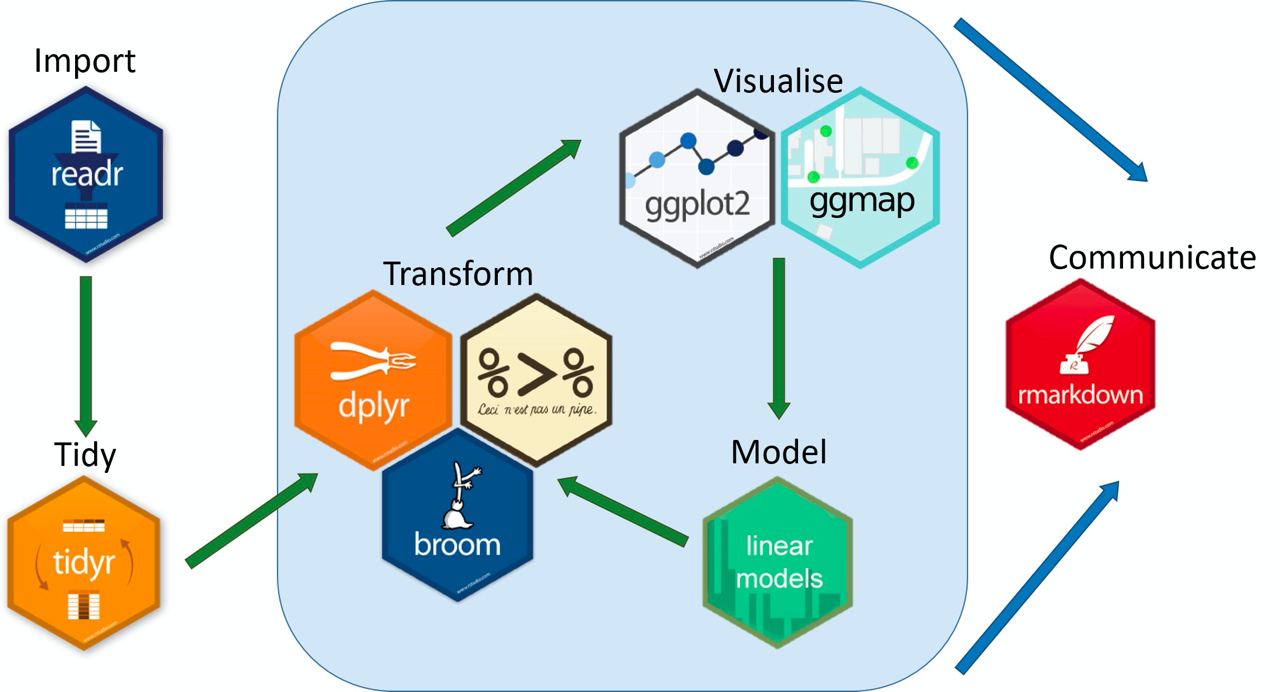
{tidyverse} es un «universo» de paquetes para garanatizar un flujo de trabajo (de inicio a fin) eficiente, coherente y lexicográficamente sencillo de entender, basado en la idea de que nuestros datos están limpios y ordenados (tidy)
¿Qué es tidyverse?

{tibble}: optimizando data.frame{tidyr}: limpieza de datos{readr}: carga datos rectangulares (.csv){dplyr}: gramática para depurar{stringr}: manejo de textos{ggplot2}: visualización de datos{tidymodels}: modelización/predicción
También tenemos los paquetes {purrr} para el manejo de listas, {forcast} para cualitativas, {lubridate} para fechas, {readxl} para importar archivos .xls y .xlsx, {rvest} para web scraping y {rmarkdown} para comunicar resultados.
¿Qué es tidyverse?

{tibble}: optimizando data.frame{tidyr}: limpieza de datos{readr}: carga datos rectangulares (.csv){dplyr}: gramática para depurar{stringr}: manejo de textos{ggplot2}: visualización de datos{tidymodels}: modelización/predicción
También tenemos los paquetes {purrr} para el manejo de listas, {forcast} para cualitativas, {lubridate} para fechas, {readxl} para importar archivos .xls y .xlsx, {rvest} para web scraping y {rmarkdown} para comunicar resultados.
Filosofía base: tidy data
Tidy datasets are all alike, but every messy dataset is messy in its own way (Hadley Wickham, Chief Scientist en RStudio)
TIDYVERSE
El universo de paquetes {tidyverse} se basa en la idea introducido por Hadley Wickham (el Dios al que rezo) de estandarizar el formato los datos para
- sistematizar la depuración
- hacer más sencillo su manipulación.
- código legible
Reglas del tidy data
Lo primero por tanto será entender qué son los conjuntos tidydata ya que todo {tidyverse} se basa en que los datos están estandarizados.
- Cada variable en una única columna
- Cada individuo en una fila diferente
- Cada celda con un único valor
- Cada dataset en un tibble
- Si queremos cruzar múltiples tablas debemos tener una columna común

Tubería (pipe)
En {tidyverse} será clave el operador pipe (tubería) definido como |> (ctrl+shift+M): será una tubería que recorre los datos y los transforma.
En R base, si queremos aplicar tres funciones first(), second() y third() en orden, sería
Apunte importante
Desde la versión 4.1.0 de R disponemos de |>, un pipe nativo disponible fuera de tidyverse, sustituyendo al antiguo pipe %>% que dependía del paquete {magrittr} (bastante problemático).
Tubería (pipe)
La principal ventaja es que el código sea muy legible (casi literal) pudiendo hacer grandes operaciones con los datos con apenas código.
Datos SUCIOS: messy data
¿Pero qué aspecto tienen los datos no tidy? Vamos a cargar la tabla table4a del paquete {tidyr} (ya lo tenemos cargado del entorno tidyverse).
# A tibble: 3 × 3
country `1999` `2000`
<chr> <dbl> <dbl>
1 Afghanistan 745 2666
2 Brazil 37737 80488
3 China 212258 213766
¿Qué puede estar fallando?
Pivotar: pivot_longer()
❎ Cada fila representa dos observaciones (1999 y 2000) → las columnas 1999 y 2000 en realidad deberían ser en sí valores de una variable y no nombres de columnas.
Incluiremos una nueva columna que nos guarde el año y otra que guarde el valor de la variable de interés en cada uno de esos años. Y lo haremos con la función pivot_longer(): pivotaremos la tabla a formato long:
Pivotar: pivot_longer()
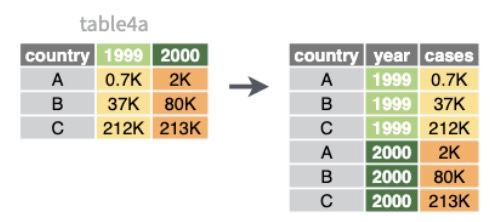
cols: nombre de las variables a pivotarnames_to: nombre de la nueva variable a la quemandamos la cabecera de la tabla (los nombres).values_to: nombre de la nueva variable a la que vamos a mandar los datos.
Datos SUCIOS: messy data
Veamos otro ejemplo con la tabla table2
# A tibble: 12 × 4
country year type count
<chr> <dbl> <chr> <dbl>
1 Afghanistan 1999 cases 745
2 Afghanistan 1999 population 19987071
3 Afghanistan 2000 cases 2666
4 Afghanistan 2000 population 20595360
5 Brazil 1999 cases 37737
6 Brazil 1999 population 172006362
7 Brazil 2000 cases 80488
8 Brazil 2000 population 174504898
9 China 1999 cases 212258
10 China 1999 population 1272915272
11 China 2000 cases 213766
12 China 2000 population 1280428583
¿Qué puede estar fallando?
Pivotar: pivot_wider()
# A tibble: 12 × 4
country year type count
<chr> <dbl> <chr> <dbl>
1 Afghanistan 1999 cases 745
2 Afghanistan 1999 population 19987071
3 Afghanistan 2000 cases 2666
4 Afghanistan 2000 population 20595360
5 Brazil 1999 cases 37737
6 Brazil 1999 population 172006362
7 Brazil 2000 cases 80488
8 Brazil 2000 population 174504898
9 China 1999 cases 212258
10 China 1999 population 1272915272
11 China 2000 cases 213766
12 China 2000 population 1280428583❎ Cada observación está dividido en dos filas → los registros con el mismo año deberían ser el mismo
Lo que haremos será lo opuesto: con pivot_wider() ensancharemos la tabla
# A tibble: 6 × 4
country year cases population
<chr> <dbl> <dbl> <dbl>
1 Afghanistan 1999 745 19987071
2 Afghanistan 2000 2666 20595360
3 Brazil 1999 37737 172006362
4 Brazil 2000 80488 174504898
5 China 1999 212258 1272915272
6 China 2000 213766 1280428583Datos SUCIOS: messy data
Veamos otro ejemplo con la tabla table3
# A tibble: 6 × 3
country year rate
<chr> <dbl> <chr>
1 Afghanistan 1999 745/19987071
2 Afghanistan 2000 2666/20595360
3 Brazil 1999 37737/172006362
4 Brazil 2000 80488/174504898
5 China 1999 212258/1272915272
6 China 2000 213766/1280428583¿Qué puede estar fallando?
Separar: separate()
❎ Cada celda contiene varios valores
Lo que haremos será hacer uso de la función separate() para mandar separar cada valor a una columna diferente.
# A tibble: 6 × 4
country year cases pop
<chr> <dbl> <chr> <chr>
1 Afghanistan 1999 745 19987071
2 Afghanistan 2000 2666 20595360
3 Brazil 1999 37737 172006362
4 Brazil 2000 80488 174504898
5 China 1999 212258 1272915272
6 China 2000 213766 1280428583Separar: separate()
# A tibble: 6 × 4
country year cases pop
<chr> <dbl> <chr> <chr>
1 Afghanistan 1999 745 19987071
2 Afghanistan 2000 2666 20595360
3 Brazil 1999 37737 172006362
4 Brazil 2000 80488 174504898
5 China 1999 212258 1272915272
6 China 2000 213766 1280428583Fíjate que los datos, aunque los ha separado, los ha mantenido como texto cuando en realidad deberían ser variables numéricas. Para ello podemos añadir el argumento opcional convert = TRUE
# A tibble: 6 × 4
country year cases pop
<chr> <dbl> <int> <int>
1 Afghanistan 1999 745 19987071
2 Afghanistan 2000 2666 20595360
3 Brazil 1999 37737 172006362
4 Brazil 2000 80488 174504898
5 China 1999 212258 1272915272
6 China 2000 213766 1280428583Datos SUCIOS: messy data
Veamos el último ejemplo con la tabla table5
# A tibble: 6 × 4
country century year rate
<chr> <chr> <chr> <chr>
1 Afghanistan 19 99 745/19987071
2 Afghanistan 20 00 2666/20595360
3 Brazil 19 99 37737/172006362
4 Brazil 20 00 80488/174504898
5 China 19 99 212258/1272915272
6 China 20 00 213766/1280428583¿Qué puede estar fallando?
Unir unite()
❎ Tenemos mismos valores divididos en dos columnas
Usaremos unite() para unir los valores de siglo y año en una misma columna
# A tibble: 6 × 3
country year_completo rate
<chr> <chr> <chr>
1 Afghanistan 1999 745/19987071
2 Afghanistan 2000 2666/20595360
3 Brazil 1999 37737/172006362
4 Brazil 2000 80488/174504898
5 China 1999 212258/1272915272
6 China 2000 213766/1280428583💻 Tu turno
Intenta realizar los siguientes ejercicios sin mirar las soluciones
📝 Echa un vistazo a la tabla table4b del paquete {tidyr}. ¿Es tidydata? En caso negativo, ¿qué falla? ¿Cómo convertirla a tidy data en caso de que no lo sea ya?
📝 Echa un vistazo a la tabla relig_income del paquete {tidyr}. ¿Es tidydata? En caso negativo, ¿qué falla? ¿Cómo convertirla a tidy data en caso de que no lo sea ya?
📝 Echa un vistazo a la tabla billboard del paquete {tidyr}. ¿Es tidydata? En caso negativo, ¿qué falla? ¿Cómo convertirla a tidy data en caso de que no lo sea ya?
🐣 Caso práctico
En el paquete {tidyr} contamos con el dataset who (dataset de la Organización Mundial de la Salud)
- ¿Qué significan los datos? ¿Cuántas variables y observaciones tenemos?
- ¿Cuántos tipos de variables tenemos?
- ¿Todas las variables son necesarias? Elimina la información redundante.
- Convierte a tidydata la base de datos realizando todas las opciones que consideres (consejo: usa papel y boli para bocetar como debería quedar la base de datos).
Clase 3: manipulación en R
Manipulación en R
Clase 3: tidyverse (filas)
Operaciones con filas
¿Qué es tidyverse?

{tibble}: optimizando data.frame{tidyr}: limpieza de datos{readr}: carga datos rectangulares (.csv){dplyr}: gramática para depurar{stringr}: manejo de textos{ggplot2}: visualización de datos{tidymodels}: modelización/predicción
También tenemos los paquetes {purrr} para el manejo de listas, {forcast} para cualitativas, {lubridate} para fechas, {readxl} para importar archivos .xls y .xlsx, {rvest} para web scraping y {rmarkdown} para comunicar resultados.
Preprocesamiento: dplyr
Dentro de {tidyverse} usaremos el paquete {dplyr} para el preprocesamiento y depuración de datos de datos.
La idea es que el código sea legible, como si fuese una lista de instrucciones que al leerla nos diga de manera muy evidente lo que está haciendo.
Hipótesis: tidydata
Toda la depuración que vamos a realizar es sobre la hipótesis de que nuestros datos están en tidydata

Recuerda que en {tidyverse} será clave el operador pipe (tubería) definido como |> (ctrl+shift+M): será una tubería que recorre los datos y los transforma.
Muestreo
Una de las operaciones más comunes es lo que se conoce en estadística como muestreo: una selección o filtrado de registros (una submuestra)
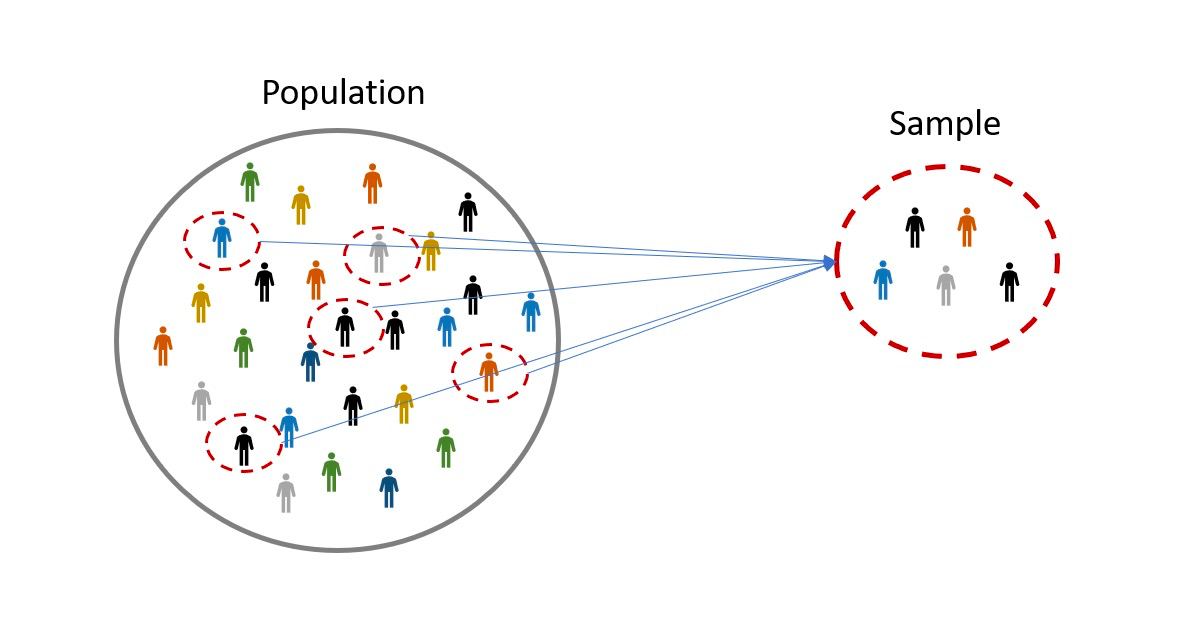
- No aleatorio (por cuotas): en base a condiciones lógicas sobre los registros (
filter())
- No aleatorio (intencional/discreccional): en base a posición (
slice())
- Aleatorio simple (
slice_sample())
- Aleatorio estratificado (
group_by()+slice_sample())
Filtrar filas: filter()
El más simple es cuando filtramos registros en base a alguna condición lógica: con filter() se seleccionarán solo individuos que cumplan ciertas condiciones (muestreo no aleatorio por condiciones)
==,!=: igual o distinto que (|> filter(variable == "a"))>,<: mayor o menor que (|> filter(variable < 3))>=,<=: mayor o igual o menor o igual que (|> filter(variable >= 5))%in%: valores pertenencen a un listado de opciones (|> filter(variable %in% c("azul", "verde")))between(variable, val1, val2): si los valores (continuos) caen dentro de un rango de valores (|> filter(between(variable, 160, 180)))
Filtrar filas: filter()
Dichas condiciones lógicas las podemos combinar de diferentes maneras (y, o, o excluyente)
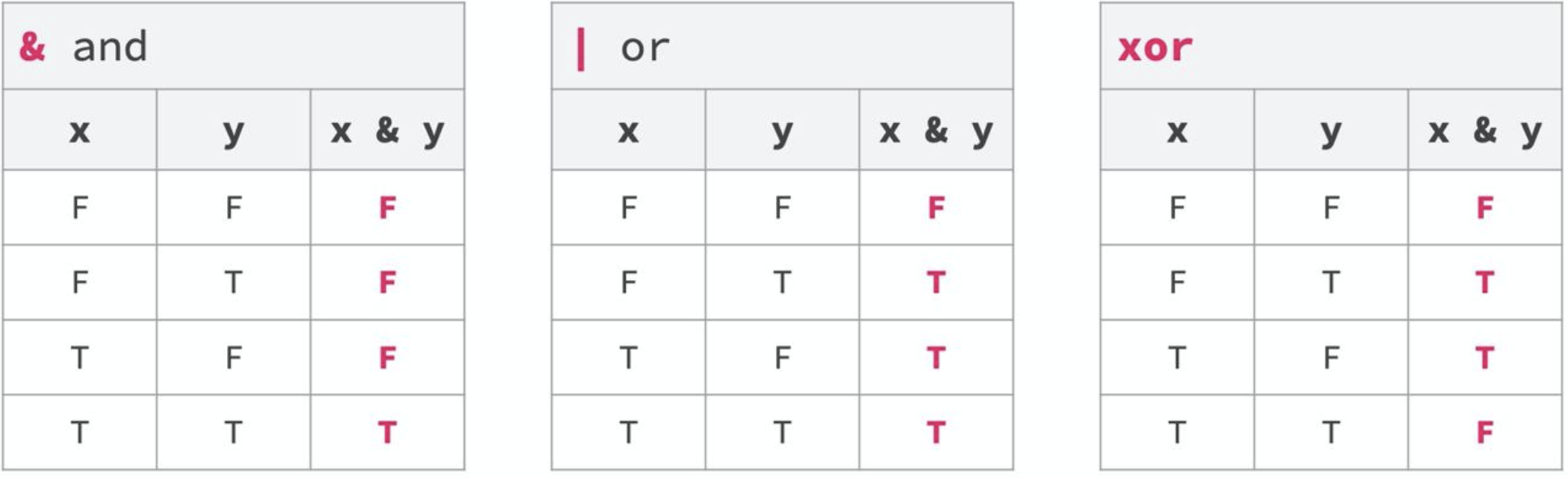
Importante
Recuerda que dentro de filter() debe ir siempre algo que devuelva un vector de valores lógicos.
Filtrar filas: filter()
¿Cómo harías para… filtrar los personajes de ojos marrones?
¿Qué tipo de variable es? –> La variable eye_color es cualitativa así que está representada por textos
# A tibble: 21 × 14
name height mass hair_color skin_color eye_color birth_year sex gender
<chr> <int> <dbl> <chr> <chr> <chr> <dbl> <chr> <chr>
1 Leia Or… 150 49 brown light brown 19 fema… femin…
2 Biggs D… 183 84 black light brown 24 male mascu…
3 Han Solo 180 80 brown fair brown 29 male mascu…
4 Yoda 66 17 white green brown 896 male mascu…
5 Boba Fe… 183 78.2 black fair brown 31.5 male mascu…
6 Lando C… 177 79 black dark brown 31 male mascu…
7 Arvel C… NA NA brown fair brown NA male mascu…
8 Wicket … 88 20 brown brown brown 8 male mascu…
9 Padmé A… 185 45 brown light brown 46 fema… femin…
10 Quarsh … 183 NA black dark brown 62 male mascu…
# ℹ 11 more rows
# ℹ 5 more variables: homeworld <chr>, species <chr>, films <list>,
# vehicles <list>, starships <list>Filtrar filas: filter()
¿Cómo harías para… filtrar los personajes que no tienen ojos marrones?
# A tibble: 66 × 14
name height mass hair_color skin_color eye_color birth_year sex gender
<chr> <int> <dbl> <chr> <chr> <chr> <dbl> <chr> <chr>
1 Luke Sk… 172 77 blond fair blue 19 male mascu…
2 C-3PO 167 75 <NA> gold yellow 112 none mascu…
3 R2-D2 96 32 <NA> white, bl… red 33 none mascu…
4 Darth V… 202 136 none white yellow 41.9 male mascu…
5 Owen La… 178 120 brown, gr… light blue 52 male mascu…
6 Beru Wh… 165 75 brown light blue 47 fema… femin…
7 R5-D4 97 32 <NA> white, red red NA none mascu…
8 Obi-Wan… 182 77 auburn, w… fair blue-gray 57 male mascu…
9 Anakin … 188 84 blond fair blue 41.9 male mascu…
10 Wilhuff… 180 NA auburn, g… fair blue 64 male mascu…
# ℹ 56 more rows
# ℹ 5 more variables: homeworld <chr>, species <chr>, films <list>,
# vehicles <list>, starships <list>Filtrar filas: filter()
¿Cómo harías para … filtrar los personajes que tengan los ojos marrones o azules?
# A tibble: 40 × 14
name height mass hair_color skin_color eye_color birth_year sex gender
<chr> <int> <dbl> <chr> <chr> <chr> <dbl> <chr> <chr>
1 Luke Sk… 172 77 blond fair blue 19 male mascu…
2 Leia Or… 150 49 brown light brown 19 fema… femin…
3 Owen La… 178 120 brown, gr… light blue 52 male mascu…
4 Beru Wh… 165 75 brown light blue 47 fema… femin…
5 Biggs D… 183 84 black light brown 24 male mascu…
6 Anakin … 188 84 blond fair blue 41.9 male mascu…
7 Wilhuff… 180 NA auburn, g… fair blue 64 male mascu…
8 Chewbac… 228 112 brown unknown blue 200 male mascu…
9 Han Solo 180 80 brown fair brown 29 male mascu…
10 Jek Ton… 180 110 brown fair blue NA <NA> <NA>
# ℹ 30 more rows
# ℹ 5 more variables: homeworld <chr>, species <chr>, films <list>,
# vehicles <list>, starships <list>Filtrar filas: filter()
Fíjate que %in% es equivalente a concatenar varios == con una conjunción o (|)
# A tibble: 40 × 14
name height mass hair_color skin_color eye_color birth_year sex gender
<chr> <int> <dbl> <chr> <chr> <chr> <dbl> <chr> <chr>
1 Luke Sk… 172 77 blond fair blue 19 male mascu…
2 Leia Or… 150 49 brown light brown 19 fema… femin…
3 Owen La… 178 120 brown, gr… light blue 52 male mascu…
4 Beru Wh… 165 75 brown light blue 47 fema… femin…
5 Biggs D… 183 84 black light brown 24 male mascu…
6 Anakin … 188 84 blond fair blue 41.9 male mascu…
7 Wilhuff… 180 NA auburn, g… fair blue 64 male mascu…
8 Chewbac… 228 112 brown unknown blue 200 male mascu…
9 Han Solo 180 80 brown fair brown 29 male mascu…
10 Jek Ton… 180 110 brown fair blue NA <NA> <NA>
# ℹ 30 more rows
# ℹ 5 more variables: homeworld <chr>, species <chr>, films <list>,
# vehicles <list>, starships <list>Filtrar filas: filter()
¿Cómo harías para … filtrar los personajes que midan entre 120 y 160 cm?
¿Qué tipo de variable es? –> La variable height es cuantitativa continua así que deberemos filtrar por rangos de valores (intervalos) –> usaremos between()
# A tibble: 6 × 14
name height mass hair_color skin_color eye_color birth_year sex gender
<chr> <int> <dbl> <chr> <chr> <chr> <dbl> <chr> <chr>
1 Leia Org… 150 49 brown light brown 19 fema… femin…
2 Mon Moth… 150 NA auburn fair blue 48 fema… femin…
3 Nien Nunb 160 68 none grey black NA male mascu…
4 Watto 137 NA black blue, grey yellow NA male mascu…
5 Gasgano 122 NA none white, bl… black NA male mascu…
6 Cordé 157 NA brown light brown NA <NA> <NA>
# ℹ 5 more variables: homeworld <chr>, species <chr>, films <list>,
# vehicles <list>, starships <list>Filtrar filas: filter()
¿Cómo harías… filtrar los personajes que tengan ojos y no sean humanos?
# A tibble: 3 × 14
name height mass hair_color skin_color eye_color birth_year sex gender
<chr> <int> <dbl> <chr> <chr> <chr> <dbl> <chr> <chr>
1 Yoda 66 17 white green brown 896 male mascu…
2 Wicket S… 88 20 brown brown brown 8 male mascu…
3 Eeth Koth 171 NA black brown brown NA male mascu…
# ℹ 5 more variables: homeworld <chr>, species <chr>, films <list>,
# vehicles <list>, starships <list>Filtrar filas: filter()
¿Cómo harías… filtrar los personajes que tengan ojos y no sean humanos, o que tengan más de 60 años? Piénsalo bien: los paréntesis son importantes: no es lo mismo \((a+b)*c\) que \(a+(b*c)\)
# A tibble: 18 × 14
name height mass hair_color skin_color eye_color birth_year sex gender
<chr> <int> <dbl> <chr> <chr> <chr> <dbl> <chr> <chr>
1 C-3PO 167 75 <NA> gold yellow 112 none mascu…
2 Wilhuff… 180 NA auburn, g… fair blue 64 male mascu…
3 Chewbac… 228 112 brown unknown blue 200 male mascu…
4 Jabba D… 175 1358 <NA> green-tan… orange 600 herm… mascu…
5 Yoda 66 17 white green brown 896 male mascu…
6 Palpati… 170 75 grey pale yellow 82 male mascu…
7 Wicket … 88 20 brown brown brown 8 male mascu…
8 Qui-Gon… 193 89 brown fair blue 92 male mascu…
9 Finis V… 170 NA blond fair blue 91 male mascu…
10 Quarsh … 183 NA black dark brown 62 male mascu…
11 Shmi Sk… 163 NA black fair brown 72 fema… femin…
12 Mace Wi… 188 84 none dark brown 72 male mascu…
13 Ki-Adi-… 198 82 white pale yellow 92 male mascu…
14 Eeth Ko… 171 NA black brown brown NA male mascu…
15 Cliegg … 183 NA brown fair blue 82 male mascu…
16 Dooku 193 80 white fair brown 102 male mascu…
17 Bail Pr… 191 NA black tan brown 67 male mascu…
18 Jango F… 183 79 black tan brown 66 male mascu…
# ℹ 5 more variables: homeworld <chr>, species <chr>, films <list>,
# vehicles <list>, starships <list>Eliminar ausentes: drop_na()
Hay un filtro especial para una de las operaciones más habituales en depuración: retirar los ausentes. Para ello podemos usar dentro de un filtro is.na(), que nos devuelve TRUE/FALSE en función de si es ausente, o bien …
Usar drop_na(): si no indicamos variable, elimina registros con ausente en cualquier variable. Más adelante veremos como imputar esos ausentes
# A tibble: 7 × 4
name mass height hair_color
<chr> <dbl> <int> <chr>
1 Luke Skywalker 77 172 blond
2 C-3PO 75 167 <NA>
3 R2-D2 32 96 <NA>
4 Darth Vader 136 202 none
5 Leia Organa 49 150 brown
6 Owen Lars 120 178 brown, grey
7 Beru Whitesun Lars 75 165 brown # A tibble: 7 × 4
name mass height hair_color
<chr> <dbl> <int> <chr>
1 Luke Skywalker 77 172 blond
2 Darth Vader 136 202 none
3 Leia Organa 49 150 brown
4 Owen Lars 120 178 brown, grey
5 Beru Whitesun Lars 75 165 brown
6 Biggs Darklighter 84 183 black
7 Obi-Wan Kenobi 77 182 auburn, white💻 Tu turno
Intenta realizar los siguientes ejercicios sin mirar las soluciones
📝 Selecciona del conjunto de starwars solo los personajes que sean androides o cuyo valor en species sea desconocido
📝 Selecciona del conjunto de starwars solo los personajes cuyo peso esté entre 65 y 90 kg.
📝 Tras limpiar de ausentes en todas las variables, selecciona del conjunto de starwars solo los personajes que sean humanos y que vengan de Tatooine
📝 Selecciona del conjunto original de starwars los personajes no humanos, male en el sexo y que midan entre 120 y 170 cm, o los personajes con ojos marrones o rojos.
📝 Busca información en la ayuda de la función str_detect() del paquete {stringr} (cargado en {tidyverse}). Consejo: prueba antes las funciones que vayas a usar con algún vector de prueba para poder comprobar su funcionamiento. Tras saber lo que hace, filtra solo aquellos personajes con apellido Skywalker
Rebanadas de datos: slice()
A veces nos puede interesar realizar un muestreo no aleatorio discreccional, o lo que es lo mismo, filtrar por posición: con slice(posiciones) podremos seleccionar filas concretas pasando como argumento un vector de índices
# A tibble: 1 × 4
name height mass hair_color
<chr> <int> <dbl> <chr>
1 Luke Skywalker 172 77 blond # A tibble: 4 × 8
name height mass hair_color skin_color eye_color birth_year sex
<chr> <int> <dbl> <chr> <chr> <chr> <dbl> <chr>
1 C-3PO 167 75 <NA> gold yellow 112 none
2 Beru Whitesun L… 165 75 brown light blue 47 fema…
3 Obi-Wan Kenobi 182 77 auburn, w… fair blue-gray 57 male
4 Qui-Gon Jinn 193 89 brown fair blue 92 male Rebanadas de datos: slice()
Disponemos de opciones por defecto:
- con
slice_head(n = ...)yslice_tail(n = ...)podemos obtener la cabecera y cola de la tabla
Rebanadas de datos: slice()
Disponemos de opciones por defecto:
- con
slice_max()yslice_min()obtenemos la filas con menor/mayor valor de una variable (si empate, todas salvo quewith_ties = FALSE) que indicamos enorder_by = ...
# A tibble: 2 × 4
name height mass hair_color
<chr> <int> <dbl> <chr>
1 Ratts Tyerel 79 15 none
2 Yoda 66 17 white Aleatorio: slice_sample()
El conocido como muestreo aleatorio simple se basa en seleccionar individuos aleatoriamente, de forma que cada uno tenga ciertas probabilidades de ser seleccionado. Con slice_sample(n = ...) podemos extraer n registros aleatoriamente (a priori equiprobables).
# A tibble: 2 × 14
name height mass hair_color skin_color eye_color birth_year sex gender
<chr> <int> <dbl> <chr> <chr> <chr> <dbl> <chr> <chr>
1 Dooku 193 80 white fair brown 102 male mascu…
2 Jek Tono… 180 110 brown fair blue NA <NA> <NA>
# ℹ 5 more variables: homeworld <chr>, species <chr>, films <list>,
# vehicles <list>, starships <list>Importante…
«Aleatorio» no implica equiprobable: es igual de aleatorio un dado normal que uno trucado. No hay cosas «más aleatorias» que otras, simplemente tienen subyacente distintas leyes de probabilidad.
Aleatorio: slice_sample()
También podremos indicarle la proporción de datos a samplear (en lugar del número) y si queremos que sea con reemplazamiento (que se puedan repetir).
# 5% de registros aleatorios con reemplazamiento
starwars |>
slice_sample(prop = 0.05, replace = TRUE)# A tibble: 4 × 14
name height mass hair_color skin_color eye_color birth_year sex gender
<chr> <int> <dbl> <chr> <chr> <chr> <dbl> <chr> <chr>
1 Kit Fisto 196 87 none green black NA male mascu…
2 Ben Quad… 163 65 none grey, gre… orange NA male mascu…
3 Jar Jar … 196 66 none orange orange 52 male mascu…
4 Plo Koon 188 80 none orange black 22 male mascu…
# ℹ 5 more variables: homeworld <chr>, species <chr>, films <list>,
# vehicles <list>, starships <list>Aleatorio: slice_sample()
Como decíamos, «aleatorio» no es igual que «equiprobable», así que podemos pasarle un vector de probabilidades. Por ejemplo, vamos a forzar que sea muy improbable sacar una fila que no sean las dos primeras
# A tibble: 2 × 14
name height mass hair_color skin_color eye_color birth_year sex gender
<chr> <int> <dbl> <chr> <chr> <chr> <dbl> <chr> <chr>
1 C-3PO 167 75 <NA> gold yellow 112 none mascu…
2 Luke Sky… 172 77 blond fair blue 19 male mascu…
# ℹ 5 more variables: homeworld <chr>, species <chr>, films <list>,
# vehicles <list>, starships <list># A tibble: 2 × 14
name height mass hair_color skin_color eye_color birth_year sex gender
<chr> <int> <dbl> <chr> <chr> <chr> <dbl> <chr> <chr>
1 C-3PO 167 75 <NA> gold yellow 112 none mascu…
2 Luke Sky… 172 77 blond fair blue 19 male mascu…
# ℹ 5 more variables: homeworld <chr>, species <chr>, films <list>,
# vehicles <list>, starships <list>Paréntesis: sample()
La función slice_sample() es simplemente una integración de {tidyverse} de la función básica de R conocida como sample() que nos permite muestrear elementos
Por ejemplo, vamos a muestrear 10 tiradas de un dado, indicándole
- soporte de nuestra variable aleatorio (valores permitidos en
x) - tamaño muestral (
size) - reemplazamiento (si
TRUEentonces pueden salir repetidas, como en el caso del dado)
Paréntesis: sample()
La opción anterior lo que genera son sucesos de una variable aleatoria equiprobable pero al igual que antes, podemos asignarle un vector de probabilidades o función de masa concreta con el argumento prob = ...
Paréntesis: sample()
¿Cómo harías el siguiente enunciado?
Supongamos que en una ciudad se han estudiado episodios de gripe estacional. Sean las variables aleatorias \(X_m\) y \(X_p\) tal que \(X_m=1\) si la madre tiene gripe, \(X_m=0\) si la madre no tiene gripe, \(X_p=1\) si el padre tiene gripe y \(X_p=0\) si el padre no tiene gripe. El modelo teórico asociado a este tipo de epidemias indica que la distribución conjunta viene dada por \(P(X_m = 1, X_p=1)=0.02\), \(P(X_m = 1, X_p=0)=0.08\), \(P(X_m = 1, X_p=0)=0.1\) y \(P(X_m = 0, X_p=0)=0.8\)
Genera una muestra de tamaño \(n = 1000\) (soporte "10", "01", "00" y "11") haciendo uso de runif() y haciendo uso de sample()
💻 Tu turno
Intenta realizar los siguientes ejercicios sin mirar las soluciones
📝 Selecciona solo los personajes que sean humanos y de ojos marrones, para después ordernarlos en altura descendente y peso ascendente.
📝 Extrae aleatoriamente 10 personajes pero de forma que la probabilidad de que salga cada uno sea proporcional a su peso (más pesados, más probable)
Reordenar filas: arrange()
También podemos ordenar filas en función de alguna variable con arrange()
# A tibble: 5 × 6
name height mass hair_color skin_color eye_color
<chr> <int> <dbl> <chr> <chr> <chr>
1 Ratts Tyerel 79 15 none grey, blue unknown
2 Yoda 66 17 white green brown
3 Wicket Systri Warrick 88 20 brown brown brown
4 R2-D2 96 32 <NA> white, blue red
5 R5-D4 97 32 <NA> white, red red Por defecto de menor a mayor pero podemos invertir el orden con desc()
# A tibble: 5 × 3
name height mass
<chr> <int> <dbl>
1 Yarael Poof 264 NA
2 Tarfful 234 136
3 Lama Su 229 88
4 Chewbacca 228 112
5 Roos Tarpals 224 82Eliminar duplicados: distinct()
Muchas veces necesitaremos asegurarnos que no hay duplicados en alguna variable (DNI) y podemos eliminar filas duplicadas con distinct().
# A tibble: 5 × 1
sex
<chr>
1 male
2 none
3 female
4 hermaphroditic
5 <NA> Para mantener todas las columnas de la tabla usaremos .keep_all = TRUE.
# A tibble: 3 × 14
name height mass hair_color skin_color eye_color birth_year sex gender
<chr> <int> <dbl> <chr> <chr> <chr> <dbl> <chr> <chr>
1 Luke Sky… 172 77 blond fair blue 19 male mascu…
2 C-3PO 167 75 <NA> gold yellow 112 none mascu…
3 Leia Org… 150 49 brown light brown 19 fema… femin…
# ℹ 5 more variables: homeworld <chr>, species <chr>, films <list>,
# vehicles <list>, starships <list>Añadir filas: bind_rows()
Por último, podemos concatenar nuevas filas con bind_rows() con las nuevas observaciones en tabla (si no cuadran columnas rellena con ausentes)
# A tibble: 2 × 2
nombre edad
<chr> <dbl>
1 javi 33
2 laura 50💻 Tu turno
Intenta realizar los siguientes ejercicios sin mirar las soluciones
📝 Para saber que valores únicos hay en el color de pelo, elimina duplicados de la variable hair_color, eliminando antes los ausentes de dicha variable.
📝 De los personajes que son humanos y miden más de 160 cm, elimina duplicados en color de ojos, elimina ausentes en peso, selecciona los 3 más altos, y orden de mayor a menor peso. Devuelve la tabla.
Resumen
La clave de {tidyverse} es la legibilidad: es importantísimo que el código se entienda, por nuestro yo el futuro pero también por la transparencia algorítmica hacia los demás
Por ejemplo: quitaremos ausentes de la variable peso, filtraremos los personajes humanos y altura superior a 140cm, sin duplicados en el color de pelo, extrayendo los 5 más altos y obteniendo 2 personajes aleatorios finalmente.
Resumen
La clave de {tidyverse} es la legibilidad: es importantísimo que el código se entienda, por nuestro yo el futuro pero también por la transparencia algorítmica hacia los demás
Por ejemplo: quitaremos ausentes de la variable peso, filtraremos los personajes humanos y altura superior a 140cm, sin duplicados en el color de pelo, extrayendo los 5 más altos y obteniendo 2 personajes aleatorios finalmente.
Resumen
La clave de {tidyverse} es la legibilidad: es importantísimo que el código se entienda, por nuestro yo el futuro pero también por la transparencia algorítmica hacia los demás
Por ejemplo: quitaremos ausentes de la variable peso, filtraremos los personajes humanos y altura superior a 140cm, sin duplicados en el color de pelo, extrayendo los 5 más altos y obteniendo 2 personajes aleatorios finalmente.
Resumen
La clave de {tidyverse} es la legibilidad: es importantísimo que el código se entienda, por nuestro yo el futuro pero también por la transparencia algorítmica hacia los demás
Por ejemplo: quitaremos ausentes de la variable peso, filtraremos los personajes humanos y altura superior a 140cm, sin duplicados en el color de pelo, extrayendo los 5 más altos y obteniendo 2 personajes aleatorios finalmente.
Resumen
La clave de {tidyverse} es la legibilidad: es importantísimo que el código se entienda, por nuestro yo el futuro pero también por la transparencia algorítmica hacia los demás
Por ejemplo: quitaremos ausentes de la variable peso, filtraremos los personajes humanos y altura superior a 140cm, sin duplicados en el color de pelo, extrayendo los 5 más altos y obteniendo 2 personajes aleatorios finalmente.
Selección columnas: select()
La opción más sencilla para seleccionar variables por nombre es select(), dando como argumentos los nombres de columnas sin comillas.
# A tibble: 87 × 2
name hair_color
<chr> <chr>
1 Luke Skywalker blond
2 C-3PO <NA>
3 R2-D2 <NA>
4 Darth Vader none
5 Leia Organa brown
6 Owen Lars brown, grey
7 Beru Whitesun Lars brown
8 R5-D4 <NA>
9 Biggs Darklighter black
10 Obi-Wan Kenobi auburn, white
# ℹ 77 more rowsSelección columnas: select()
La función select() nos permite seleccionar varias variables a la vez, incluso concatenando sus nombres como si fuesen índices numéricos
# A tibble: 4 × 6
name height mass hair_color skin_color eye_color
<chr> <int> <dbl> <chr> <chr> <chr>
1 Luke Skywalker 172 77 blond fair blue
2 C-3PO 167 75 <NA> gold yellow
3 R2-D2 96 32 <NA> white, blue red
4 Darth Vader 202 136 none white yellow Selección columnas: select()
Tenemos además palabras reservadas: everything() todas las variables…
# A tibble: 4 × 14
mass homeworld name height hair_color skin_color eye_color birth_year sex
<dbl> <chr> <chr> <int> <chr> <chr> <chr> <dbl> <chr>
1 77 Tatooine Luke … 172 blond fair blue 19 male
2 75 Tatooine C-3PO 167 <NA> gold yellow 112 none
3 32 Naboo R2-D2 96 <NA> white, bl… red 33 none
4 136 Tatooine Darth… 202 none white yellow 41.9 male
# ℹ 5 more variables: gender <chr>, species <chr>, films <list>,
# vehicles <list>, starships <list>…y last_col() para referirnos a la última columna.
# A tibble: 4 × 5
name height mass homeworld starships
<chr> <int> <dbl> <chr> <list>
1 Luke Skywalker 172 77 Tatooine <chr [2]>
2 C-3PO 167 75 Tatooine <chr [0]>
3 R2-D2 96 32 Naboo <chr [0]>
4 Darth Vader 202 136 Tatooine <chr [1]>Selección columnas: select()
También podemos jugar con patrones en el nombre, aquellas que comiencen por un prefijo (starts_with()), terminen con un sufijo (ends_with()), contengan un texto (contains()) o cumplan una expresión regular (matches()).
# variables cuyo nombre acaba en "color" y contengan sexo o género
starwars |> select(ends_with("color"), matches("sex|gender"))# A tibble: 87 × 5
hair_color skin_color eye_color sex gender
<chr> <chr> <chr> <chr> <chr>
1 blond fair blue male masculine
2 <NA> gold yellow none masculine
3 <NA> white, blue red none masculine
4 none white yellow male masculine
5 brown light brown female feminine
6 brown, grey light blue male masculine
7 brown light blue female feminine
8 <NA> white, red red none masculine
9 black light brown male masculine
10 auburn, white fair blue-gray male masculine
# ℹ 77 more rowsSelección columnas: select()
Incluso podemos seleccionar por rango numérico si tenemos variables con un prefijo y números.
Selección columnas: select()
Por último, podemos seleccionar columnas por tipo de dato haciendo uso de where() y dentro una función que devuelva un valor lógico de tipo de dato.
# A tibble: 87 × 11
height mass birth_year name hair_color skin_color eye_color sex gender
<int> <dbl> <dbl> <chr> <chr> <chr> <chr> <chr> <chr>
1 172 77 19 Luke Sk… blond fair blue male mascu…
2 167 75 112 C-3PO <NA> gold yellow none mascu…
3 96 32 33 R2-D2 <NA> white, bl… red none mascu…
4 202 136 41.9 Darth V… none white yellow male mascu…
5 150 49 19 Leia Or… brown light brown fema… femin…
6 178 120 52 Owen La… brown, gr… light blue male mascu…
7 165 75 47 Beru Wh… brown light blue fema… femin…
8 97 32 NA R5-D4 <NA> white, red red none mascu…
9 183 84 24 Biggs D… black light brown male mascu…
10 182 77 57 Obi-Wan… auburn, w… fair blue-gray male mascu…
# ℹ 77 more rows
# ℹ 2 more variables: homeworld <chr>, species <chr>Mover columnas: relocate()
Para facilitar la recolocación de variables tenemos una función para ello, relocate(), indicándole en .after o .before detrás o delante de qué columnas queremos moverlas.
# A tibble: 87 × 14
species name height mass hair_color skin_color eye_color birth_year sex
<chr> <chr> <int> <dbl> <chr> <chr> <chr> <dbl> <chr>
1 Human Luke S… 172 77 blond fair blue 19 male
2 Droid C-3PO 167 75 <NA> gold yellow 112 none
3 Droid R2-D2 96 32 <NA> white, bl… red 33 none
4 Human Darth … 202 136 none white yellow 41.9 male
5 Human Leia O… 150 49 brown light brown 19 fema…
6 Human Owen L… 178 120 brown, gr… light blue 52 male
7 Human Beru W… 165 75 brown light blue 47 fema…
8 Droid R5-D4 97 32 <NA> white, red red NA none
9 Human Biggs … 183 84 black light brown 24 male
10 Human Obi-Wa… 182 77 auburn, w… fair blue-gray 57 male
# ℹ 77 more rows
# ℹ 5 more variables: gender <chr>, homeworld <chr>, films <list>,
# vehicles <list>, starships <list>Renombrar: rename()
A veces también podemos querer modificar la «metainformación» de los datos, renombrando columnas. Para ello usaremos de rename() poniendo primero el nombre nuevo y luego el antiguo.
# A tibble: 87 × 14
nombre altura peso hair_color skin_color eye_color birth_year sex gender
<chr> <int> <dbl> <chr> <chr> <chr> <dbl> <chr> <chr>
1 Luke Sk… 172 77 blond fair blue 19 male mascu…
2 C-3PO 167 75 <NA> gold yellow 112 none mascu…
3 R2-D2 96 32 <NA> white, bl… red 33 none mascu…
4 Darth V… 202 136 none white yellow 41.9 male mascu…
5 Leia Or… 150 49 brown light brown 19 fema… femin…
6 Owen La… 178 120 brown, gr… light blue 52 male mascu…
7 Beru Wh… 165 75 brown light blue 47 fema… femin…
8 R5-D4 97 32 <NA> white, red red NA none mascu…
9 Biggs D… 183 84 black light brown 24 male mascu…
10 Obi-Wan… 182 77 auburn, w… fair blue-gray 57 male mascu…
# ℹ 77 more rows
# ℹ 5 more variables: homeworld <chr>, species <chr>, films <list>,
# vehicles <list>, starships <list>Extraer columnas: pull()
Si observas la salida de los select() sigue siendo una tabla tibble, ya que nos preserva la naturaleza de nuestros datos.
Extraer columnas: pull()
A veces no querremos dicha estructura sino extraer literalmente la columna en un vector, algo que podemos hacer con pull()
[1] "Luke Skywalker" "C-3PO" "R2-D2"
[4] "Darth Vader" "Leia Organa" "Owen Lars"
[7] "Beru Whitesun Lars" "R5-D4" "Biggs Darklighter"
[10] "Obi-Wan Kenobi" "Anakin Skywalker" "Wilhuff Tarkin"
[13] "Chewbacca" "Han Solo" "Greedo"
[16] "Jabba Desilijic Tiure" "Wedge Antilles" "Jek Tono Porkins"
[19] "Yoda" "Palpatine" "Boba Fett"
[22] "IG-88" "Bossk" "Lando Calrissian"
[25] "Lobot" "Ackbar" "Mon Mothma"
[28] "Arvel Crynyd" "Wicket Systri Warrick" "Nien Nunb"
[31] "Qui-Gon Jinn" "Nute Gunray" "Finis Valorum"
[34] "Padmé Amidala" "Jar Jar Binks" "Roos Tarpals"
[37] "Rugor Nass" "Ric Olié" "Watto"
[40] "Sebulba" "Quarsh Panaka" "Shmi Skywalker"
[43] "Darth Maul" "Bib Fortuna" "Ayla Secura"
[46] "Ratts Tyerel" "Dud Bolt" "Gasgano"
[49] "Ben Quadinaros" "Mace Windu" "Ki-Adi-Mundi"
[52] "Kit Fisto" "Eeth Koth" "Adi Gallia"
[55] "Saesee Tiin" "Yarael Poof" "Plo Koon"
[58] "Mas Amedda" "Gregar Typho" "Cordé"
[61] "Cliegg Lars" "Poggle the Lesser" "Luminara Unduli"
[64] "Barriss Offee" "Dormé" "Dooku"
[67] "Bail Prestor Organa" "Jango Fett" "Zam Wesell"
[70] "Dexter Jettster" "Lama Su" "Taun We"
[73] "Jocasta Nu" "R4-P17" "Wat Tambor"
[76] "San Hill" "Shaak Ti" "Grievous"
[79] "Tarfful" "Raymus Antilles" "Sly Moore"
[82] "Tion Medon" "Finn" "Rey"
[85] "Poe Dameron" "BB8" "Captain Phasma" 💻 Tu turno
Intenta realizar los siguientes ejercicios sin mirar las soluciones
📝 Filtra el conjunto de personajes y quédate solo con aquellos que en la variable height no tengan un dato ausente. Con los datos obtenidos del filtro anterior, selecciona solo las variables name, height, así como todas aquellas variables que CONTENGAN la palabra color en su nombre.
📝 Con los datos obtenidos del ejercicio anterior, traduce el nombre de las columnas a castellano
📝 Con los datos obtenidos del ejercicio anterior, coloca la variable de color de pelo justo detrás de la variable de nombres.
📝 Con los datos obtenidos del ejercicio anterior, comprueba cuántas modalidades únicas hay en la variable de color de pelo (sin usar unique()).
📝 Del conjunto de datos originales, elimina las columnas de tipo lista, y tras ello elimina duplicados en la variable eye_color. Tras eliminar duplicados extrae dicha columna en un vector.
Modificar columnas: mutate()
En muchas ocasiones querremos modificar o crear variables con mutate().
Vamos a crear por ejemplo una nueva variable height_m con la altura en metros.
# A tibble: 87 × 15
name height mass hair_color skin_color eye_color birth_year sex gender
<chr> <int> <dbl> <chr> <chr> <chr> <dbl> <chr> <chr>
1 Luke Sk… 172 77 blond fair blue 19 male mascu…
2 C-3PO 167 75 <NA> gold yellow 112 none mascu…
3 R2-D2 96 32 <NA> white, bl… red 33 none mascu…
4 Darth V… 202 136 none white yellow 41.9 male mascu…
5 Leia Or… 150 49 brown light brown 19 fema… femin…
6 Owen La… 178 120 brown, gr… light blue 52 male mascu…
7 Beru Wh… 165 75 brown light blue 47 fema… femin…
8 R5-D4 97 32 <NA> white, red red NA none mascu…
9 Biggs D… 183 84 black light brown 24 male mascu…
10 Obi-Wan… 182 77 auburn, w… fair blue-gray 57 male mascu…
# ℹ 77 more rows
# ℹ 6 more variables: homeworld <chr>, species <chr>, films <list>,
# vehicles <list>, starships <list>, height_m <dbl>Modificar columnas: mutate()
Además con los argumentos opcionales podemos recolocar la columna modificada
# A tibble: 87 × 16
height_m IMC name height mass hair_color skin_color eye_color birth_year
<dbl> <dbl> <chr> <int> <dbl> <chr> <chr> <chr> <dbl>
1 1.72 26.0 Luke … 172 77 blond fair blue 19
2 1.67 26.9 C-3PO 167 75 <NA> gold yellow 112
3 0.96 34.7 R2-D2 96 32 <NA> white, bl… red 33
4 2.02 33.3 Darth… 202 136 none white yellow 41.9
5 1.5 21.8 Leia … 150 49 brown light brown 19
6 1.78 37.9 Owen … 178 120 brown, gr… light blue 52
7 1.65 27.5 Beru … 165 75 brown light blue 47
8 0.97 34.0 R5-D4 97 32 <NA> white, red red NA
9 1.83 25.1 Biggs… 183 84 black light brown 24
10 1.82 23.2 Obi-W… 182 77 auburn, w… fair blue-gray 57
# ℹ 77 more rows
# ℹ 7 more variables: sex <chr>, gender <chr>, homeworld <chr>, species <chr>,
# films <list>, vehicles <list>, starships <list>Modificar columnas: mutate()
Importante…
Cuando aplicamos mutate(), debemos de acordarnos que las operaciones se realizan de manera vectorial, elemento a elemento, por lo que la función que usemos dentro debe devolver un vector de igual longitud. En caso contrario, devolverá una constante
# A tibble: 87 × 15
constante name height mass hair_color skin_color eye_color birth_year sex
<dbl> <chr> <int> <dbl> <chr> <chr> <chr> <dbl> <chr>
1 97.3 Luke… 172 77 blond fair blue 19 male
2 97.3 C-3PO 167 75 <NA> gold yellow 112 none
3 97.3 R2-D2 96 32 <NA> white, bl… red 33 none
4 97.3 Dart… 202 136 none white yellow 41.9 male
5 97.3 Leia… 150 49 brown light brown 19 fema…
6 97.3 Owen… 178 120 brown, gr… light blue 52 male
7 97.3 Beru… 165 75 brown light blue 47 fema…
8 97.3 R5-D4 97 32 <NA> white, red red NA none
9 97.3 Bigg… 183 84 black light brown 24 male
10 97.3 Obi-… 182 77 auburn, w… fair blue-gray 57 male
# ℹ 77 more rows
# ℹ 6 more variables: gender <chr>, homeworld <chr>, species <chr>,
# films <list>, vehicles <list>, starships <list>Recategorizar: if_else()
También podemos combinar mutate() con la expresión de control if_else() para recategorizar la variable: si se cumple una condición, hace una cosa, en caso contrario otra.
starwars |>
mutate(human = if_else(species == "Human", "Human", "Not Human"),
.after = name) |>
select(name:mass)# A tibble: 87 × 4
name human height mass
<chr> <chr> <int> <dbl>
1 Luke Skywalker Human 172 77
2 C-3PO Not Human 167 75
3 R2-D2 Not Human 96 32
4 Darth Vader Human 202 136
5 Leia Organa Human 150 49
6 Owen Lars Human 178 120
7 Beru Whitesun Lars Human 165 75
8 R5-D4 Not Human 97 32
9 Biggs Darklighter Human 183 84
10 Obi-Wan Kenobi Human 182 77
# ℹ 77 more rowsRecategorizar: case_when()
Para recategorizaciones más complejas tenemos case_when(), por ejemplo, para crear una categoría de los personajes en función de su altura.
starwars |>
drop_na(height) |>
mutate(altura = case_when(height < 120 ~ "enanos",
height < 160 ~ "bajito",
height < 180 ~ "normal",
height < 200 ~ "alto",
TRUE ~ "gigante"), .before = name)# A tibble: 81 × 15
altura name height mass hair_color skin_color eye_color birth_year sex
<chr> <chr> <int> <dbl> <chr> <chr> <chr> <dbl> <chr>
1 normal Luke S… 172 77 blond fair blue 19 male
2 normal C-3PO 167 75 <NA> gold yellow 112 none
3 enanos R2-D2 96 32 <NA> white, bl… red 33 none
4 gigante Darth … 202 136 none white yellow 41.9 male
5 bajito Leia O… 150 49 brown light brown 19 fema…
6 normal Owen L… 178 120 brown, gr… light blue 52 male
7 normal Beru W… 165 75 brown light blue 47 fema…
8 enanos R5-D4 97 32 <NA> white, red red NA none
9 alto Biggs … 183 84 black light brown 24 male
10 alto Obi-Wa… 182 77 auburn, w… fair blue-gray 57 male
# ℹ 71 more rows
# ℹ 6 more variables: gender <chr>, homeworld <chr>, species <chr>,
# films <list>, vehicles <list>, starships <list>💻 Tu turno
Intenta realizar los siguientes ejercicios sin mirar las soluciones
📝 Selecciona solo las variables nombre, altura y así como todas aquellas variables relacionadas con el color, a la vez que te quedas solo con aquellos que no tengan ausente en la altura.
📝 Con los datos obtenidos del ejercicio anterior, traduce el nombre de las columnas a castellano.
📝 Con los datos obtenidos del ejercicio anterior, coloca la variable de color de pelo justo detrás de la variable de nombres.
📝 Con los datos originales, comprueba cuántas modalidades únicas hay en la variable de color de pelo.
📝 Del dataset original, selecciona solo las variables numéricas y de tipo texto. Tras ello define una nueva variable llamada under_18 que nos recategorice la variable de edad: TRUE si es menor de edad y FALSE en caso contrario
📝 Del dataset original, crea una nueva columna llamada auburn (cobrizo/caoba) que nos diga TRUE si el color de pelo contiene dicha palabra y FALSE en caso contrario (reminder str_detect()).
📝 Del dataset original, incluye una columna que calcule el IMC. Tras ello, crea una nueva variable que valga NA si no es humano, delgadez por debajo de 18, normal entre 18 y 30, sobrepeso por encima de 30.
🐣 Caso práctico
Haciendo uso de todo lo aprendido, vamos a proceder a crear una tabla con datos de bebés de tamaño n = 20 en donde simulemos el sexo de los bebés y su peso
- Crea un
tibblecon dos columnas, una llamadaid_bebey otra llamadasexo. En el primer caso debe ir de 1 a 20. En el segundo caso, simula su sexo de manera que haya un 0.5 de probabilidad dechicoy 0.5 dechica.
- Conocido el sexo, crea una tercera columna llamada
pesoen la que simules dicho valor. Supondremos que para los chicos el peso sigue una distribución \(N(\mu = 3.266kg, \sigma = 0.514)\) y que para las chicas sigue una distribución \(N(\mu = 3.155kg, \sigma = 0.495)\).
Contar: count()
Hasta ahora solo hemos transformado o consultado los datos pero no hemos generado estadísticas. Empecemos por lo sencillo: ¿cómo contar (frecuencias)?
Cuando lo usamos en solitario count() nos devolverá simplemente el número de registros , pero cuando lo usamos con variables count() calcula lo que se conoce como frecuencias: número de elementos de cada modalidad.
Contar: count()
Además si pasamos varias variables nos calcula lo que se conoce como una tabla de contigencia. Con sort = TRUE nos devolverá el conteo ordenado (más frecuentes primero).
Agrupar: group_by()
Una de las funciones más potentes a combinar con las acciones vistas es group_by(), que nos permitirá agrupar nuestros registros previamente
Agrupar: group_by()
Cuando apliquemos group_by() es importante entender que NO MODIFICA los datos, sino que nos crea una variable de grupo (subtablas por cada grupo) que modificará las acciones futuras: las operaciones se aplicarán a cada subtabla por separado
Por ejemplo, imaginemos que queremos extraer el personaje más alto con slice_max().
# A tibble: 1 × 14
name height mass hair_color skin_color eye_color birth_year sex gender
<chr> <int> <dbl> <chr> <chr> <chr> <dbl> <chr> <chr>
1 Yarael P… 264 NA none white yellow NA male mascu…
# ℹ 5 more variables: homeworld <chr>, species <chr>, films <list>,
# vehicles <list>, starships <list>Agrupar: group_by()
¿Y si queremos extraer el personaje más alto pero…de cada uno de los sexos?
# A tibble: 5 × 14
name height mass hair_color skin_color eye_color birth_year sex gender
<chr> <int> <dbl> <chr> <chr> <chr> <dbl> <chr> <chr>
1 Taun We 213 NA none grey black NA fema… femin…
2 Jabba De… 175 1358 <NA> green-tan… orange 600 herm… mascu…
3 Yarael P… 264 NA none white yellow NA male mascu…
4 IG-88 200 140 none metal red 15 none mascu…
5 Gregar T… 185 85 black dark brown NA <NA> <NA>
# ℹ 5 more variables: homeworld <chr>, species <chr>, films <list>,
# vehicles <list>, starships <list>Agrupar: group_by()
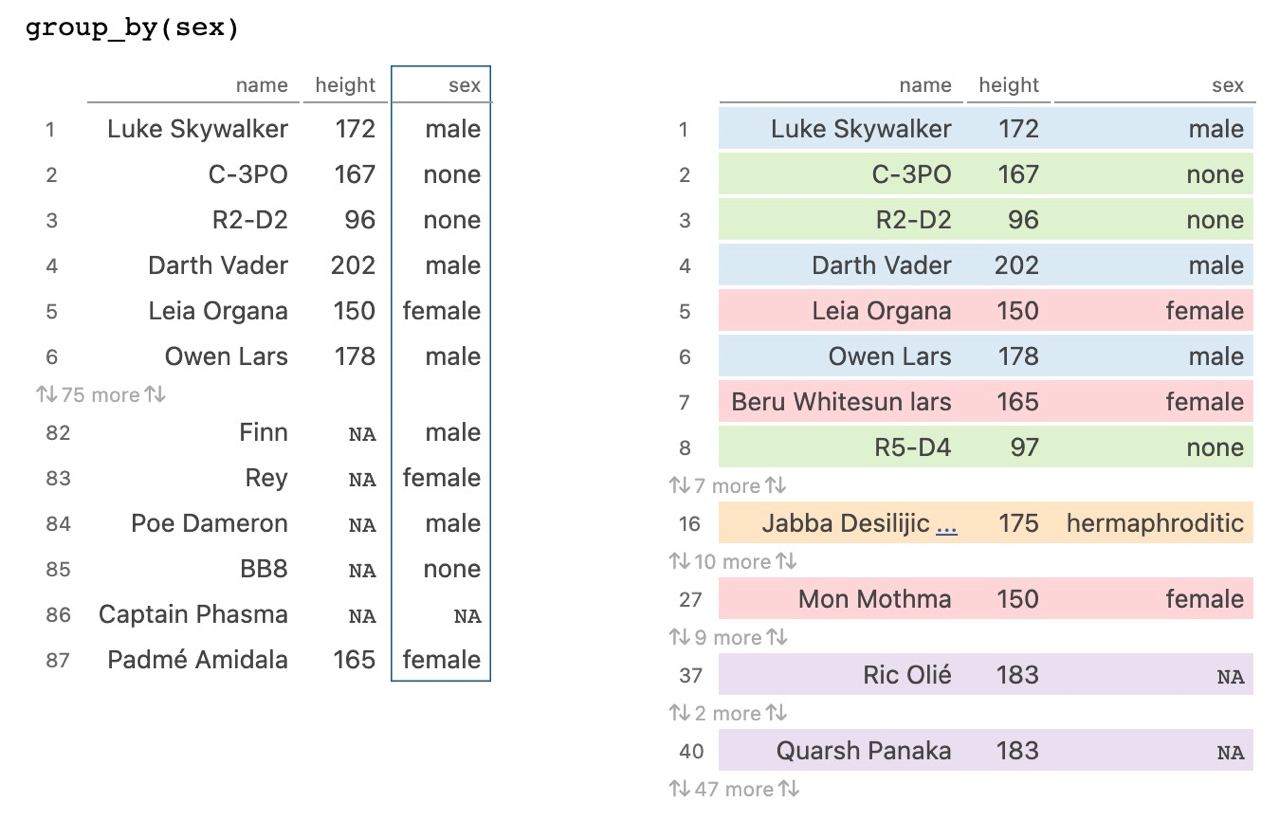
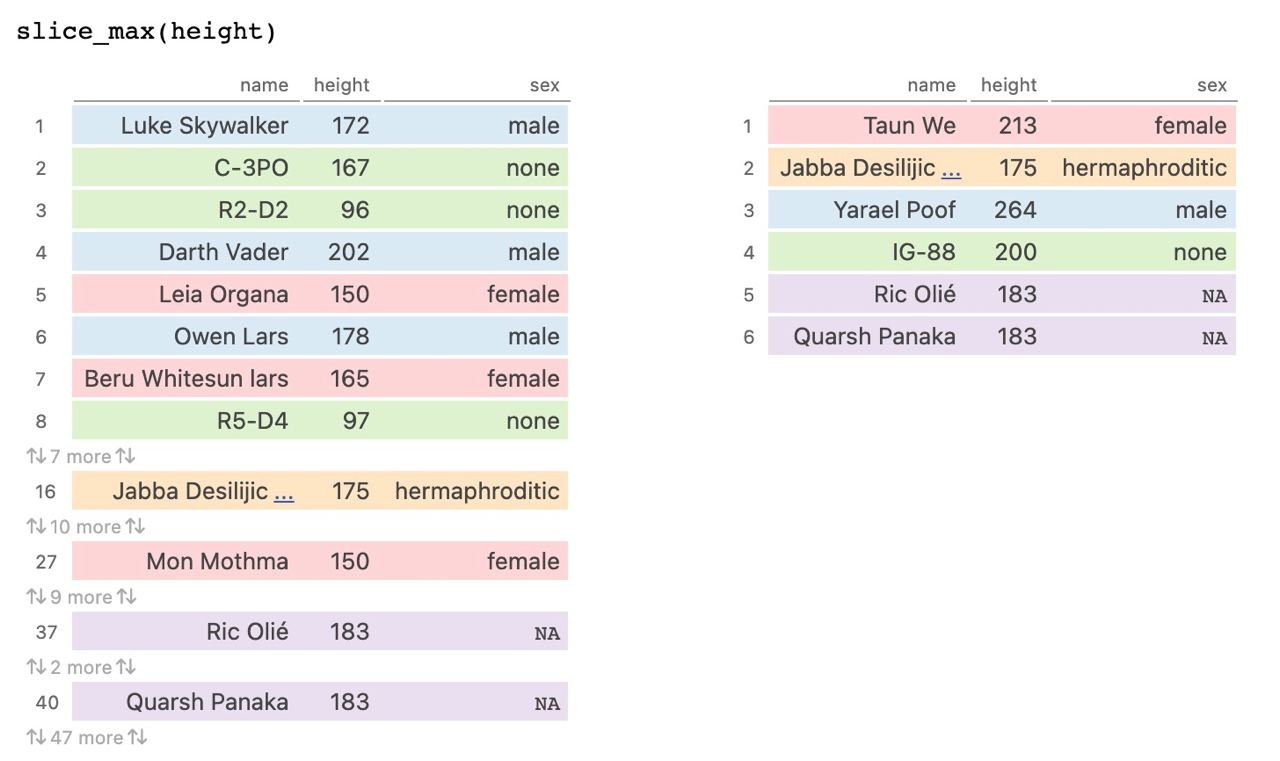
La web https://tidydatatutor.com/ permite visualizar las operaciones de {tidyverse} (con el pipe antiguo)
Agrupar: group_by()
Importante
Recuerda siempre hacer ungroup para eliminar la variable de grupo creada
En la nueva versión de {dplyr} ahora se permite incluir la variable de grupo en la llamada a muchas funciones con el argumento by = ... o .by = ...
# A tibble: 5 × 6
name height mass hair_color skin_color eye_color
<chr> <int> <dbl> <chr> <chr> <chr>
1 Yarael Poof 264 NA none white yellow
2 IG-88 200 140 none metal red
3 Taun We 213 NA none grey black
4 Jabba Desilijic Tiure 175 1358 <NA> green-tan, brown orange
5 Gregar Typho 185 85 black dark brown Fila-a-fila: rowwise()
Una opción muy útil usada antes de una operación también es rowwise(): toda operación que venga después se aplicará en cada fila por separado. Por ejemplo, vamos a definir un conjunto dummy de notas.
Si aplicamos la media directamente el valor será idéntico ya que nos ha hecho la media global, pero nos gustaría sacar una media por registro. Para eso usaremos rowwise()
Resumir: summarise()
Por último tenemos summarise(), que nos permitirá sacar resúmenes estadísticos. Por ejemplo, vamos a calcular la media de las alturas.
# A tibble: 1 × 1
media_altura
<dbl>
1 175.Cuidado
Fíjate que mutate() devuelve tantas filas como registros originales, mientras que con summarise() calcula un nuevo dataset de resumen, solo incluyendo aquello que esté indicado.
Resumir: summarise()
Si además esto lo combinamos con la agrupación de group_by() o .by = ..., en pocas líneas de código puedes obtener estadísticas desagreagadas
Resumir: reframe()
En el nuevo {dplyr} han incluido reframe() para evitar problemas de summarise() cuando devolvemos más de un valor por variable.
Warning: Returning more (or less) than 1 row per `summarise()` group was deprecated in
dplyr 1.1.0.
ℹ Please use `reframe()` instead.
ℹ When switching from `summarise()` to `reframe()`, remember that `reframe()`
always returns an ungrouped data frame and adjust accordingly.# A tibble: 5 × 1
`quantile(mass)`
<dbl>
1 15
2 55.6
3 79
4 84.5
5 1358 Selectores: across()
Un truco es hacer uso de selectores across() y where(). El primero nos permite actuar sobre varias columnas por nombre (con mutate() o summarise())
# A tibble: 5 × 2
sex medias$height $mass
<chr> <dbl> <dbl>
1 male 179. 80.2
2 none 131. 69.8
3 female 172. 54.7
4 hermaphroditic 175 1358
5 <NA> 175 81 El segundo, where(), nos permite hacer lo mismo pero seleccionando por tipo.
# A tibble: 6 × 5
sex gender height mass birth_year
<chr> <chr> <dbl> <dbl> <dbl>
1 male masculine 179. 80.2 84.8
2 none masculine 140 69.8 53.3
3 female feminine 172. 54.7 47.2
4 hermaphroditic masculine 175 1358 600
5 <NA> <NA> 175 81 NaN
6 none feminine 96 NaN NaN 💻 Tu turno
Intenta realizar los siguientes ejercicios sin mirar las soluciones
📝 Calcula cuántos personajes hay de cada especie, ordenados de más a menor frecuencia.
📝 Tras eliminar ausentes en las variables de peso y estatura, añade una nueva variable que nos calcule el IMC de cada personaje, y determina el IMC medio de nuestros personajes desagregada por sexo
📝 Obtén la edad del personaje más joven y más viejo de cada sexo.
🐣 Caso práctico
Vamos antes a hacer un repaso de lo aprendido en {tidyverse}
- Carga la tabla billboard del paquete
{tidyr}.
- Antes de nada, selecciona solo las primeras 52 semanas. Tras ello convierte el dataset a tidydata con los formatos y tipos adecuados para cada variable
🐣 Caso práctico
- Determina cuántas canciones tiene cada artistal
- Determina las 5 canciones que más semanas aparecen en la lista de éxitos.
🐣 Caso práctico
- Determina el artista con más canciones en la lista
🐣 Caso práctico
- Obtén una tabla resumen con el ranking medio de cada artista (contando solo el ranking más alto alcanzado por sus canciones), así como el número de canciones (distintas) que ha colocado en el top 100.
🐣 Caso práctico (2)
Vamos a usar el dataset biopsy que podemos encontrar en el agregador de datasets https://vincentarelbundock.github.io/Rdatasets/index.html. El dataset contiene datos de 699 pacientes a lo que se les realizó una biopsia de pecho, obteniendo 11 variables (una que hace de id y 10 escalas medidas de 1 a 10)
Puedes ver la documentación en https://vincentarelbundock.github.io/Rdatasets/doc/MASS/biopsy.html
- Crea de cero un informe en Quarto donde hagas los ejercicios posteriores (usa un archivo de estilos para que quede estético)
- Importa el
.csvdesde la propia web (a través del enlace del archivo)
- Habrás visto que hay 12 columnas en realidad importadas (la primera nos sobra ya que es solo un contador de filas). Vuelve a cargarlo seleccionando en la carga solo desde
IDhastaclass. Convierte a tidydata
🐣 Caso práctico (2)
- La variable
IDdebería ser identificador de cada registro: elimina duplicados por dicha variable del dataset anterior.
- Del dataset anterior filtra solo los pacientes con tumor maligno y la variable
V9con valor 4 o inferior, eliminando además cualquier registro que contenga ausente en cualquiera de la variables.
- Del dataset anterior obtén una muestra del 20% de los datos (cada registro puede ser elegido con la misma probabilidad), y órdenalos por de mayor a menor por la variable
V1y, en caso de empate, de menor a mayor por la variableV2
Clase 4: importar/exportar en R. Comunicar
Importar/exportar. Comunicar con Quarto
Clase 4: importar y exportar
Repasando tidydata. Importar/exportar datos en R
Importar/exportar datos
Hasta ahora solo hemos usado datos cargados ya en paquetes pero muchas veces necesitaremos importar datos de manera externa. Una de las principales fortalezas de R es que podemos importar datos de manera muy sencilla en distintos formatos:
- Formatos nativos de R: formatos
.rda,.RDatay.rds
- Datos rectangulares (tabulados): formatos
.csvy.tsv
- Datos sin tabular: formato
.txt
- Datos en excel: formatos
.xlsy.xlsx
- Datos desde SAS/Stata/SPSS: formatos
.sas7bdat,.savy.dat
- Datos desde API: Google Drive, aemet, catastro, twitter, spotify, etc
Datos
Puedes descargar la carpeta de datos desde Google Drive.
Recuerda descomprimir la carpeta. Lo recomendable es crear un proyecto, donde guardes los scripts y los datos.
Formatos nativos de R
Los ficheros más simples para importar en R (y que suele ocupar menos espacio en disco) son sus propias extensiones nativas: archivos con formatos .RData, .rda y .rds.
Para cargar los dos primeros simplemente necesitamos usar la función nativa load() indicándole la ruta del archivo.
- Archivo
.RData: vamos a importar un dataset con las distintas características de los viajeros del Titanic, incluyendo quién sobrevivió y quién murió.
# A tibble: 5 × 12
PassengerId Survived Pclass Name Sex Age SibSp Parch Ticket Fare Cabin
<int> <int> <int> <fct> <fct> <dbl> <int> <int> <fct> <dbl> <fct>
1 1 0 3 Braund… male 22 1 0 A/5 2… 7.25 ""
2 2 1 1 Cuming… fema… 38 1 0 PC 17… 71.3 "C85"
3 3 1 3 Heikki… fema… 26 0 0 STON/… 7.92 ""
4 4 1 1 Futrel… fema… 35 1 0 113803 53.1 "C12…
5 5 0 3 Allen,… male 35 0 0 373450 8.05 ""
# ℹ 1 more variable: Embarked <fct>Formatos nativos de R
- Archivo
.rda: vamos a importar un dataset con datos de cáncer de pecho de Royston and Altman (2013), incluyendo 2982 pacientes y sus características
# A tibble: 7 × 15
pid year age meno size grade nodes pgr er hormon chemo rtime recur
<int> <int> <int> <int> <fct> <int> <int> <int> <int> <int> <int> <dbl> <int>
1 1 1992 74 1 <=20 3 0 35 291 0 0 1799 0
2 2 1984 79 1 20-50 3 0 36 611 0 0 2828 0
3 3 1983 44 0 <=20 2 0 138 0 0 0 6012 0
4 4 1985 70 1 20-50 3 0 0 12 0 0 2624 0
5 5 1983 75 1 <=20 3 0 260 409 0 0 4915 0
6 6 1983 52 0 <=20 3 0 139 303 0 0 5888 0
7 7 1993 40 0 <=20 2 0 13 4 0 0 2491 0
# ℹ 2 more variables: dtime <dbl>, death <int>Formatos nativos de R
- Archivo
.rds: para este tipo debemos usarreadRDS(), y necesitamos incorporar un argumentofilecon la ruta. En este caso vamos a importar datos de cáncer de pulmón del North Central Cancer Treatment Group.
# A tibble: 5 × 10
inst time status age sex ph.ecog ph.karno pat.karno meal.cal wt.loss
<dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 3 306 2 74 1 1 90 100 1175 NA
2 3 455 2 68 1 0 90 90 1225 15
3 3 1010 1 56 1 0 90 90 NA 15
4 5 210 2 57 1 1 90 60 1150 11
5 1 883 2 60 1 0 100 90 NA 0Importante
Las rutas deben ir siempre sin espacios, ni eñes, ni tildes. Y fíjate que los archivos cargados con load() se cargan automáticamente en el environment (con el nombre guardado originalmente, sin necesidad de asignarlo a nada), pero las funciones read() solo se carga de manera local (sino se guarda, no existe a futuro)
Datos rectangulares: readr
El paquete {readr} dentro del entorno {tidyverse} contiene distintas funciones útiles para la carga de datos rectangulares (sin formatear).
read_csv(): archivos.csvcuyo separador sea la comaread_csv2(): punto y comaread_tsv(): tabulador.read_table(): espacio.read_delim(): función genérica para archivos delimitados por caracteres.

Todos necesitan como argumento la ruta del archivo amén de otros opcionales (saltar o no cabecera, decimales, etc). Ver más en https://readr.tidyverse.org/
Datos tabulados (.csv, .tsv)
La principal ventaja de {readr} es que automatiza el formateo para pasar de un archivo plano (sin formato) a un tibble (en filas y columnas, con formato).
- Archivo
.csv: conread_csv()cargaremos archivos separados por coma, pasando como argumento la ruta enfile = .... Vamos a importar el datasetchickens.csv(sobre pollos de dibujos animados, why not). Si te fijas en la salida nos proporciona el tipo de variables.
# A tibble: 5 × 4
chicken sex eggs_laid motto
<chr> <chr> <dbl> <chr>
1 Foghorn Leghorn rooster 0 That's a joke, ah say, that's a jok…
2 Chicken Little hen 3 The sky is falling!
3 Ginger hen 12 Listen. We'll either die free chick…
4 Camilla the Chicken hen 7 Bawk, buck, ba-gawk.
5 Ernie The Giant Chicken rooster 0 Put Captain Solo in the cargo hold. Datos tabulados (.csv, .tsv)
El formato de las variables normalmente lo hará read_csv() de forma automática, y podemos consultarlo con spec()
Datos tabulados (.csv, .tsv)
Aunque lo haga normalmente bien de forma automática podemos especificar el formato explícitamente en col_types = list() (en formato lista, con col_xxx() para cada tipo de variable, por ejemplo una la pondremos como cualitativa o factor).
chickens <-
read_csv(file = "./datos/chickens.csv",
col_types = list(col_character(), col_factor(), col_double(), col_character()))
chickens# A tibble: 5 × 4
chicken sex eggs_laid motto
<chr> <fct> <dbl> <chr>
1 Foghorn Leghorn rooster 0 That's a joke, ah say, that's a jok…
2 Chicken Little hen 3 The sky is falling!
3 Ginger hen 12 Listen. We'll either die free chick…
4 Camilla the Chicken hen 7 Bawk, buck, ba-gawk.
5 Ernie The Giant Chicken rooster 0 Put Captain Solo in the cargo hold. Datos tabulados (.csv, .tsv)
Incluso podemos indicar que variables que queremos seleccionar (sin ocupar memoria), indicándoselo en col_select = ...
Datos sin tabular (.txt)
Vamos a usar de nuevo read_csv() con el archivo massey-rating.txt.
# A tibble: 10 × 1
`UCC PAY LAZ KPK RT COF BIH DII ENG ACU Rank Team Conf`
<chr>
1 1 1 1 1 1 1 1 1 1 1 1 Ohio St B10
2 2 2 2 2 2 2 2 2 4 2 2 Oregon P12
3 3 4 3 4 3 4 3 4 2 3 3 Alabama SEC
4 4 3 4 3 4 3 5 3 3 4 4 TCU B12
5 6 6 6 5 5 7 6 5 6 11 5 Michigan St B10
6 7 7 7 6 7 6 11 8 7 8 6 Georgia SEC
7 5 5 5 7 6 8 4 6 5 5 7 Florida St ACC
8 8 8 9 9 10 5 7 7 10 7 8 Baylor B12
9 9 11 8 13 11 11 12 9 14 9 9 Georgia Tech ACC
10 13 10 13 11 8 9 10 11 9 10 10 Mississippi SEC Si te fijas nos interpreta todo como una sola columna: no tiene comas el archivo y no sabe por donde separar
¿Qué sucede cuando el separador no es el correcto?
Datos sin tabular (.txt)
Para ello tenemos
read_csv2()cuando el separador sea el punto y coma,read_tsv()cuando el sea un tabulador yread_table()cuando el sea un espacioread_delim()en general
# A tibble: 10 × 13
UCC PAY LAZ KPK RT COF BIH DII ENG ACU Rank Team Conf
<dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <chr> <chr>
1 1 1 1 1 1 1 1 1 1 1 1 Ohio St
2 2 2 2 2 2 2 2 2 4 2 2 Oreg… P12
3 3 4 3 4 3 4 3 4 2 3 3 Alab… SEC
4 4 3 4 3 4 3 5 3 3 4 4 TCU B12
5 6 6 6 5 5 7 6 5 6 11 5 Mich… St
6 7 7 7 6 7 6 11 8 7 8 6 Geor… SEC
7 5 5 5 7 6 8 4 6 5 5 7 Flor… St
8 8 8 9 9 10 5 7 7 10 7 8 Bayl… B12
9 9 11 8 13 11 11 12 9 14 9 9 Geor… Tech
10 13 10 13 11 8 9 10 11 9 10 10 Miss… SEC Datos en excel (.xls, .xlsx)
Otro de los paquetes fundamentales de importación será el paquete {readxl} para importar datos desde una Excel. Tres funciones serán claves:
read_xls()específica para.xls,read_xlsx()específica para.xlsxread_excel(): para ambas
Vamos a importar deaths.xlsx con registros de fallecimientos de famosos
# A tibble: 8 × 6
`Lots of people` ...2 ...3 ...4 ...5 ...6
<chr> <chr> <chr> <chr> <chr> <chr>
1 simply cannot resist writing <NA> <NA> <NA> <NA> some not…
2 at the top <NA> of their sp…
3 or merging <NA> <NA> <NA> cells
4 Name Profession Age Has kids Date of birth Date of …
5 David Bowie musician 69 TRUE 17175 42379
6 Carrie Fisher actor 60 TRUE 20749 42731
7 Chuck Berry musician 90 TRUE 9788 42812
8 Bill Paxton actor 61 TRUE 20226 42791 Datos en excel (.xls, .xlsx)
# A tibble: 8 × 6
`Lots of people` ...2 ...3 ...4 ...5 ...6
<chr> <chr> <chr> <chr> <chr> <chr>
1 simply cannot resist writing <NA> <NA> <NA> <NA> some not…
2 at the top <NA> of their sp…
3 or merging <NA> <NA> <NA> cells
4 Name Profession Age Has kids Date of birth Date of …
5 David Bowie musician 69 TRUE 17175 42379
6 Carrie Fisher actor 60 TRUE 20749 42731
7 Chuck Berry musician 90 TRUE 9788 42812
8 Bill Paxton actor 61 TRUE 20226 42791 Algo por desgracia muy habitual es que haya algún tipo de comentario o texto al inicio del archivo, teniendo que saltarnos dichas filas.
Datos en excel (.xls, .xlsx)
Podemos saltarnos dichas filas directamente en la carga con skip = ... (indicando el número de filas que nos saltamos)
# A tibble: 5 × 6
Name Profession Age `Has kids` `Date of birth` `Date of death`
<chr> <chr> <chr> <chr> <dttm> <chr>
1 David Bowie musician 69 TRUE 1947-01-08 00:00:00 42379
2 Carrie Fisher actor 60 TRUE 1956-10-21 00:00:00 42731
3 Chuck Berry musician 90 TRUE 1926-10-18 00:00:00 42812
4 Bill Paxton actor 61 TRUE 1955-05-17 00:00:00 42791
5 Prince musician 57 TRUE 1958-06-07 00:00:00 42481 Datos en excel (.xls, .xlsx)
Además con col_names = ... podemos renombrar ya las columnas en la importación (si proporcionamos nombres asume la 1ª línea ya como un dato)
# A tibble: 7 × 6
name profession age kids birth death
<chr> <chr> <chr> <chr> <dttm> <chr>
1 David Bowie musician 69 TRUE 1947-01-08 00:00:00 42379
2 Carrie Fisher actor 60 TRUE 1956-10-21 00:00:00 42731
3 Chuck Berry musician 90 TRUE 1926-10-18 00:00:00 42812
4 Bill Paxton actor 61 TRUE 1955-05-17 00:00:00 42791
5 Prince musician 57 TRUE 1958-06-07 00:00:00 42481
6 Alan Rickman actor 69 FALSE 1946-02-21 00:00:00 42383
7 Florence Henderson actor 82 TRUE 1934-02-14 00:00:00 42698Datos en excel (.xls, .xlsx)
En ocasiones las fechas de Excel están mal formateadas (sorpresa): podemos hacer uso de convertToDate() del paquete {openxlsx} para convertirlo
# A tibble: 7 × 6
name profession age kids birth death
<chr> <chr> <chr> <chr> <dttm> <date>
1 David Bowie musician 69 TRUE 1947-01-08 00:00:00 2016-01-10
2 Carrie Fisher actor 60 TRUE 1956-10-21 00:00:00 2016-12-27
3 Chuck Berry musician 90 TRUE 1926-10-18 00:00:00 2017-03-18
4 Bill Paxton actor 61 TRUE 1955-05-17 00:00:00 2017-02-25
5 Prince musician 57 TRUE 1958-06-07 00:00:00 2016-04-21
6 Alan Rickman actor 69 FALSE 1946-02-21 00:00:00 2016-01-14
7 Florence Henderson actor 82 TRUE 1934-02-14 00:00:00 2016-11-24Datos en excel (.xls, .xlsx)
También podemos cargar un Excel con varias hojas: para indicarle la hoja (bien por su nombre bien por su número) usaremos el argumento sheet = ...
# A tibble: 5 × 11
mpg cyl disp hp drat wt qsec vs am gear carb
<dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 21 6 160 110 3.9 2.62 16.5 0 1 4 4
2 21 6 160 110 3.9 2.88 17.0 0 1 4 4
3 22.8 4 108 93 3.85 2.32 18.6 1 1 4 1
4 21.4 6 258 110 3.08 3.22 19.4 1 0 3 1
5 18.7 8 360 175 3.15 3.44 17.0 0 0 3 2Desde SAS/STATA/SPSS
El paquete {haven} dentro de la órbita tidyverse nos permitirá importar archivos de los 3 software de pago más importantes: SAS, SPSS y Stata
Exportar
De la misma manera que podemos importar también podemos exportar
- exportado en
.RData(opción recomendada para variables guardadas enR). Recuerda que esta extensión solo se podrá usar enR. Para ello nos basta con usarsave(objeto, file = ruta)
Exportar
De la misma manera que podemos importar también podemos exportar
- exportado en
.RDS(opción recomendada para variables guardadas enR). Recuerda que esta extensión solo se podrá usar enR. Para ello nos basta con usarsaveRDS(objeto, file = ruta)
Exportar
De la misma manera que podemos importar también podemos exportar
- exportado en
.csv. Para ello nos basta con usarwrite_csv(objeto, file = ruta)
Exportar
De la misma manera que podemos importar también podemos exportar
- exportado en
.xlsx. Para ello nos basta con usarwrite.xlsx(objeto, file = ruta)del paquete{openxlsx}
Desde web
Una de las principales ventajas de R es que podemos hacer uso de todas las funciones anteriores de importar pero directamente desde una web, sin necesidad de realizar la descarga manual: en lugar de pasarle la ruta local le indicaremos el enlace. Por ejemplo, vamos a descargar los datos de covid del ISCIII (https://cnecovid.isciii.es/covid19/#documentaci%C3%B3n-y-datos)
# A tibble: 700 × 8
provincia_iso sexo grupo_edad fecha num_casos num_hosp num_uci num_def
<chr> <chr> <chr> <date> <dbl> <dbl> <dbl> <dbl>
1 A H 0-9 2020-01-01 0 0 0 0
2 A H 10-19 2020-01-01 0 0 0 0
3 A H 20-29 2020-01-01 0 0 0 0
4 A H 30-39 2020-01-01 0 0 0 0
5 A H 40-49 2020-01-01 0 0 0 0
6 A H 50-59 2020-01-01 0 0 0 0
7 A H 60-69 2020-01-01 0 0 0 0
8 A H 70-79 2020-01-01 0 0 0 0
9 A H 80+ 2020-01-01 0 0 0 0
10 A H NC 2020-01-01 0 0 0 0
# ℹ 690 more rowsDesde google drive
Otra opción disponible (sobre todo si trabajamos con otras personas que trabajan) es importar desde una hoja de cálculo Google Drive, haciendo uso de read_sheet() del paquete {googlesheets4}
La primera vez te pedirá un permiso de tidyverse para interactuar con vuestro drive
Desde API (owid)
Una opción también muy interesante es la carga de datos desde una API: un intermediario entre una app o proveedor datos y nuestro R. Por ejemplo, vamos a cargar la librería {owidR}, que nos permite la descarga de datos de la web https://ourworldindata.org/. La función owid_covid() nos carga sin darnos cuenta más de 300 000 registros con más de 50 variables de 238 países
Desde API (owid)
Este paquete tiene la función owid_search() para buscar datasets por palabras clave, por ejemplo, emissions, dándonos un dataset con el título de la base de datos y su id para luego usarla.
Desde API (aemet)
En muchas ocasiones para conectar con la API tendremos antes que registrarnos y obtener una clave, es el caso del paquete {climaemet} para acceder a datos meteorológicos (https://opendata.aemet.es/centrodedescargas/inicio)
Una vez que tenemos la clave de la API la registramos en nuestro RStudio para poder usarla a futuro
library(climaemet)
# Definir la clave
apikey <- "eyJhbGciOiJIUzI1NiJ9.eyJzdWIiOiJqYXZhbHYwOUB1Y20uZXMiLCJqdGkiOiI4YTU1ODUxMS01MTE3LTQ4MTYtYmM4OS1hYmVkNDhiODBkYzkiLCJpc3MiOiJBRU1FVCIsImlhdCI6MTY2NjQ2OTcxNSwidXNlcklkIjoiOGE1NTg1MTEtNTExNy00ODE2LWJjODktYWJlZDQ4YjgwZGM5Iiwicm9sZSI6IiJ9.HEMR77lZy2ASjmOxJa8ppx2J8Za1IViurMX3p1reVBU"
aemet_api_key(apikey, install = TRUE, overwrite = TRUE)Desde API (aemet)
Con dicho paquete podemos hacer una búsqueda de estaciones para conocer tanto su código postal como su código identificador dentro de la red AEMET
# A tibble: 291 × 7
indicativo indsinop nombre provincia altitud longitud latitud
<chr> <chr> <chr> <chr> <dbl> <dbl> <dbl>
1 0252D "08186" ARENYS DE MAR BARCELONA 74 2.54 41.6
2 0076 "08181" BARCELONA AEROPUERTO BARCELONA 4 2.07 41.3
3 0200E "" BARCELONA, FABRA BARCELONA 408 2.12 41.4
4 0201D "08180" BARCELONA BARCELONA 6 2.2 41.4
5 0149X "08174" MANRESA BARCELONA 291 1.84 41.7
6 0229I "08192" SABADELL AEROPUERTO BARCELONA 146 2.10 41.5
7 0255B "08188" SANTA SUSANNA BARCELONA 40 2.70 41.7
8 0367 "08184" GIRONA AEROPUERTO GIRONA 143 2.76 41.9
9 0370B "" GIRONA, ANTIC INSTITUT GIRONA 95 2.83 42.0
10 0372C "08120" PORQUERES GIRONA 157 2.76 42.1
# ℹ 281 more rowsDesde API (aemet)
Por ejemplo, la estación del aeropuerto de El Prat, Barcelona, es el código "0076"
# A tibble: 23 × 23
idema lon fint prec alt vmax vv dv lat dmax
<chr> <dbl> <dttm> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 0076 2.07 2023-11-27 15:00:00 0 4 8.2 5.2 250 41.3 260
2 0076 2.07 2023-11-27 16:00:00 0 4 8.8 5.7 250 41.3 250
3 0076 2.07 2023-11-27 17:00:00 0 4 7.7 5.5 250 41.3 260
4 0076 2.07 2023-11-27 18:00:00 0 4 6.2 1.8 310 41.3 260
5 0076 2.07 2023-11-27 19:00:00 0 4 5.7 2.9 20 41.3 20
6 0076 2.07 2023-11-27 20:00:00 0 4 6.2 3.1 360 41.3 360
7 0076 2.07 2023-11-27 21:00:00 0 4 5.1 1.5 260 41.3 340
8 0076 2.07 2023-11-27 22:00:00 0 4 5.7 3.8 280 41.3 280
9 0076 2.07 2023-11-27 23:00:00 0 4 9.8 5.3 310 41.3 300
10 0076 2.07 2023-11-28 00:00:00 0 4 7.2 1 360 41.3 300
# ℹ 13 more rows
# ℹ 13 more variables: ubi <chr>, pres <dbl>, hr <dbl>, stdvv <dbl>, ts <dbl>,
# pres_nmar <dbl>, tamin <dbl>, ta <dbl>, tamax <dbl>, tpr <dbl>, vis <dbl>,
# stddv <dbl>, inso <dbl>💻 Tu turno
Intenta realizar los siguientes ejercicios sin mirar las soluciones
📝 El dataset who que hemos usado en ejercicios anteriores, expórtalo a un formato nativo de R en la carpeta datos del proyecto
📝 Carga el dataset who pero desde la carpeta de datos (importa el archivo creado en el ejercicio anterior)
📝 Repite lo mismo (exportar e importar) en 4 formatos: .csv, .xlsx, .sav (spss) y .dta (stata)
Código
# csv
library(readr)
write_csv(who, file = "./datos/who.csv")
who_data <- read_csv(file = "./datos/who.csv")
# excel
library(openxlsx)
write.xlsx(who, file = "./datos/who.xlsx")
who_data <- read_xlsx(path = "./datos/who.xlsx")
# sas y stata
library(haven)
write_sav(who, path = "./datos/who.sav")
who_data <- read_spss(path = "./datos/who.sav")
write_dta(who, path = "./datos/who.dta")
who_data <- read_dta(path = "./datos/who.dta")Clase 4: comunicar
Comunicar resultados: rmd y Quarto
Comunicar: rmd y Quarto
Una de las principales fortalezas de R es la facilidad para generar informes, libros, webs, apuntes y hasta diapositivas (este mismo material por ejemplo). Para ello instalaremos antes
- el paquete
{rmarkdown}(para generar archivos.rmd)
- instalar Quarto (el «nuevo»
.rmdahora como.qmd)

Comunicar: rmd y Quarto
Hasta ahora solo hemos programado en scripts (archivos .R) dentro de proyectos, pero en muchas ocasiones no trabajaremos solos y necesitaremos comunicar los resultados en diferentes formatos:
- apuntes (para nosotros mismos)
- diapositivas
- web
- informes
Para todo ello usaremos Quarto (nuevo rmarkdown)
Comunicar: rmd y Quarto
Los archivos de extensión .qmd (o .rmd) nos permitirán fácilmente combinar:
- Markdown: lenguaje tipado que nos permite crear contenido simple (tipo wordpress, con texto, negritas, cursivas, etc) con un diseño legible.
- Matemáticas (latex): lenguaje para escribir notación matemática como \(x^2\) o \(\sqrt{y}\) o \(\int_{a}^{b} f(x) dx\)
- Código y salidas: podremos no solo mostrar el paso final sino el código que has ido realizando (no solo en
R), con cajitas de código llamadas chunks.
- Imágenes, gráficas, tablas, estilos (css, js), etc.
Comunicar: rmd y Quarto
La principal ventaja de realizar este tipo de material en Quarto/Rmarkdown es que, al hacerlo desde RStudio, puedes generar un informe o una presentación sin salirte del entorno de programación en el que estás trabajando
De esta forma podrás analizar los datos, resumirlos y a la vez comunicarlos con la misma herramienta.
Recientemente el equipo de RStudio desarrolló Quarto, una versión mejorada de Rmarkdown (archivos .qmd), con un formato un poco más estético y simple. Tienes toda la documentación y ejemplos en https://quarto.org/
Nuestro primer informe

Vamos a crear el primer fichero rmarkdown con Quarto con extensión .qmd. Para ello solo necesitaremos hacer click en
File << New File << Quarto Document
Nuestro primer informe

Tras hacerlo nos aparecerán varias opciones de formatos de salida:
- archivo
.pdf - archivo
.html(recomendable): documento dinámico, permite la interacción con el usuario, como una «página web». - archivo
.doc(nada recomendable)
De momento dejaremos marcado el formato HTML que viene por defecto, y escribiremos el título de nuestro documento. Tras ello tendremos nuestro archivo .qmd (ya no es un script .R como los que hemos abierto hasta ahora).
Nuestro primer informe

Deberías tener algo similar a la captura de la imagen con dos modos de edición: Source (con código, la opción recomendada hasta que lo domines) y Visual (más parecido a un blog)
Para ejecutar TODO el documento debes clickar Render on Save y darle a guardar.
Cabecera de un qmd

Deberías haber obtenido una salida en html similar a esta (y se te ha generado en tu ordenador un archivo html)
Nuestro primer informe

Un fichero .qmd se divide básicamente en tres partes:
Cabecera: la parte que tienes al inicio entre
---.Texto: que podremos formatear y mejorar con negritas (escrito como negritas, con doble astérisco al inicio y final), cursivas (cursivas, con barra baja al inicio y final) o destacar nombres de funciones o variables de R. Puedes añadir ecuaciones como \(x^2\) (he escrito
$x^2$, entre dólares).Código R
Cabecera de un qmd
La cabecera están en formato YAML y contiene los metadatos del documento:

titleysubtitle: el título/subtítulo del documentoauthor: autor del mismodate: fechaformat: formato de salida (podremos personalizar)theme: si tienes algún archivo de estilostoc: si quieres índice o notoc-location: posición del índicetoc-title: título del índicetoc-depth: profundidad del índice
editor: si estás en modo visual o source.
Texto de un qmd
Respecto a la escritura solo hay una cosa importante: salvo que indiquemos lo contrario, TODO lo que vamos a escribir es texto (normal). No código R.
Vamos a empezar escribiendo una sección al inicio (# Intro y detrás por ej. la frase
Este material ha sido diseñado por el profesor Javier Álvarez Liébana, docente en la Universidad Complutense de Madrid
Además al Running Code le añadiremos una almohadilla #: las almohadillas FUERA DE CHUNKS nos servirán para crear epígrafes (secciones) en el documento
Índice de un qmd


Para que el índice capture dichas secciones modificaremos la cabecera del archivo como se observa en la imagen (puedes cambiar la localización del índice y el título si quieres para probar).
Texto en un qmd
Vamos a personalizar un poco el texto haciendo lo siguiente:

Vamos a añadir negrita al nombre (poniendo ** al inicio y al final).
Vamos añadir cursiva a la palabra material (poniendo _ al inicio y al final).
Vamos añadir un enlace https://www.ucm.es, asociándolo al nombre de la Universidad. Para ello el título lo ponemos entre corchetes y justo detrás el enlace entre paréntesis
[«Universidad Complutense de Madrid»](https://www.ucm.es)
Código en un qmd
Para añadir código R debemos crear nuestras cajas de código llamadas chunks: altos en el camino en nuestro texto markdown donde podremos incluir código de casi cualquier lenguaje (y sus salidas).

Para incluir uno deberá de ir encabezado de la siguiente forma tienes un atajo Command + Option + I (Mac) o Ctrl + Shift + I (Windows)
Código en un qmd
Dentro de dicha cajita (que tiene ahora otro color en el documento) escribiremos código R como lo veníamos haciendo hasta ahora en los scripts.
Etiquetando chunks
Los chunks pueden tener un nombre o etiqueta, de forma que podamos referenciarlos de nuevo para no repetir código.
Ejecutando chunks


En cada chunk aparecen dos botones:
botón de play: activa la ejecución y salida de ese chunk particular (lo puedes visualizar dentro de tu propio
RStudio)botón de rebobinar: activa la ejecución y salida de todos los chunk hasta ese (sin llegar a él)
Además podemos incluir código R dentro de la línea de texto (en lugar de mostrar el texto x ejecuta el código R mostrando la variable).
Personalización de chunks
Los chunks podemos personalizarlos con opciones al inicio del chunk precedido de #|:
#| echo: false: ejecuta código y se muestra resultado pero no visualiza código en la salida.#| include: false: ejecuta código pero no muestra resultado y no visualiza código en la salida.#| eval: false: no ejecuta código, no muestra resultado pero sí visualiza código en la salida.#| message: false: ejecuta código pero no muestra mensajes de salida.#| warning: false: ejecuta código pero no muestra mensajes de warning.#| error: true: ejecuta código y permite que haya errores mostrando el mensaje de error en la salida.

Estas opciones podemos aplicarlas chunk a chunk o fijar los parámetros de forma global con knitr::opts_chunk$set() al inicio del documento (dentro de un chunk).
Organizando qmd
Además de texto y código podemos introducir lo siguiente:
Ecuaciones: puedes añadir además ecuaciones como \(x^2\) (he escrito
$x^2$, la ecuación entre dólares).Listas: puedes itemizar elementos poniendo
*
* Paso 1: ...
* Paso 2: ...
- Cross-references: puedes etiquetar partes del documento (la etiqueta se construye con
{#nombre-seccion}) y llamarlas luego con[Sección](@nombre-seccion)
Gráficas/imágenes en qmd


Por último, también podemos añadir pies de gráficas o imágenes añadiendo #| fig-cap: "..."
Fíjate que el caption está en el margen (por ejemplo). Puedes cambiarlo introduciendo ajustes en la cabecera (todo lo relativo a figuras empieza por fig-, y puedes ver las opciones tabulando). Tienes más información en https://quarto.org/

Añadir estilos


Por último puedes añadir un tema personalizado incluyendo un archivo de estilos (archivo en formato .scss o .css). Te he dejado uno en https://github.com/dadosdelaplace/R-ayuntamiento/tree/main/material.
Importante
El archivo de estilos debe estar en la misma carpeta que el archivo .qmd
🐣 Caso práctico
En la carpeta de datos tienes el dataset breast-cancer-wisconsin-data.csv. Crea un archivo .qmd y personalízalo incluyendo lo siguiente:
- Importa el archivo csv a un
tibble. ¿Es tidydata? ¿Cuántos pacientes y variables tenemos?
- El dataset representa datos de cáncer de pecho (
ididentificador,diagnosisel diagnóstico maligno/benigno y el resto propiedades del tumor). Usando SOLO LO APRENDIDO, convierte a tidydata. ¿Qué % tenían un tumor maligno y qué % uno benigno?
- ¿Cuál de los dos tipos de tumores tienen, de media, un radio más elevado?
Clase 5: funciones y visualización de datos
Uso de funciones. Visualización de datos
Clase 5: funciones
¿Qué es una función? ¿Cómo se definen?
Creando funciones
No solo podemos usar funciones predeterminadas que vienen ya cargadas en paquetes, además podemos crear nuestras propias funciones para automatizar tareas.
¿Cómo crear nuestra propia función? Veamos su esquema básico:
- Nombre: por ejemplo
name_fun(sin espacios ni caracteres extraños). Al nombre le asignamos la palabra reservadafunction().
- Definir argumentos de entrada (dentro de
function()).
Creando funciones
No solo podemos usar funciones predeterminadas que vienen ya cargadas en paquetes, además podemos crear nuestras propias funciones para automatizar tareas.
¿Cómo crear nuestra propia función? Veamos su esquema básico:
- Cuerpo de la función dentro de
{ }.
Creando funciones
arg1, arg2, ...: serán los argumentos de entrada, los argumentos que toma la función para ejecutar el código que tiene dentrocódigo: líneas de código que queramos que ejecute la función.return(var_salida): se introducirán los argumentos de salida.
Importante
Todas las variables que definamos dentro de la función son variables locales: solo existirán dentro de la función salvo que especifiquemos lo contrario.
Creando funciones
Veamos un ejemplo muy simple de función para calcular el área de un rectángulo.
Dado que el área de un rectángulo se calcula como el producto de sus lados, necesitaremos precisamente eso, sus lados: esos serán los argumentos de entrada y el valor a devolver será justo su área (\(lado_1 * lado_2\)).
Creando funciones
También podemos hacer una definición directa, sin almacenar variables por el camino.
¿Cómo aplicar la función?
Argumentos por defecto
Imagina ahora que nos damos cuenta que el 90% de las veces usamos dicha función para calcular por defecto el área de un cuadrado (es decir, solo necesitamos un lado). Para ello, podemos definir argumentos por defecto en la función: tomarán dicho valor salvo que le asignemos otro.
¿Por qué no asignar lado_2 = lado_1 por defecto, para ahorrar líneas de código y tiempo?
Argumentos por defecto
Ahora por defecto el segundo lado será igual al primero (si se lo añadimos usará ambos).
Salida múltiple
Compliquemos un poco la función y añadamos en la salida los valores de cada lado, etiquetados como lado_1 y lado_2, empaquetando la salida en una lista.
Introducción a listas
Veamos un pequeño resumen de los datos que ya conocemos:
vectores: colección de elementos de igual tipo. Pueden ser números, caracteres o valores lógicos, entre otros.
matrices: colección BIDIMENSIONAL de elementos de igual tipo e igual longitud.
data.frame / tibble: colección BIDIMENSIONAL de elementos de igual longitud pero de cualquier tipo.
Las listas serán colecciones de variables de diferente tipo y diferente longitud, con estructuras totalmente heterógeneas (incluso una lista puede tener dentro a su vez otra lista).
Introducción a listas
Vamos a crear nuestra primera lista con list() con tres elementos: el nombre de nuestros padres/madres, nuestro lugar de nacimiento y edades de nuestros hermanos.
variable_1 <- c("Paloma", "Gregorio")
variable_2 <- "Madrid"
variable_3 <- c(25, 30, 26)
lista <- list("progenitores" = variable_1, "lugar_nacimiento" = variable_2,
"edades_hermanos" = variable_3)
lista$progenitores
[1] "Paloma" "Gregorio"
$lugar_nacimiento
[1] "Madrid"
$edades_hermanos
[1] 25 30 26Introducción a listas
Si observas el objeto que hemos definido como lista, su longitud del es de 3 ya que tenemos guardados tres elementos: un vector de caracteres (de longitud 2), un caracter (vector de longitud 1), y un vector de números (de longitud 3)
Introducción a listas
Si los juntásemos con un tibble(), al tener distinta longitud, obtendríamos un error.
Introducción a listas
- Acceder por índice: con el operador
[[i]]accedemos al elemento i-ésimo de la lista.
- Acceder por nombre: con el operador
$nombre_elementoaccedemos por su nombre.
Salida múltiple
Antes nos daba igual el orden de los argumentos pero ahora el orden de los argumentos de entrada importa, ya que en la salida incluimos lado_1 y lado_2.
Funciones: generando conocimiento
Parece una tontería lo que hemos hecho pero hemos cruzado una frontera importante: hemos pasado de consumir conocimiento (código de otros paquetes, elaborado por otros/as), a generar conocimiento, creando nuestras propias funciones.
Variables locales vs globales
Un aspecto importante sobre el que reflexionar con las funciones: ¿qué sucede si nombramos a una variable dentro de una función a la que se nos ha olvidado asignar un valor dentro de la misma?
Debemos ser cautos al usar funciones en R, ya que debido a la «regla lexicográfica», si una variable no se define dentro de la función, R buscará dicha variable en el entorno de variables.
Variables locales vs globales
Si una variable ya está definida fuera de la función (entorno global), y además es usada dentro de cambiando su valor, el valor solo cambia dentro pero no en el entorno global.
Variables locales vs globales
Si queremos que además de cambiar localmente lo haga globalmente deberemos usar la doble asignación (<<-).
💻 Tu turno
Intenta realizar los siguientes ejercicios sin mirar las soluciones
📝 Modifica el código inferior para definir una función llamada funcion_suma, de forma que dados dos elementos, devuelve su suma.
📝 Modifica el código inferior para definir una función llamada funcion_producto, de forma que dados dos elementos, devuelve su producto, pero que por defecto calcule el cuadrado
📝 Define una función llamada igualdad_nombres que, dados dos nombres, nos diga si son iguales o no. Hazlo considerando importantes las mayúsculas, y sin que importen las mayúsculas. Echa un vistazo al paquete {stringr}.
Código
# Distinguiendo mayúsculas
igualdad_nombres <- function(persona_1, persona_2) {
return(persona_1 == persona_2)
}
igualdad_nombres("Javi", "javi")
igualdad_nombres("Javi", "Lucía")
# Sin importar mayúsculas
igualdad_nombres <- function(persona_1, persona_2) {
return(toupper(persona_1) == toupper(persona_2))
}
igualdad_nombres("Javi", "javi")
igualdad_nombres("Javi", "Lucía")📝 Crea una función llamada calculo_IMC que, dados dos argumentos (peso y estatura en metros) y un nombre, devuelva una lista con el IMC y el nombre.
📝 Repite el ejercicio anterior pero con otro argumento opcional que se llame unidades (por defecto, unidades = "metros"). Desarrolla la función de forma que haga lo correcto si unidades = "metros" y si unidades = "centímetros".
📝 Crea un tibble ficticio de 7 personas, con tres variables (inventa nombre, y simula peso, estatura en centímetros), y aplica la función definida de forma que obtengamos una cuarta columna con su IMC.
🐣 Caso práctico: funciones
Define una función llamada conversor_temperatura que, dada una temperatura en Fahrenheit, Celsius o Kelvin, la convierta a cualquiera de las otras (piensa que argumentos necesita el usuario). Aplica la función a la columna Temp del conjunto airquality, e incorpórala al fichero en una nueva columna Temp_Celsius.
🐣 Caso práctico 5
Acude a https://cnecovid.isciii.es/covid19/#documentaci%C3%B3n-y-datos e importa casos_hosp_uci_def_sexo_edad_provres.csv. Documentación en https://cnecovid.isciii.es/covid19/resources/metadata_casos_hosp_uci_def_sexo_edad_provres.pdf
- ¿Cuántos casos hay notificados para edad desconocida? ¿Y desagregado por sexo? Recodifica adecuadamente las provincias
- Dado su escaso peso en el total, genera una nueva base de datos en la que borremos aquellos registros con franja de edad desconocida.
- Con la base de datos generada en el ejercicio anterior, calcula la proporción de casos con sexo desconocido. Haz lo mismo con provincia desconocida. Elimina dichos registros si el número de casos representa menos del 1% (para cada una).
🐣 Caso práctico 5
- Del dataset anterior, elimina la variables de hospitalizados y UCI. Tras ello renombra las columnas de casos y fallecidos por
casos_diariosyfallec_diarios, respectivamente. Tras ello crea dos nuevas variables llamadascasos_acumy otrafallec_acum, que contengan los casos acumulados y fallecidos acumulados para cada fecha, desagregados por provincia, tramo etario y sexo.
- ¿Cuáles fueron las 7 provincias con más casos a lo largo de toda la pandemia? ¿Y las 5 provincias con menos fallecidos? ¿Y si lo desagregamos por sexo?
🐣 Caso práctico 5
- Define una función llamada
calculo_letalidad()que, dados como argumentos un vector ordenado (por fecha) de casos y otro de fallecidos, devuelva el % de casos que han fallecido, de manera acumulada en cada fecha. Haz uso de dicha función y crea una nueva variable que represente la letalidad, en cada grupo de edad, sexo y provincia.
Tras ello, determina las 5 provincias con mayor letalidad en mujeres mayores de 80 años a fecha 01 de marzo de 2022.
Clase 5: relacionando datos
Cruzxando tablas
Relacionando datos
Al trabajar con datos no siempre tendremos la información en una sola tabla y a veces nos interesará cruzar la información de distintas fuentes.
Para ello usaremos un clásico de todo lenguaje que maneja datos: los famosos join, una herramienta que nos va a permitir cruzar una o variables tablas, haciendo uso de una columna identificadora de cada una de ellas (por ejemplo, imagina que cruzamos datos de hacienda y de antecedentes penales, haciendo join por la columna DNI).
Relacionando datos
inner_join(): solo sobreviven los registros con id en ambas tablas.full_join(): mantiene todos los registros de ambas tablas.left_join(): mantiene todos los registros de la primera tabla, y busca cuales tienen id también en la segunda (en caso de no tenerlo se rellena con NA los campos de la 2ª tabla).right_join(): mantiene todos los registros de la segunda tabla, y busca cuales tienen id también en la primera.
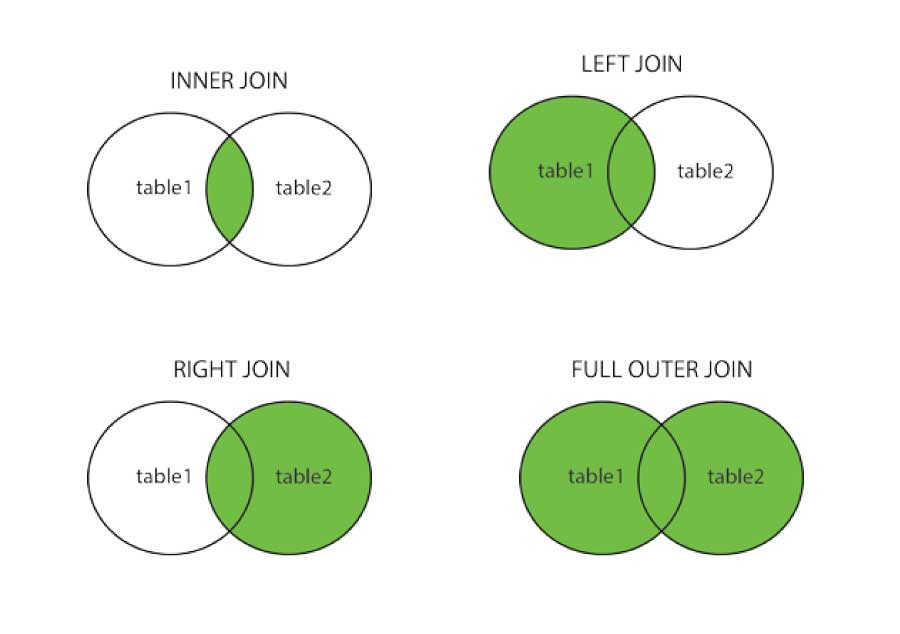
Relacionando datos
Vamos a probar los distintos joins con un ejemplo sencillo
Left join
Imagina que queremos incorporar a tb_1 la información de la tabla_2, identificando los registros por la columna key (indicando con by = "key" la columna por la que tiene que cruzar): queremos mantener todos los registros de la primera tabla y buscar cuales tienen id (mismo valor en key) también en la segunda tabla.
Left join
# A tibble: 3 × 3
key val_x val_y
<dbl> <chr> <chr>
1 1 x1 y1
2 2 x2 y2
3 3 x3 <NA> Fíjate que los registros de la primera cuya key no ha encontrado en la segunda les ha dado el valor de ausente.
Right join
El right_join() realizará la operación contraria: vamos ahora a incorporar a tb_2 la información de la tabla_2, identificando los registros por la columna key (indicando con by = "key" la columna por la que tiene que cruzar): queremos mantener todos los registros de la segunda y buscar cuales tienen id (mismo valor en key) también en la primera tabla.
Right join
# A tibble: 3 × 3
key val_x val_y
<dbl> <chr> <chr>
1 1 x1 y1
2 2 x2 y2
3 4 <NA> y3 Fíjate que ahora los registros de la segunda cuya key no ha encontrado en la primera son los que les ha dado el valor de ausente.
Claves y sufijos
Las columnas clave que usaremos para el cruce no siempre se llamarán igual.
by = c("key_2" = "key_2"): le indicaremos en qué columna de cada tabla están las claves por las que vamos a cruzar.
Claves y sufijos
Además podemos cruzar por varias columnas a la vez (interpretará como igual registro aquel que tenga el conjunto de claves igual), con by = c("var1_t1" = "var1_t2", "var2_t1" = "var2_t2", ...). Modifiquemos el ejemplo anterior
Claves y sufijos
También podría suceder que al cruzar dos tablas, haya columnas de valores que se llamen igual
Claves y sufijos
Dicho sufijo podemos especificárselo en el argumento opcional suffix = ..., que nos permita distinguir las variables de una tabla y de otra.
Full join
Los dos anteriores casos forman lo que se conoce como outer joins: cruces donde se mantienen observaciones que salgan en al menos una tabla. El tercer outer join es el conocido como full_join() que nos mantendrá las observaciones de ambas tablas, añadiendo las filas que no casen con la otra tabla.
Inner join
Frente a los outer join está lo que se conoce como inner join, con inner_join(): un cruce en el que solo se mantienen las observaciones que salgan en ambas tablas, solo mantiene aquellos registros matcheados.
Inner join
Fíjate que en términos de registros, inner_join si es conmutativa, nos da igual el orden de las tablas: lo único que cambia es el orden de las columnas que añade.
Anti/semi join
Por último tenemos dos herramientas interesantes para filtrar (no cruzar) registros: semi_join() y anti_join(). El semi join nos deja en la primera tabla los registros que cuya clave está también en la segunda (como un inner join pero sin añadir la info de la segunda tabla). Y el segundo, los anti join, hace justo lo contrario (aquellos que no están).
💻 Tu turno
Intenta realizar los siguientes ejercicios sin mirar las soluciones
Para los ejercicios usaremos las tablas disponibles en el paquete {nycflights13} (echa un vistazo antes)
- airlines: nombre de aerolíneas (con su abreviatura).
- airports: datos de aeropuertos (nombres, longitud, latitud, altitud, etc).
- flights: datos de vuelos.
- planes: datos de los aviones.
- weather: datos meteorológicos horarios de las estaciones LGA, JFK y EWR.
💻 Tu turno
Intenta realizar los siguientes ejercicios sin mirar las soluciones
📝 Del paquete {nycflights13} cruza la tabla flights con airlines. Queremos mantener todos los registros de vuelos, añadiendo la información de las aerolíneas a la tabla de aviones.
📝 A la tabla obtenida del cruce del apartado anterior, cruza después con los datos de los aviones en planes, pero incluyendo solo aquellos vuelos de los que tengamos información de sus aviones (y viceversa).
📝 Repite el ejercicio anterior pero conservando ambas variables year (en una es el año del vuelo, en la otra es el año de construcción del avión), y distinguiéndolas entre sí
📝 Al cruce obtenido del ejercicio anterior incluye la longitud y latitud de los aeropuertos en airports, distinguiendo entre la latitud/longitud del aeropuerto en destino y en origen.
📝 Filtra de airports solo aquellos aeropuertos de los que salgan vuelos. Repite el proceso filtrado solo aquellos a los que lleguen vuelos
Clase 5: inicio a la visualización
Visualización de datos
Dataviz: historia
La aparición de gráficos estadísticos es relativamente reciente en la ciencia ya que hasta la Edad Media la única visualización estaba en los mapas. 1 Las propias palabras chart y cartography derivan del mismo origen latino, charta, aunque el primer uso de coordenadas viene de los egipcios. 2 3
No es hasta la Edad Media, cuando la navegación y la astronomía empezaban a tomar relevancia, cuando aparece la primera gráfica (no propiamente estadística), del movimiento cíclico de los planetas (siglos X y XI)
Navegación y astronomía
Con una motivación similar, en torno a 1360 el matemático Nicole Oresme diseñó el primer gráfico de barras (pero no estadístico), con la idea de visualizar a la vez dos magnitudes físicas teóricas. 1

Primer gráfico estadístico
La mayoría de expertos, como Tufte 1 2, consideran este gráfico casi longitudinal como la primera visualización de datos de la historia, hecha por Van Langren en 1644, representando la distancia entre Toledo y Roma.


¿Qué es una dataviz?
¿Es una gráfica estadística? ¿Por qué sí o por qué no?

No hay ninguna INFORMACIÓN representada
¿Qué es una dataviz?
¿Es una gráfica estadística? ¿Por qué sí o por qué no?
No hay ningún PROCESO DE MEDIDA representado, no cuantifica nada (real).
¿Qué es una dataviz?
¿Es una gráfica estadística? ¿Por qué sí o por qué no?
No hay ningún DATO representado en él, es una magnitud física teórica, no un dato (medido empíricamente o simulado).
¿Qué es una dataviz?
Esas mismas preguntas se hizo Joaquín Sevilla 1, proporcionando 3 requisitos:
- Que se base en el esquema de composición de eje métrico (proceso de medida): debe medir algo.
- Debe incluir información estadística (datos)
- La relación de representatividad debe ser reversible: los datos deberían poder «recuperarse» a partir de la gráfica .
Abolición de tartas
Hay muchas formas de hacer una gráfica estadística, y no suele pasar por un gráfico de tartas ya que tienen un grave problema de reversibilidad:
Si hay muchas variables: salvo que tengas transportador de ángulos…
Si hay pocas variables: ¿aporta algo distinto (y/o mejor) que una tabla?
Abolición de tartas
El principal problema de un diagrama de sectores es que la posible información está contenida en los ángulos, pero nuestra interpretación la realizamos a través de la comparación de áreas (nuestros ojos no miden bien ángulos), las cuales dependen no solo del ángulo sino del radio.
Algo similar sucede con los mal llamados gráficos tridimensionales (son bidimensionales con perspectiva en realidad): los valores más cercanos aparecen sobredimensionados, siendo prácticamente imposible la reversibilidad por la distorsión.
Vizfails

La figura elegida (persona caminando) sin relación con lo visualizado: mala metáfora.
Los sectores señalados sin relación con el ítem a representar, lo que dificulta su interpretación.
Los colores sin codificar: no dan información de ningún tipo.
Las formas irregulares impiden la comparación de las áreas (amén de que la suma total supera el 100%).
Sin fuente
Vizfails
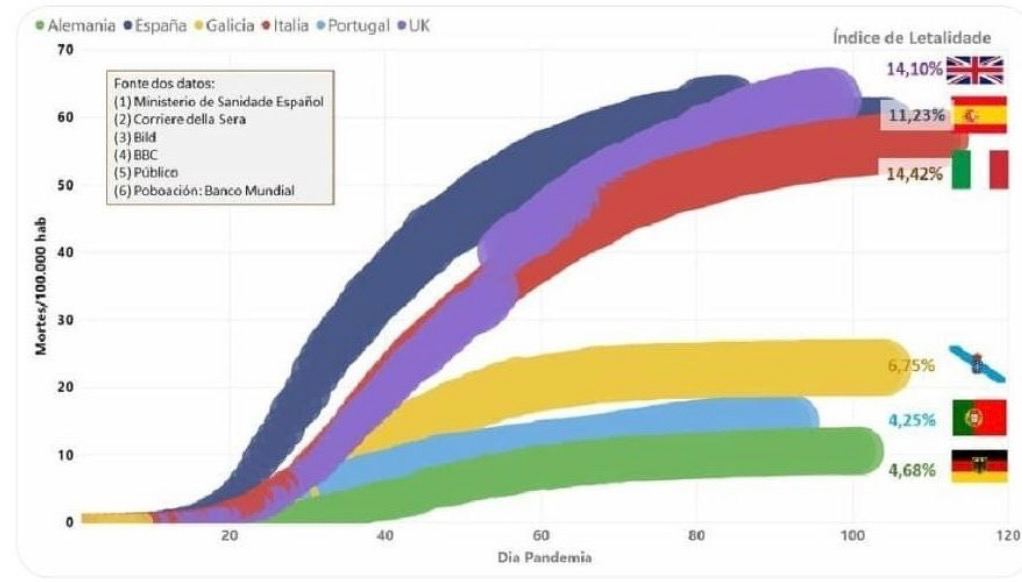
Vizfails
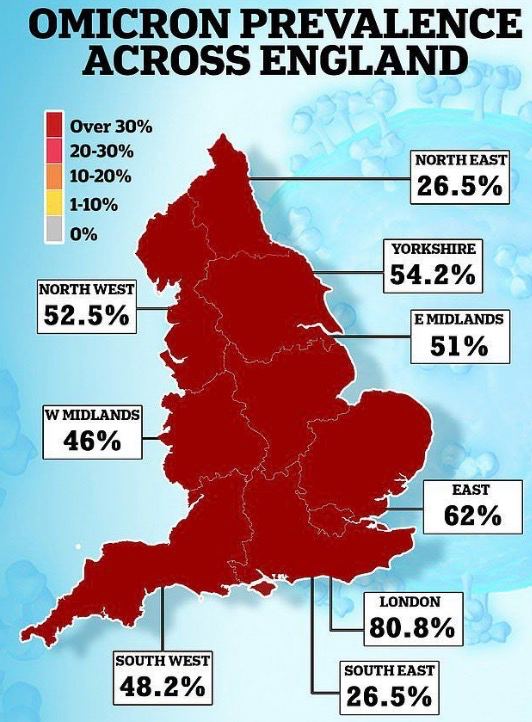
La importancia del CONTEXTO
Una buena idea puede estar mal ejecutada: la forma de llevarla a cabo es importante

Dataviz: historia
En el siglo XVII hubo un boom de la estadística al empezar a aplicarse en demografía. Uno de los autores más importantes fue J. Graunt, autor de «Natural and Political Observations Made upon the Bills of Mortality» (1662), estimando la población de Londres con las primeras tablas de natalidad y mortalidad.
Son precisamente las tablas de Graunt las que usó Christiaan Huygens para generar la primera gráfica de densidad de una distribución continua (esperanza de vida vs edad).

Gráficos de Playfair
La figura que cambió el dataviz fue, sin lugar a dudas, el economista y político William Playfair (1759-1823), publicando en 1786 el «Atlas político y comercial» 1 2 con 44 gráficas (43 series temporales y el diagrama de barras más famoso de la historia).
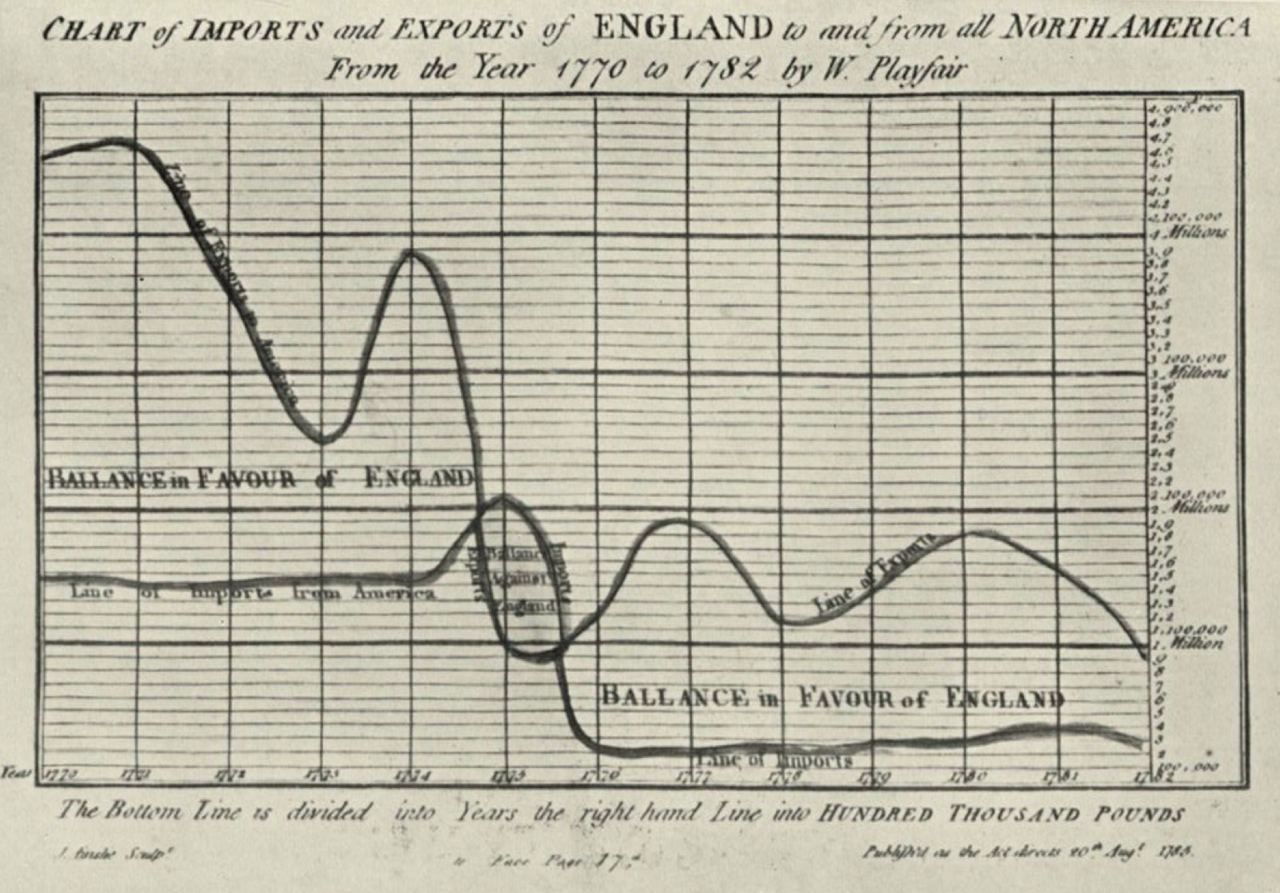
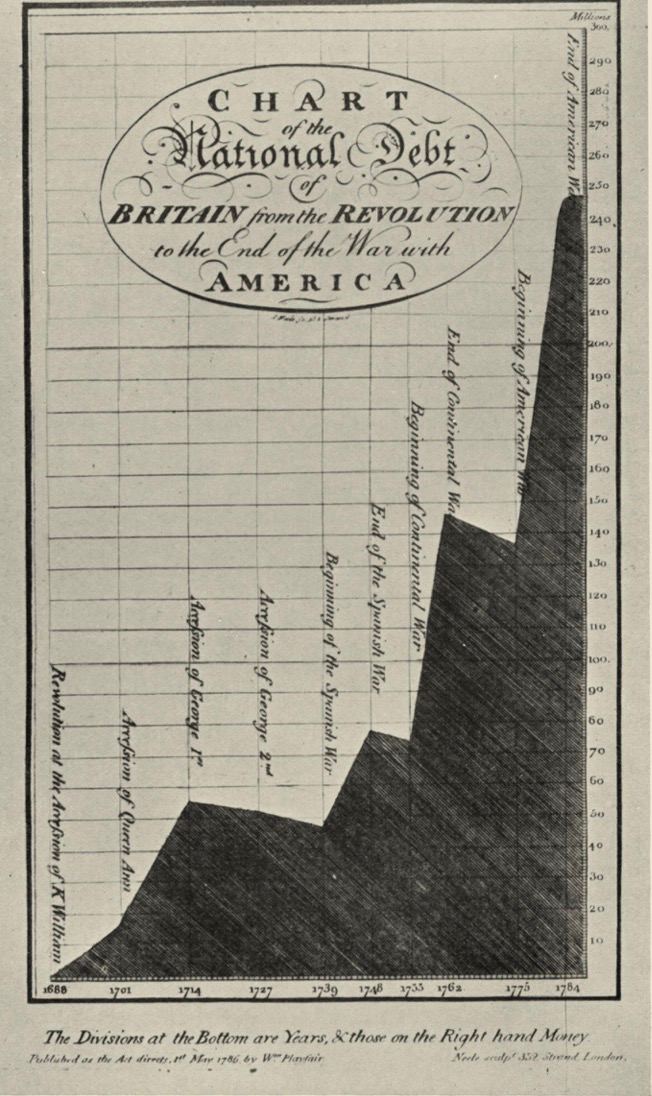
Gráficos de Playfair
Playfair no solo fue el primero en usar el dataviz para entender (y no solo describir): fue el primero en usar conceptos modernos como grid, tema o color
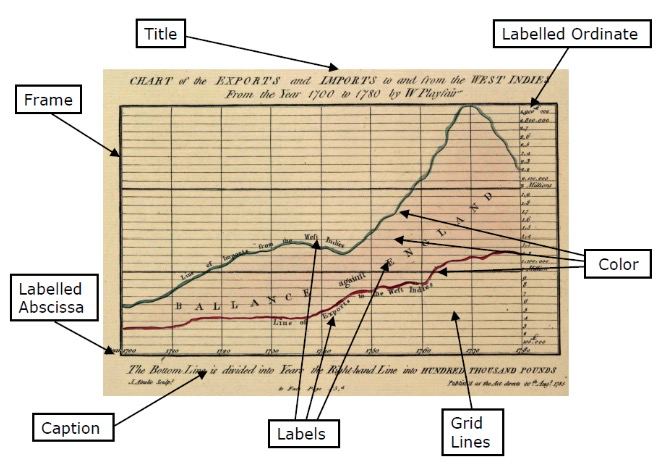
Gráficos de Playfair
Playfair es además el autor del gráfico de barras más famoso (no fue el primero pero sí quien lo hizo mainstream).
Gráficos de Playfair
Playfair además fue el primero en combinar gráficos en la misma visualización 1 2
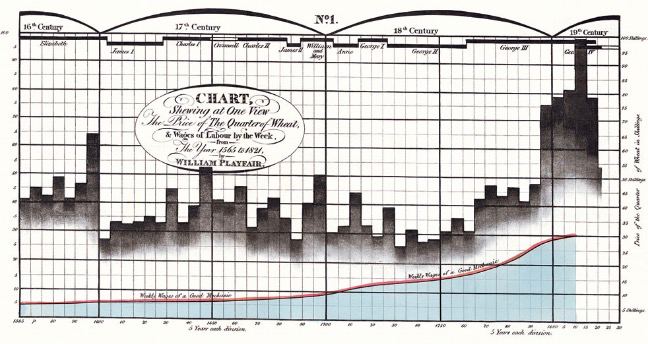

Mapas de Minard
Otro pionero en combinar visualizaciones fue Minard, autor del famoso «Carte figurative des pertes successives en hommes de l’Armée Française dans la campagne de Russie 1812-1813», según Tufte «el mejor gráfico estadístico jamás dibujado», publicado en 1869 sobre la desastrosa campaña rusa de Napoleón en 1812 (3 variables en un gráfico bidimensional)

Extraída de https://friendly.github.io/HistDataVis.
Primer scatter plot
Según J. Sevilla, se considera al astrónomo británico John Frederick William Herschel el autor del primer diagrama de dispersión o scatterplot en 1833, visualizando el movimiento de la estrella doble Virginis (tiempo en el eje horizontal, posición angular en el eje vertical)
Extraído de https://friendly.github.io/HistDataVis.
Primera pirámide poblacional
La primera pirámide de población (doble histograma de población), fue publicada por Francis Amasa Walker, superintendente del censo de EE.UU., en 1874.

Extraída de https://www.depauw.edu/learn/dew/wpaper/workingpapers/DePauw2016-02-Barreto-DemographyEconomics.pdf
Florence Nigthingale
El 21 de octubre de 1854 Florence Nigthingale fue enviada para mejorar las condiciones sanitarias de los soldados británicos en la guerra de Crimea.
A su regreso demostró que los soldados fallecían por las condiciones sanitarias. Nigthingale es la creadora del famoso diagrama de rosa, visualizando tres variables a la vez y su estacionalidad.
El 8 de febrero de 1955, The Times la describió como la «ángel guardián» de los hospitales, y acabó siendo conocida como «The Lady with the Lamp» tras un poema de H. W. Longfellow (1857).
Años después se convirtió en la primera mujer en la Royal Statistical Society.

Diagrama de rosa
Florence Nigthingale es la creadora del famoso diagrama de rosa, permitiendo pintar tres variables a la vez y su estacionalidad: tiempo (cada gajo es un mes), nº de muertes (área del gajo) y causa de la muerte (color del gajo: azules enfermedades infecciosas, rojas por heridas, negras otras causas).
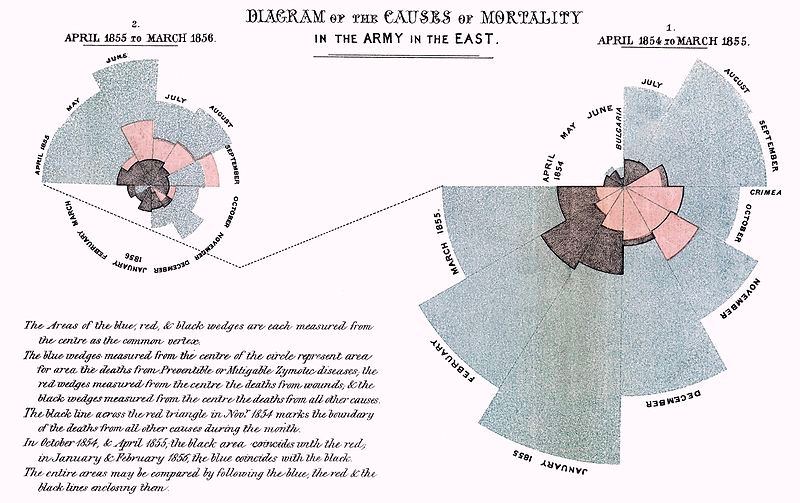
Recursos de dataviz
📚 «The Functional Art: an introduction to information graphics and visualization» de Alberto Cairo
📚 «Gramática de las gráficas: pistas para mejorar las representaciones de datos» de Joaquín Sevilla
📚 «A Brief History of Visualization» de Friendly et al. (2008)
📚 «The Grammar of Graphics» de Leland Wilkinson
📚 «The Minard System: The Graphical Works of Charles-Joseph Minard» de Sandra Rendgen
📚 «The Visual Display of Quantitative Information» de E. W. Tufte
Dataviz en R: ggplot2
El paquete {ggplot2} se basa en la idea de Wilkinson en «Grammar of graphics»: dotar a los gráficos de una gramática propia. Una de las principales fortalezas de R es la visualización con {ggplot2}.
La visualización de datos debería ser una parte fundamental de todo análisis de datos. No es solo una cuestión estética.

Dataviz en R: ggplot2
La filosofía detrás de {ggplot2} es entender los gráficos como parte del flujo de trabajo, dotándoles de una gramática. El objetivo es empezar con un lienzo en blanco e ir añadiendo capas a tu gráfico. La ventaja de {ggplot2} es poder mapear atributos estéticos (color, forma, tamaño) de objetos geométricos (puntos, barras, líneas) en función de los datos.
La documentación del paquete puedes consultarla en https://ggplot2-book.org/introduction.html

Dataviz en R: ggplot2
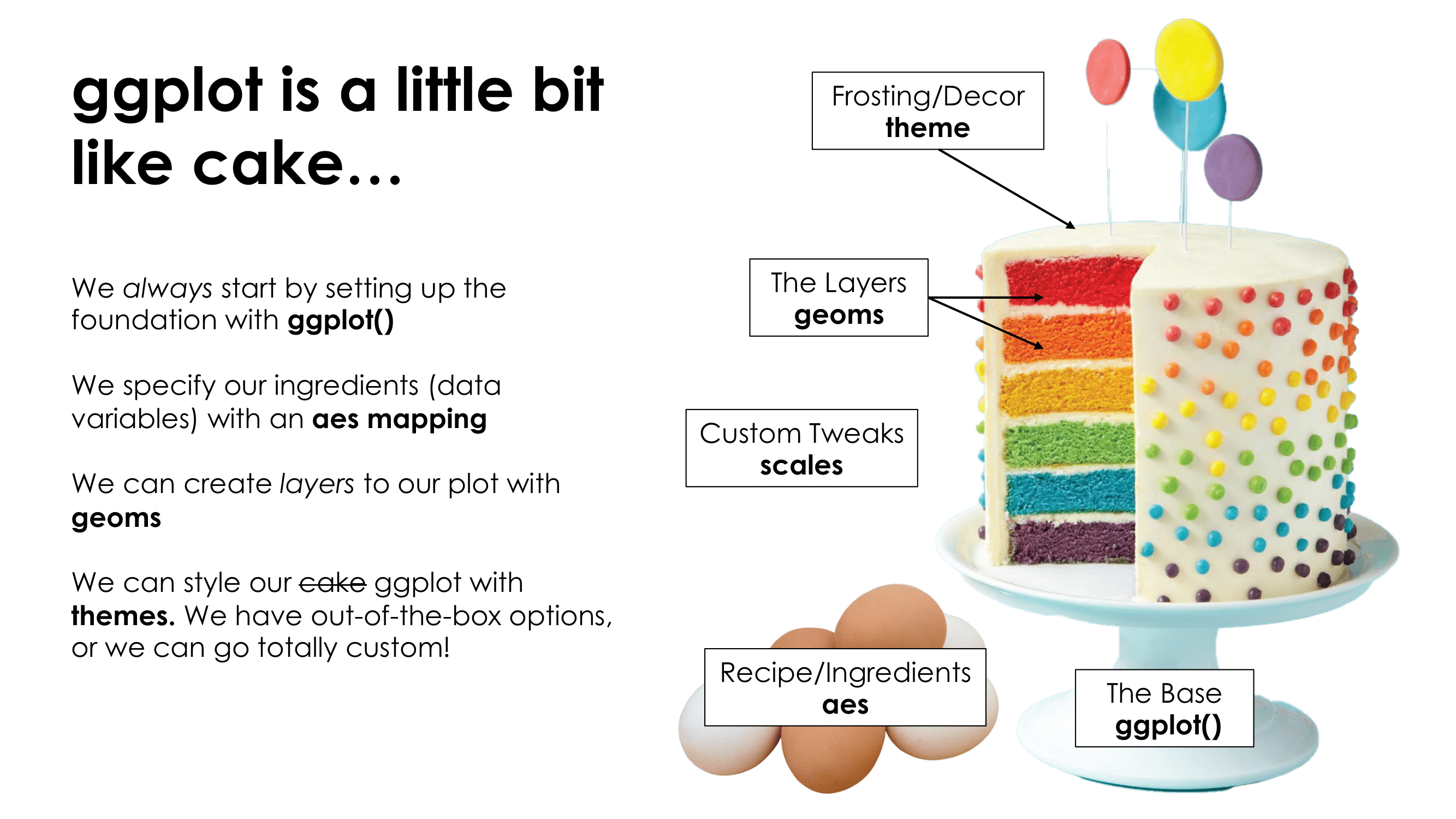
Dataviz en R: ggplot2
Un gráfico se podrá componer de capas
- Datos (data)
- Mapeado (aesthetics) de elementos estéticos: ejes, color, forma, etc (en función de los datos)
- Geometría (geom): puntos, líneas, barras, polígonos, etc.
- Componer gráficas (facet)
- Transformaciones (stat): ordenar, resumir, etc.
- Coordenadas (coord): coordenadas cartesianas, polares, grids, etc.
- Temas (theme): fuente, tamaño de letra, subtítulos, captions, leyenda, ejes, etc.
Primer intento: scatter plot
Veamos un primer intento para entender la filosofía ggplot. Imagina que queremos dibujar un scatter plot (diagrama de dispersión de puntos). Para ello vamos a usar el conjunto de datos gapminder, del paquete homónimo: un fichero con datos de esperanzas de vida, poblaciones y renta per cápita de distintos países en distintos momentos temporales.
# A tibble: 1,704 × 6
country continent year lifeExp pop gdpPercap
<fct> <fct> <int> <dbl> <int> <dbl>
1 Afghanistan Asia 1952 28.8 8425333 779.
2 Afghanistan Asia 1957 30.3 9240934 821.
3 Afghanistan Asia 1962 32.0 10267083 853.
4 Afghanistan Asia 1967 34.0 11537966 836.
5 Afghanistan Asia 1972 36.1 13079460 740.
6 Afghanistan Asia 1977 38.4 14880372 786.
7 Afghanistan Asia 1982 39.9 12881816 978.
8 Afghanistan Asia 1987 40.8 13867957 852.
9 Afghanistan Asia 1992 41.7 16317921 649.
10 Afghanistan Asia 1997 41.8 22227415 635.
# ℹ 1,694 more rowsPrimer intento: scatter plot
El fichero consta de 1704 registros y 6 variables: country, continent, year, lifeExp (esperanza de vida), pop (población) y gdpPercap (renta per cápita).
Rows: 1,704
Columns: 6
$ country <fct> "Afghanistan", "Afghanistan", "Afghanistan", "Afghanistan", …
$ continent <fct> Asia, Asia, Asia, Asia, Asia, Asia, Asia, Asia, Asia, Asia, …
$ year <int> 1952, 1957, 1962, 1967, 1972, 1977, 1982, 1987, 1992, 1997, …
$ lifeExp <dbl> 28.801, 30.332, 31.997, 34.020, 36.088, 38.438, 39.854, 40.8…
$ pop <int> 8425333, 9240934, 10267083, 11537966, 13079460, 14880372, 12…
$ gdpPercap <dbl> 779.4453, 820.8530, 853.1007, 836.1971, 739.9811, 786.1134, …Para empezar con algo sencillo filtraremos solo los datos de 1997
Ingredientes: (x, y)
¿Qué elementos necesitamos para realizar un diagrama de puntos? Para iniciar el lienzo necesitamos una base de datos y dos variables a representar.
Primera geometría: geom_point()
Rol de os ejes: (x, y)
Rol de os ejes: (x, y)
Color, size, shape: fijos
Dentro de geom_point() tenemos varios argumentos a usar:
na.rm = ...: si queremos que nos quite ausentes.color = ...: color (si tiene dimensión, color del contorno)fill = ...: color del relleno.
Color, size, shape: fijos
Color, size, shape: fijos
Color, size, shape: fijos
Color, size, shape: fijos
Color, size, shape: fijos
Los colores también podemos asignárselos por su código hexadecimal, consultando en https://htmlcolorcodes.com/es/, eligiendo el color que queramos. El código hexadecimal siempre comenzará con #
Mapeado estético: aes()
Hasta ahora los atributos estéticos se los hemos pasado fijos y constantes. Pero la verdadera potencia y versatilidad de ggplot es que podemos mapear los atributos estéticos en función de los datos en aes() para que dependan de variables de los datos.
Mapeado estético: aes()
Podemos combinarlo con lo que hemos hecho anteriormente:
color en función del continente.
tamaño en función de la población.
transparencia la fijamos constante del 50%.
A este scatter plot particular se le conoce BUBBLE CHART
Visualización multivariante
Reflexionemos sobre el gráfico anterior:
- color en función del continente.
- tamaño en función de la población
- transparencia fija del 50%
Usando los datos hemos conseguido dibujar en un gráfico bidimensional 4 variables: lifeExp y gdpPercap en los ejes , continent como color y pop como tamaño de la geometría, con muy pocas líneas de código.
Etiquetas sencillas: labs()
Podemos personalizar de manera sencilla haciendo uso de la capa labs():
title, subtitle: título/subtítulocaption: pie de gráficax, y: nombres de los ejessize, color, fill, ...: nombre en la leyenda de las variables que codifiquen los distintos atributos
ggplot(gapminder_1997,
aes(y = gdpPercap, x = lifeExp, color = continent, size = pop)) +
geom_point(alpha = 0.7) +
labs(x = "Esperanza de vida", y = "Renta per cápita",
title = "Primer ggplot", subtitle = "Datos de gapminder",
caption = "J. Álvarez Liébana", color = "continente", size = "población")Eliminar de la leyenda
Podemos eliminar variables de la leyenda con guides(atributo = "none")
Escalas (scale): ejes
Una de las capas más importantes es la capa de escalas: dentro de aes() solo le indicamos que variable mapeamos pero no sus ajustes.
Vamos a configurar el eje x para tener marcas cada 10 unidades (scale_x_continuous())
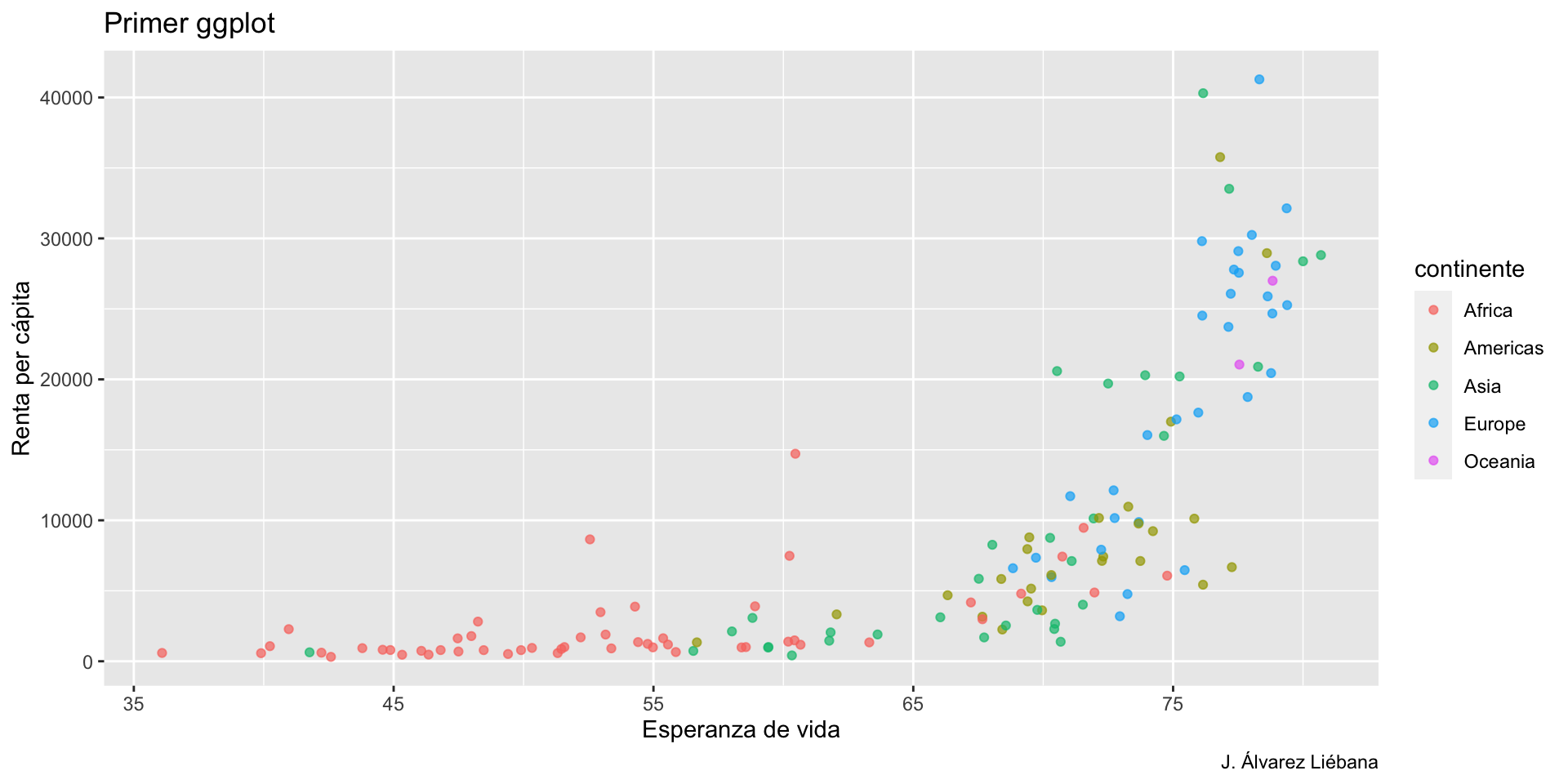
Escalas (scale): colores
La misma idea la podemos aplicar a otro atríbuto como los colores con scale_color_...() y scale_fill_...(): hemos indicado que mapeé dicho atributo por continente pero…¿qué colores usar?
Con scale_color_manual() podemos indicar manualmente una paleta (puedes buscar en https://htmlcolorcodes.com/)
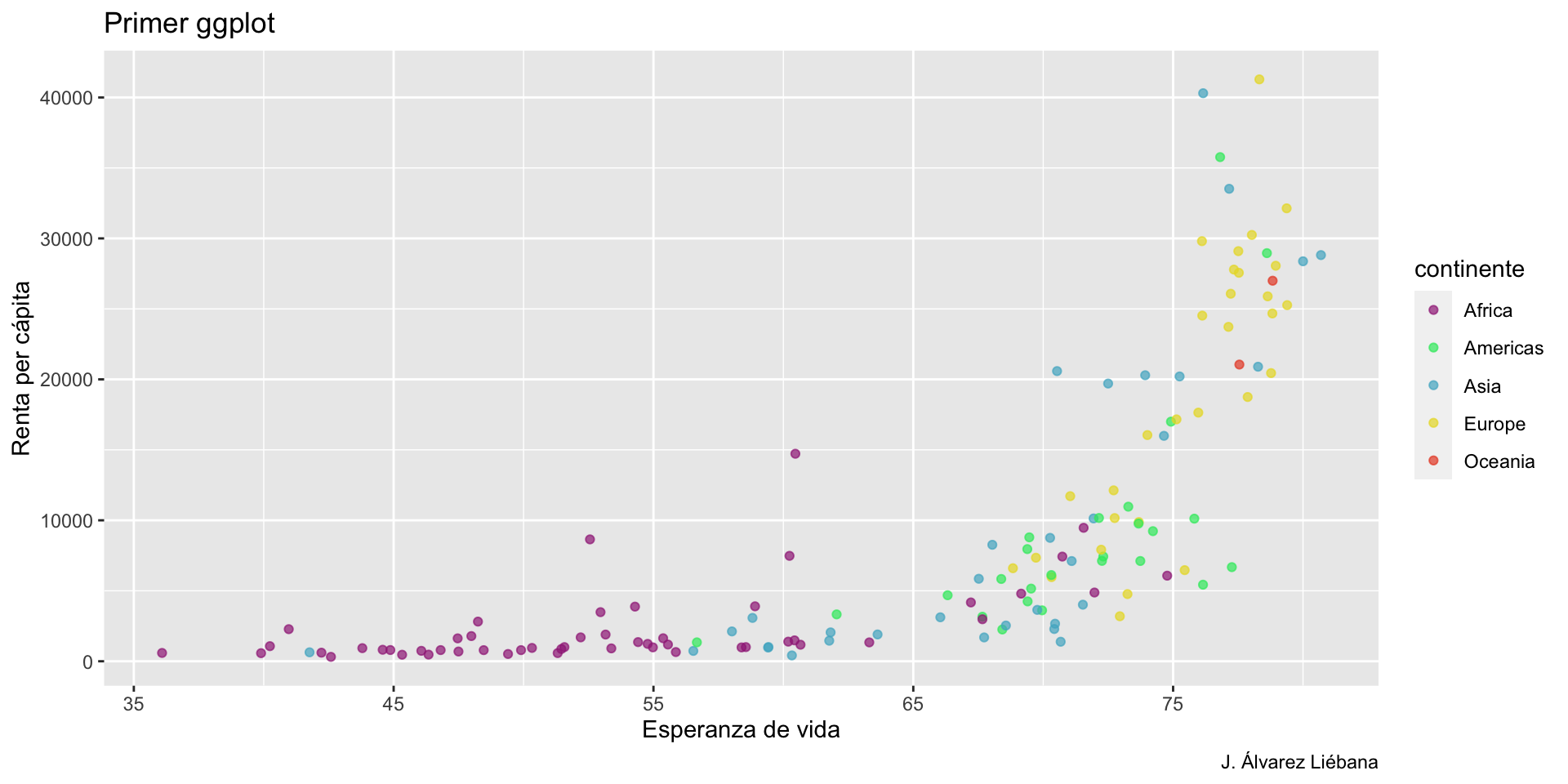
pal <- c("#A02B85", "#2DE86B", "#4FB2CA", "#E8DA2D", "#E84C2D")
ggplot(gapminder_1997, aes(y = gdpPercap, x = lifeExp, color = continent)) +
geom_point(alpha = 0.7) +
scale_x_continuous(breaks = seq(35, 85, by = 10)) +
scale_color_manual(values = pal) +
labs(x = "Esperanza de vida", y = "Renta per cápita", title = "Primer ggplot",
caption = "J. Álvarez Liébana", color = "continente")Escalas (scale): colores
Otra opción es elegir alguna de las paletas de colores diseñadas en el paquete {ggthemes}:
scale_color_economist(): paleta de colores basada en los colores de The Economist.
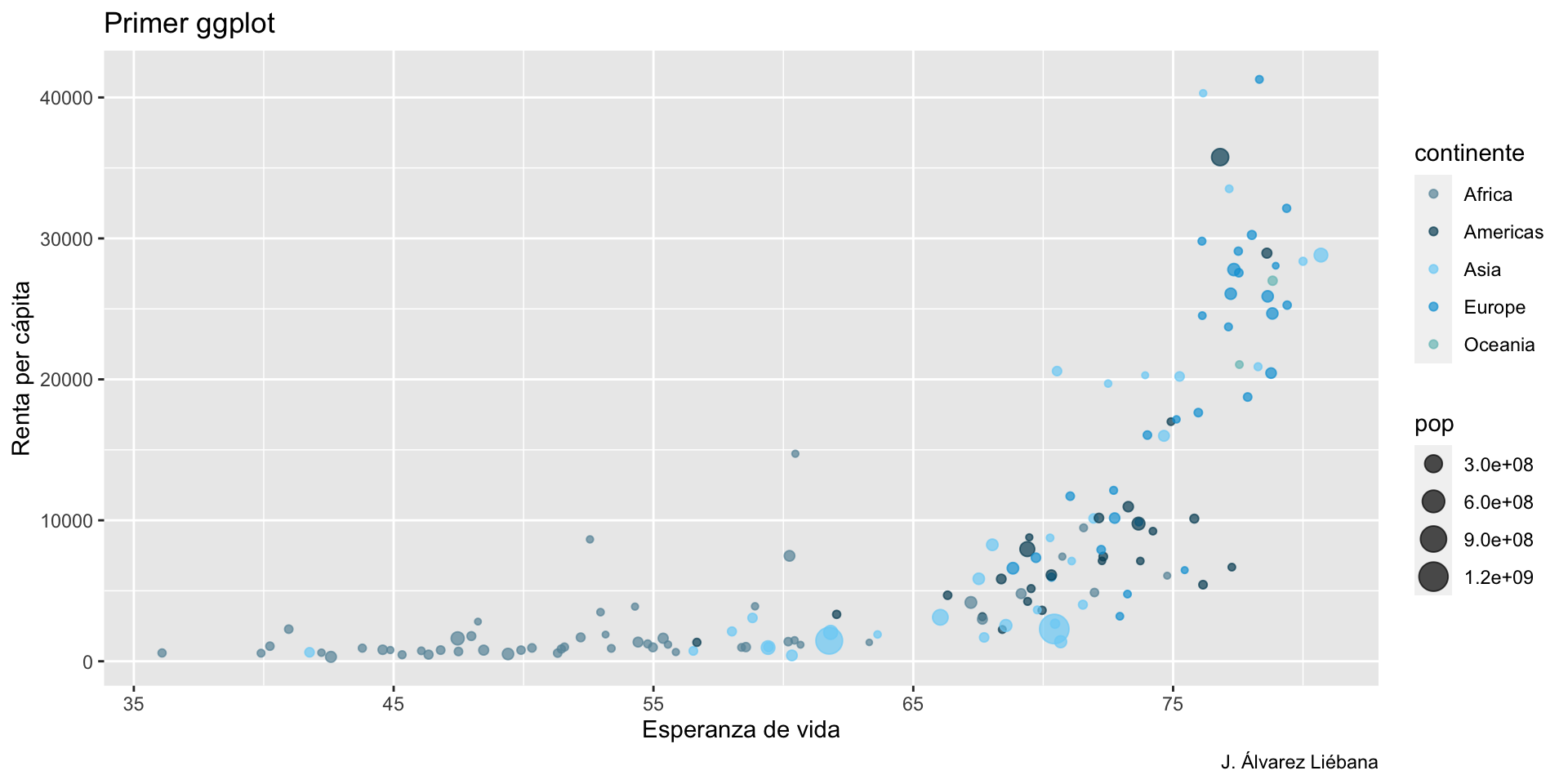
ggplot(gapminder_1997, aes(y = gdpPercap, x = lifeExp, color = continent, size = pop)) +
geom_point(alpha = 0.7) +
scale_x_continuous(breaks = seq(35, 85, by = 10)) +
ggthemes::scale_color_economist() +
labs(x = "Esperanza de vida", y = "Renta per cápita", title = "Primer ggplot",
caption = "J. Álvarez Liébana", color = "continente")Escalas (scale): colores
Otra opción es elegir alguna de las paletas de colores diseñadas en el paquete {ggthemes}:
scale_color_colorblind(): paleta de colores basada en los colores de daltónicos/as.
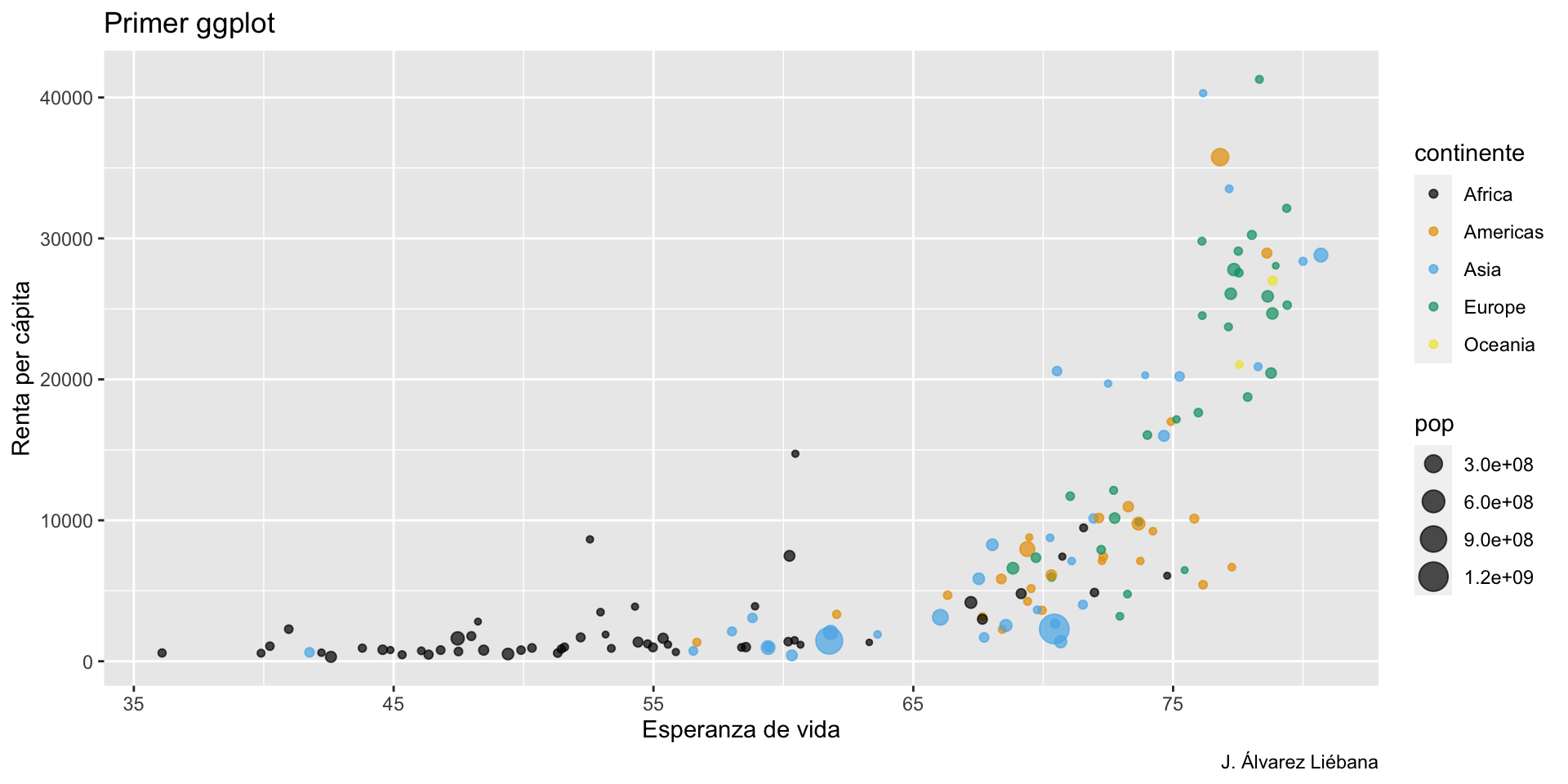
ggplot(gapminder_1997, aes(y = gdpPercap, x = lifeExp, color = continent, size = pop)) +
geom_point(alpha = 0.7) +
scale_x_continuous(breaks = seq(35, 85, by = 10)) +
ggthemes::scale_color_colorblind() +
labs(x = "Esperanza de vida", y = "Renta per cápita", title = "Primer ggplot",
caption = "J. Álvarez Liébana", color = "continente")Escalas (scale): colores
Incluso cargar paletas de colores diseñadas en base a películas o arte
- películas: paquete
{harrypotter}(repositorio de Githubaljrico/harrypotter) usandoscale_color_hp_d().

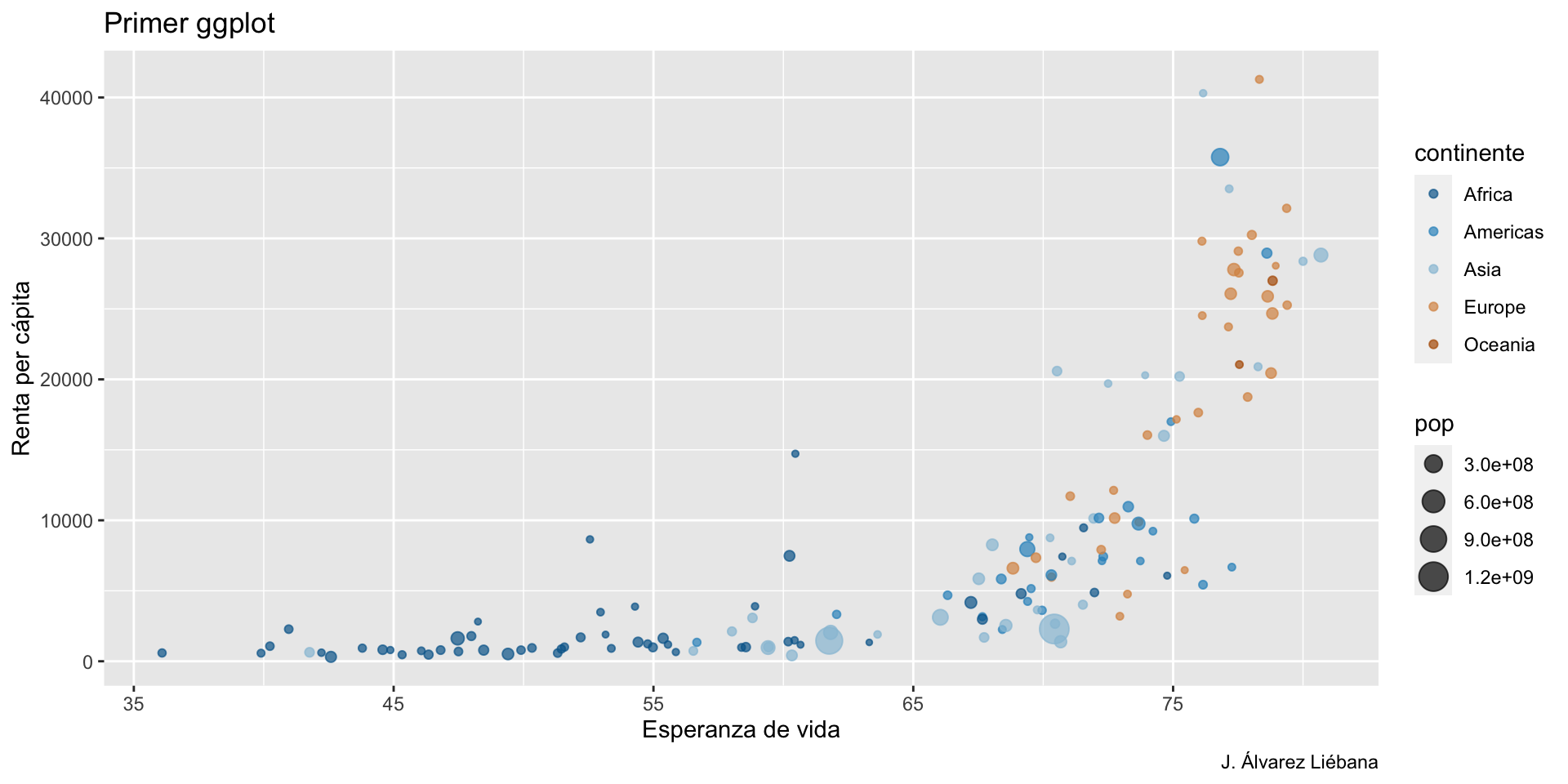
devtools::install_github(repo = "aljrico/harrypotter")
ggplot(gapminder_1997, aes(y = gdpPercap, x = lifeExp, color = continent, size = pop)) +
geom_point(alpha = 0.7) +
scale_x_continuous(breaks = seq(35, 85, by = 10)) +
harrypotter::scale_color_hp_d(option = "ravenclaw")+
labs(x = "Esperanza de vida", y = "Renta per cápita", title = "Primer ggplot",
caption = "J. Álvarez Liébana", color = "continente")Escalas (scale): colores
Incluso cargar paletas de colores diseñadas en base a películas o arte
- cuadros: paquete
{MetBrewer}(repositorio de GithubBlakeRMills/MetBrewer) usandoscale_colour_manual(values = met.brewer(...)).
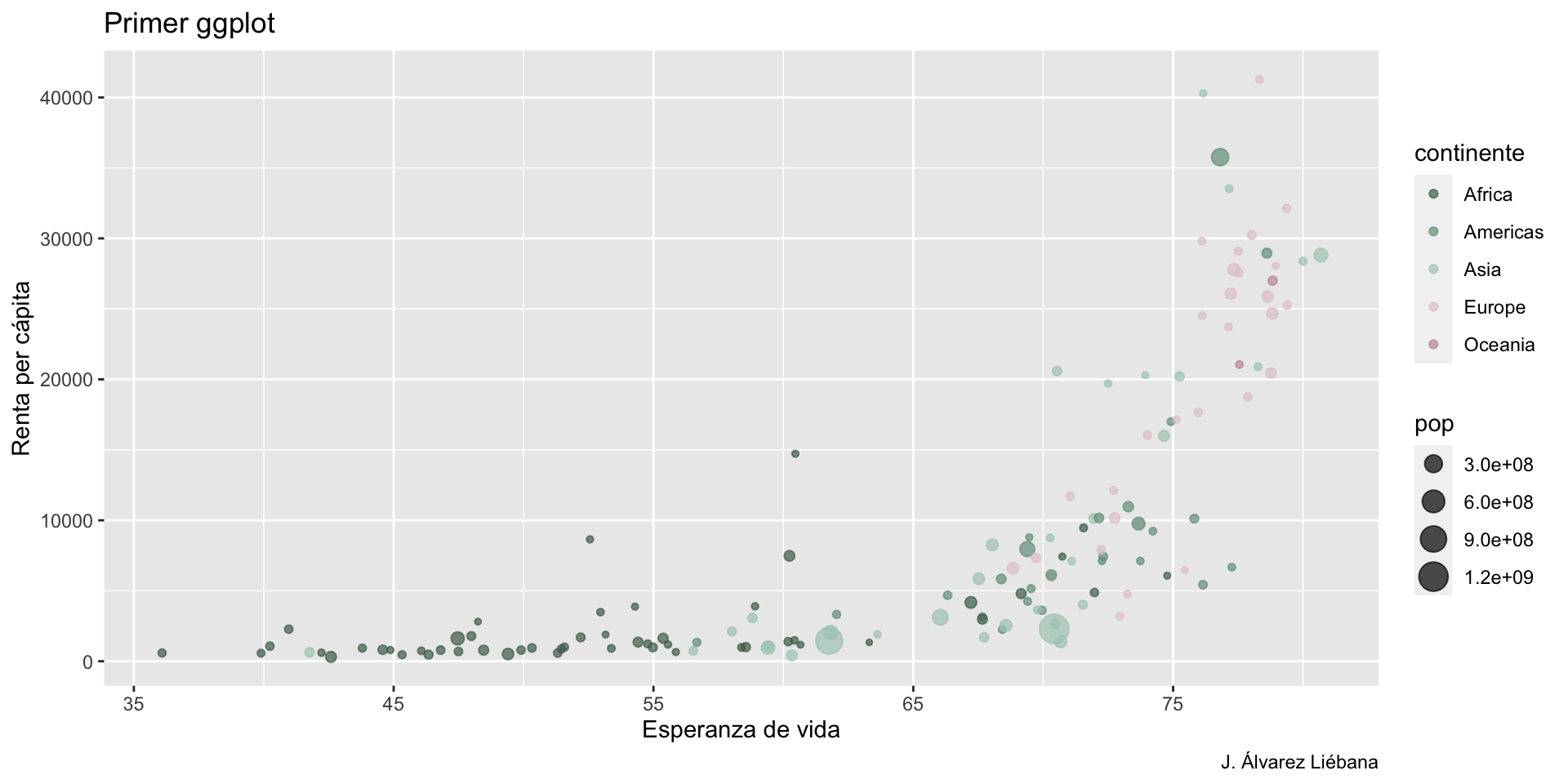
devtools::install_github(repo = "BlakeRMills/MetBrewer")
library(MetBrewer)
ggplot(gapminder_1997, aes(y = gdpPercap, x = lifeExp, color = continent, size = pop)) +
geom_point(alpha = 0.7) +
scale_x_continuous(breaks = seq(35, 85, by = 10)) +
scale_color_manual(values = met.brewer("Monet")) +
labs(x = "Esperanza de vida", y = "Renta per cápita", title = "Primer ggplot",
caption = "J. Álvarez Liébana", color = "continente")Escalas (scale): colores
Incluso cargar paletas de colores diseñadas en base a películas o arte
- discos: paquete
{peRReo}(repositorio de Githubjbgb13/peRReo) usandoscale_colour_manual(values = latin_palette()).
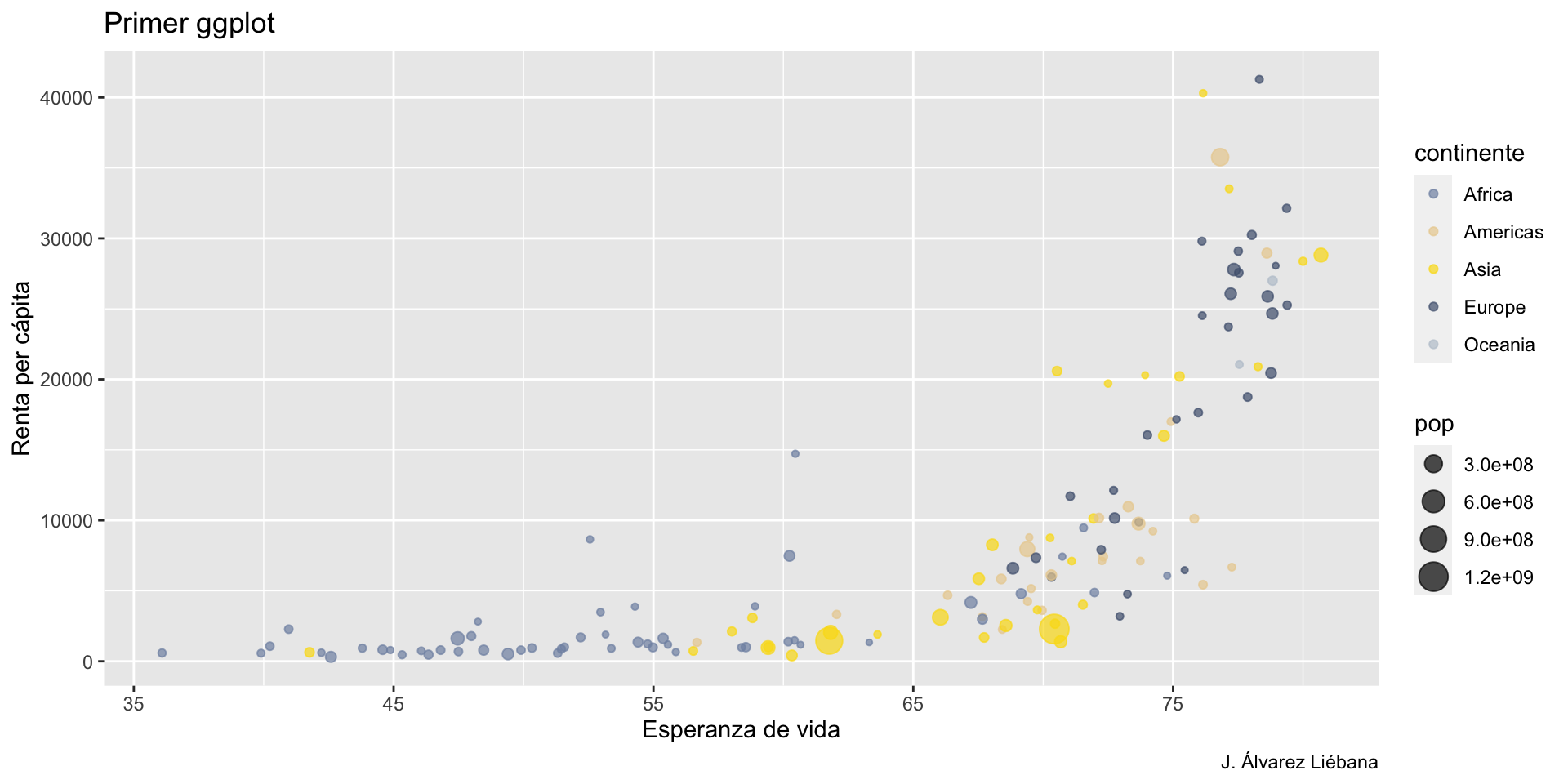
devtools::install_github(repo = "jbgb13/peRReo")
library(peRReo)
ggplot(gapminder_1997, aes(y = gdpPercap, x = lifeExp, color = continent, size = pop)) +
geom_point(alpha = 0.7) +
scale_x_continuous(breaks = seq(35, 85, by = 10)) +
scale_color_manual(values = latin_palette("rosalia")) +
labs(x = "Esperanza de vida", y = "Renta per cápita", title = "Primer ggplot",
caption = "J. Álvarez Liébana", color = "continente")Escalas : otros atributos
Lo mismo que hemos hecho para los ejes o colores podemos hacer para el resto de atríbutos estéticos
Por ejemplo, vamos a indicarle que mapeé el tamaño en función de población pero indicándole el rango de valores (continuo en este caso) entre los que moverse con scale_size_continuous()
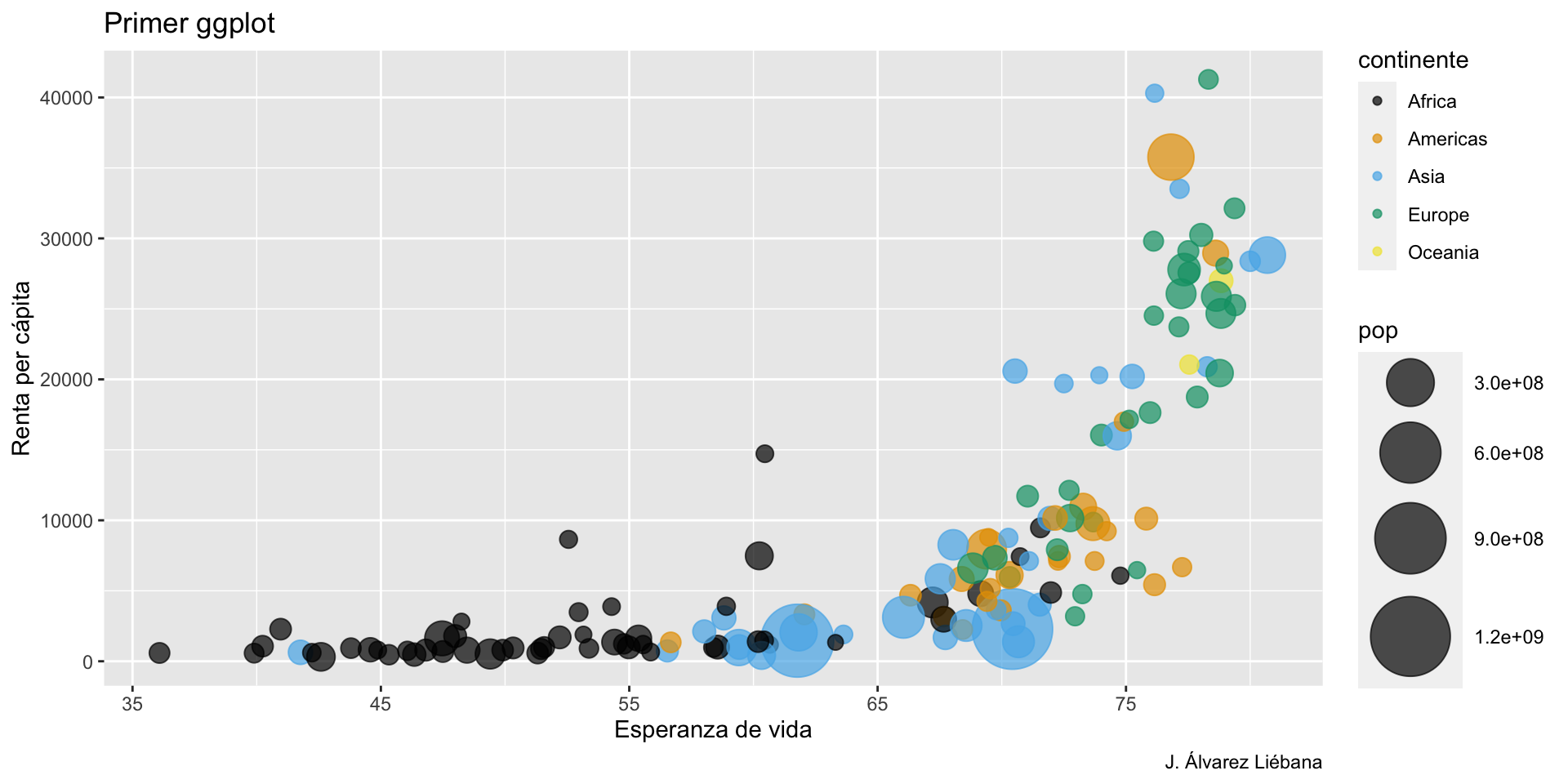
ggplot(gapminder_1997, aes(y = gdpPercap, x = lifeExp, color = continent, size = pop)) +
geom_point(alpha = 0.7) +
scale_x_continuous(breaks = seq(35, 85, by = 10)) +
ggthemes::scale_color_colorblind() +
scale_size_continuous(range = c(3, 15)) +
labs(x = "Esperanza de vida", y = "Renta per cápita", title = "Primer ggplot",
caption = "J. Álvarez Liébana", color = "continente")::: ::::
Tema (básico)
Por último en este primer gráfico, vamos personalizar el tema con alguna de las capas theme_...()
Por ejemplo, vamos a usar theme_minimal() para tener un tema “austero” y minimalista (aprenderemos a definir cada detalle de nuestro tema).
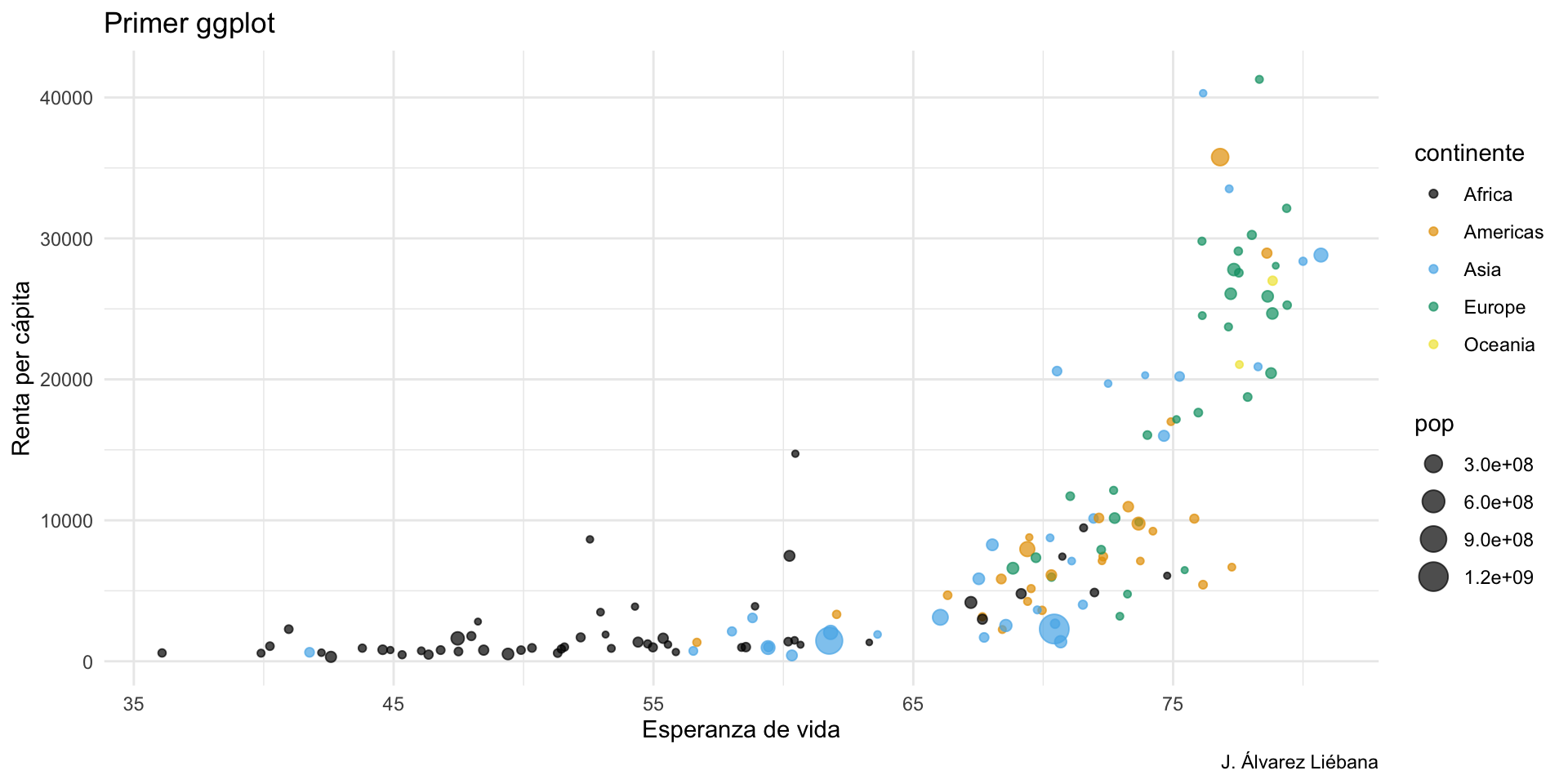
ggplot(gapminder_1997, aes(y = gdpPercap, x = lifeExp, color = continent, size = pop)) +
geom_point(alpha = 0.7) +
scale_x_continuous(breaks = seq(35, 85, by = 10)) +
ggthemes::scale_color_colorblind() +
labs(x = "Esperanza de vida", y = "Renta per cápita", title = "Primer ggplot",
caption = "J. Álvarez Liébana", color = "continente") +
theme_minimal()Resumen de capas
Vamos a hacer un pequeño resumen de lo que llevamos aprendido hasta ahora respecto a {ggplot2}
- Capa de datos: normalmente un gráfico en
{ggplot2}empieza conggplot(datos).
- Mapeo de atributos estéticos: todo lo que queramos que se codifique en función de los datos debe ir dentro de
aes()
- Capa geométrica: para decidir si queremos un scatter plot, un diagrama de barras, un histograma, etc
- Capas de escalas: para decidir los ajustes personalizados de atributos estéticos (escala del alpha o size, paleta de colores, etc)
- Tema: tema para personalizar el gráfico (y etiquetas)
Capa de escalas
Vamos a profundizar un poco dentro de nuestro scatter plot en escalas
- ¿Cómo fijar límites en los ejes? En
scale_x_continuous()yscale_y_continuous(), además de “saltos” podemos indicar límites conlimits = ...
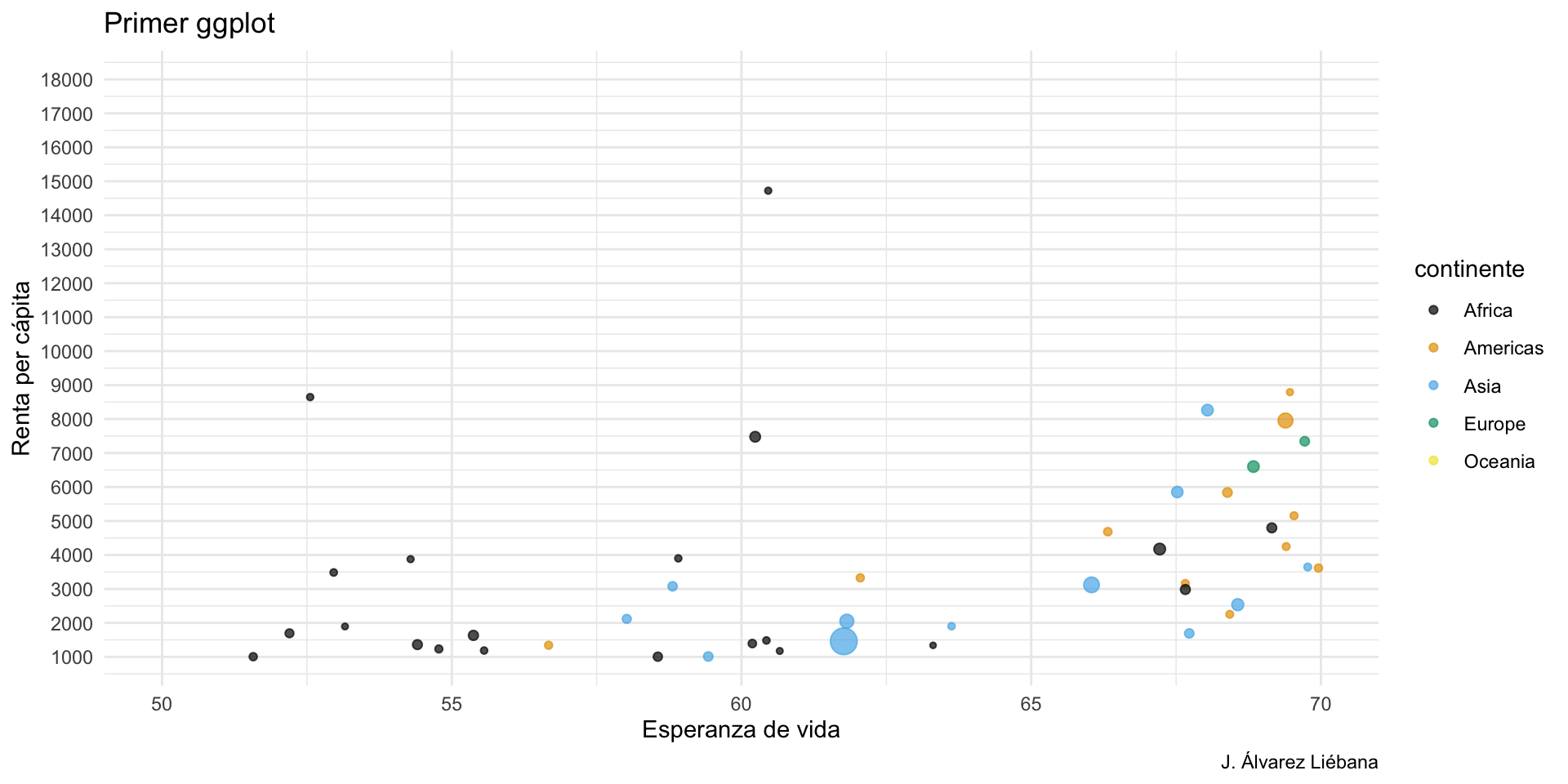
ggplot(gapminder_1997, aes(y = gdpPercap, x = lifeExp, color = continent, size = pop)) +
geom_point(alpha = 0.7) +
scale_x_continuous(limits = c(50, 70), breaks = seq(50, 70, by = 5)) +
scale_y_continuous(limits = c(1000, 18000), breaks = seq(0, 18000, by = 1000)) +
ggthemes::scale_color_colorblind() +
guides(size = "none") +
labs(x = "Esperanza de vida", y = "Renta per cápita", title = "Primer ggplot",
caption = "J. Álvarez Liébana", color = "continente") +
theme_minimal()Capa de escalas
- ¿Cómo etiquetar las unidades de los ejes? Haciendo uso del paquete
{scales}podemos añadir prefijos/sufijos conlabels = label_number(...)
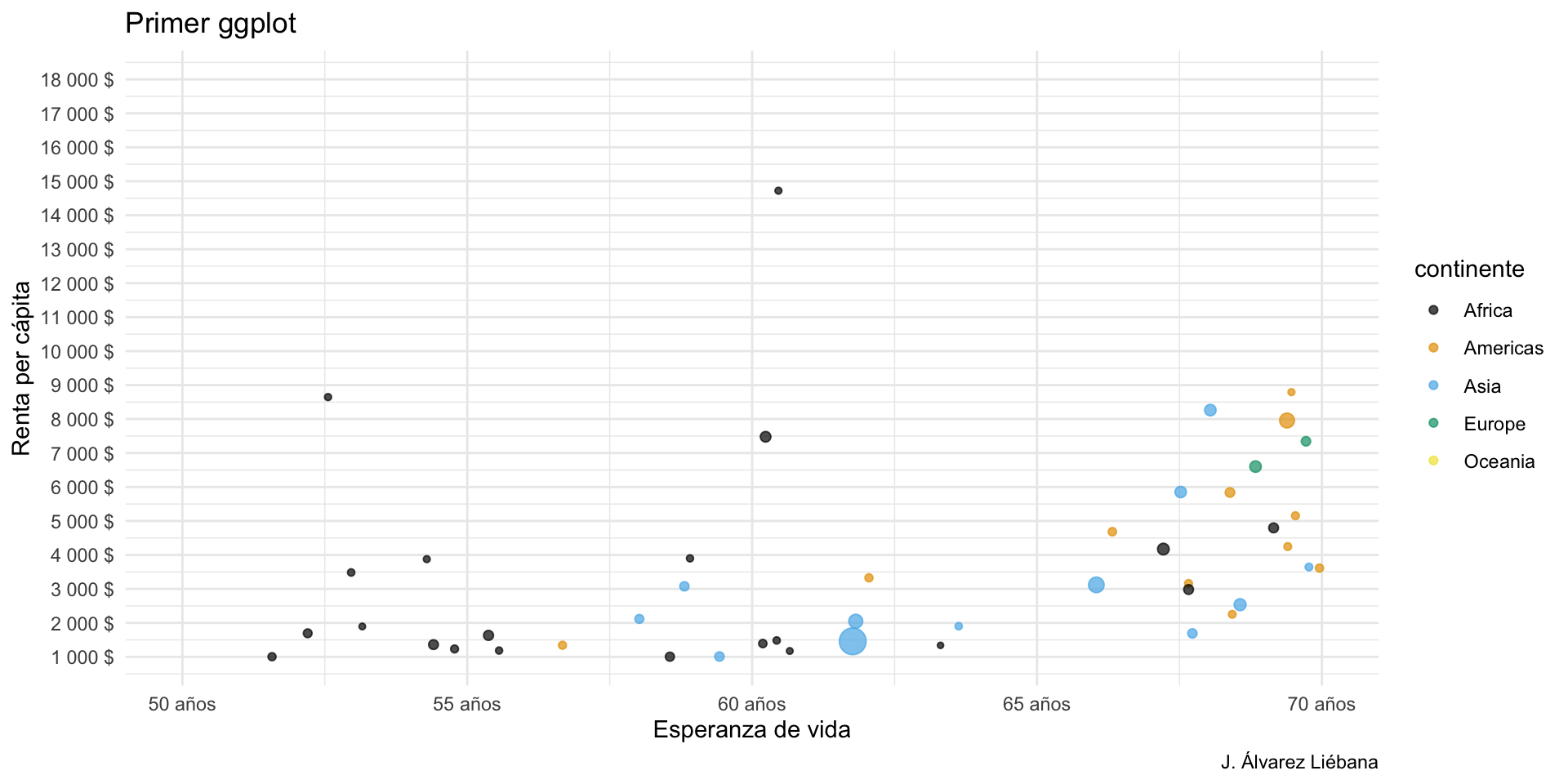
library(scales)
ggplot(gapminder_1997, aes(y = gdpPercap, x = lifeExp, color = continent, size = pop)) +
geom_point(alpha = 0.7) +
scale_x_continuous(limits = c(50, 70), breaks = seq(50, 70, by = 5),
labels = label_number(suffix = " años")) +
scale_y_continuous(limits = c(1000, 18000), breaks = seq(0, 18000, by = 1000),
labels = label_number(suffix = " $")) +
ggthemes::scale_color_colorblind() +
guides(size = "none") +
labs(x = "Esperanza de vida", y = "Renta per cápita", title = "Primer ggplot",
caption = "J. Álvarez Liébana", color = "continente") +
theme_minimal()Capa de escalas
Vamos a profundizar un poco dentro de nuestro scatter plot en escalas
- ¿Cómo cambiar los ajustes de tamaño, alpha, etc? Igual que tenemos
scale_x_...()oscale_color_...(), tenemos tambiénscale_size_...()yscale_alpha_...()
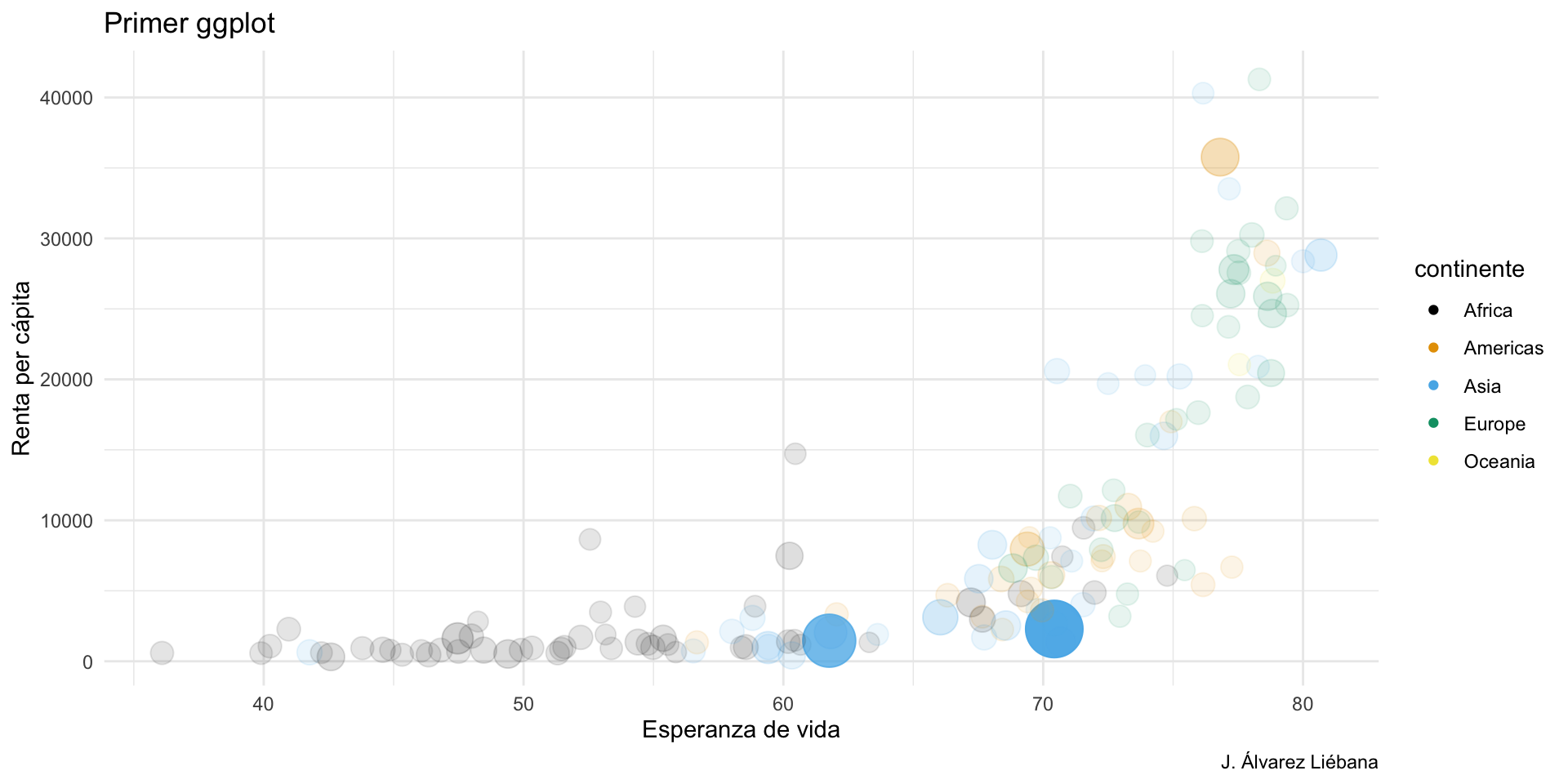
ggplot(gapminder_1997, aes(y = gdpPercap, x = lifeExp, color = continent, size = pop)) +
geom_point(aes(alpha = pop)) +
scale_size(range = c(4, 12)) +
scale_alpha(range = c(0.1, 0.5)) +
ggthemes::scale_color_colorblind() +
guides(size = "none", alpha = "none") +
labs(x = "Esperanza de vida", y = "Renta per cápita", title = "Primer ggplot",
caption = "J. Álvarez Liébana", color = "continente") +
theme_minimal()Capa de escalas
Vamos a profundizar un poco dentro de nuestro scatter plot en escalas
- ¿Cómo cambiar la escala (relación) lineal entre los ejes? Con
scale_x_sqrt()oscale_x_log10()podemos cambiar la escala de los ejes.
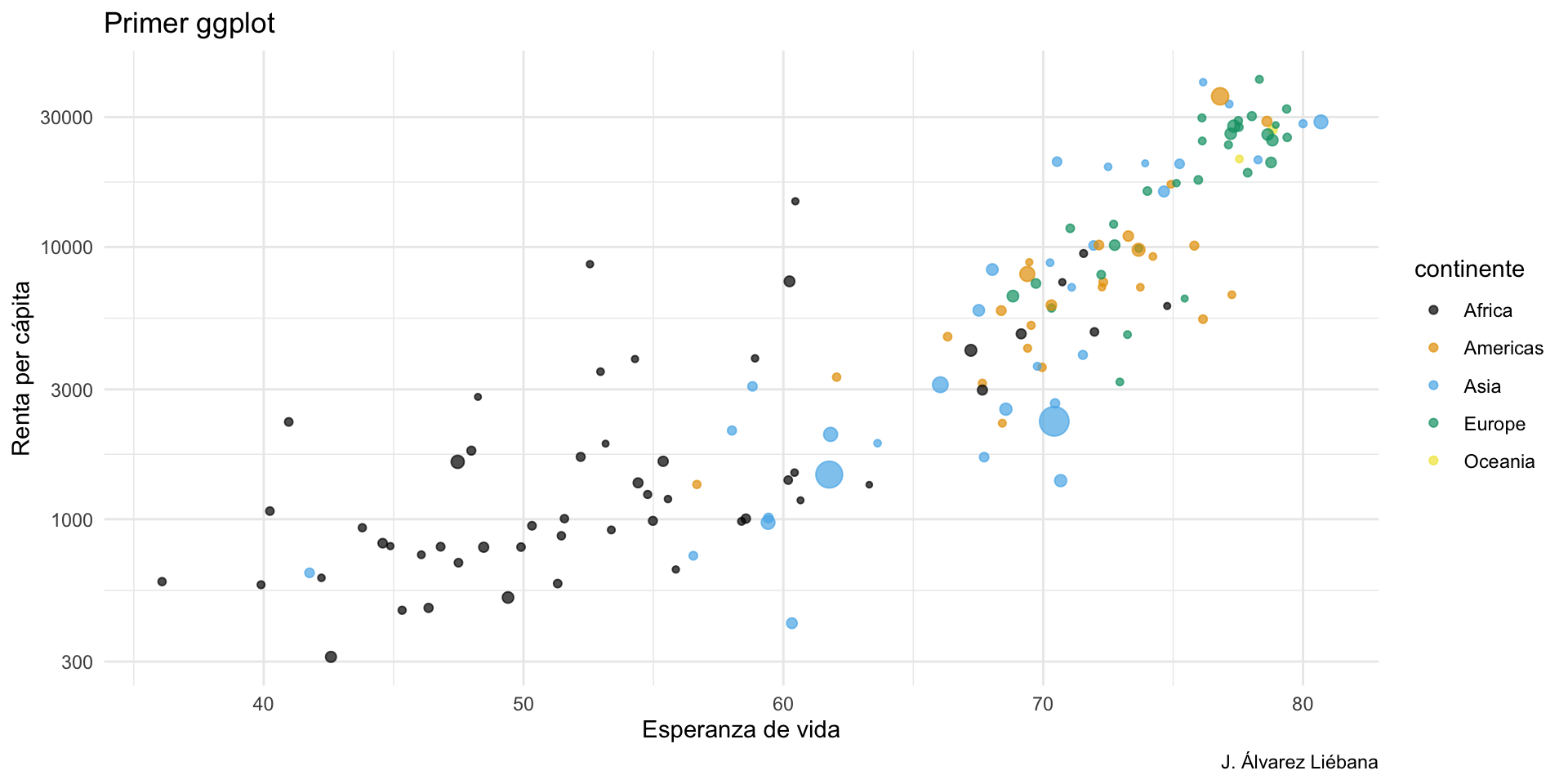
ggplot(gapminder_1997, aes(y = gdpPercap, x = lifeExp, color = continent, size = pop)) +
geom_point(alpha = 0.7) +
scale_y_log10() +
ggthemes::scale_color_colorblind() +
guides(size = "none") +
labs(x = "Esperanza de vida", y = "Renta per cápita", title = "Primer ggplot",
caption = "J. Álvarez Liébana", color = "continente") +
theme_minimal()scale_color: paleta Brewer
Una de las capas de escalas más importantes son las capas de color. Ya vimos como definir paletas manuales, ¿pero qué opciones hay para escalas continuas de colores?
Existen unas paletas de colores conocidas como ColorBrewer pudiendo definirse de manera secuencial, divergente o de manera cualitativa (ver info en https://colorbrewer2.org)
maxcolors category colorblind
BrBG 11 div TRUE
PiYG 11 div TRUE
PRGn 11 div TRUE
PuOr 11 div TRUE
RdBu 11 div TRUE
RdGy 11 div FALSE
RdYlBu 11 div TRUE
RdYlGn 11 div FALSE
Spectral 11 div FALSE
Accent 8 qual FALSE
Dark2 8 qual TRUE
Paired 12 qual TRUE
Pastel1 9 qual FALSE
Pastel2 8 qual FALSE
Set1 9 qual FALSE
Set2 8 qual TRUE
Set3 12 qual FALSE
Blues 9 seq TRUE
BuGn 9 seq TRUE
BuPu 9 seq TRUE
GnBu 9 seq TRUE
Greens 9 seq TRUE
Greys 9 seq TRUE
Oranges 9 seq TRUE
OrRd 9 seq TRUE
PuBu 9 seq TRUE
PuBuGn 9 seq TRUE
PuRd 9 seq TRUE
Purples 9 seq TRUE
RdPu 9 seq TRUE
Reds 9 seq TRUE
YlGn 9 seq TRUE
YlGnBu 9 seq TRUE
YlOrBr 9 seq TRUE
YlOrRd 9 seq TRUEscale_color: paleta Brewer
Con RColorBrewer::brewer.pal() podemos obtener el vector de n colores para una paleta dada
[1] "#D7191C" "#FDAE61" "#FFFFBF" "#ABD9E9" "#2C7BB6"scale_color: paleta Brewer
Para incluirlo podemos usar scale_colour_brewer() o bien scale_color_distiller() si queremos crear una escala continua (interpolando entre los colores)
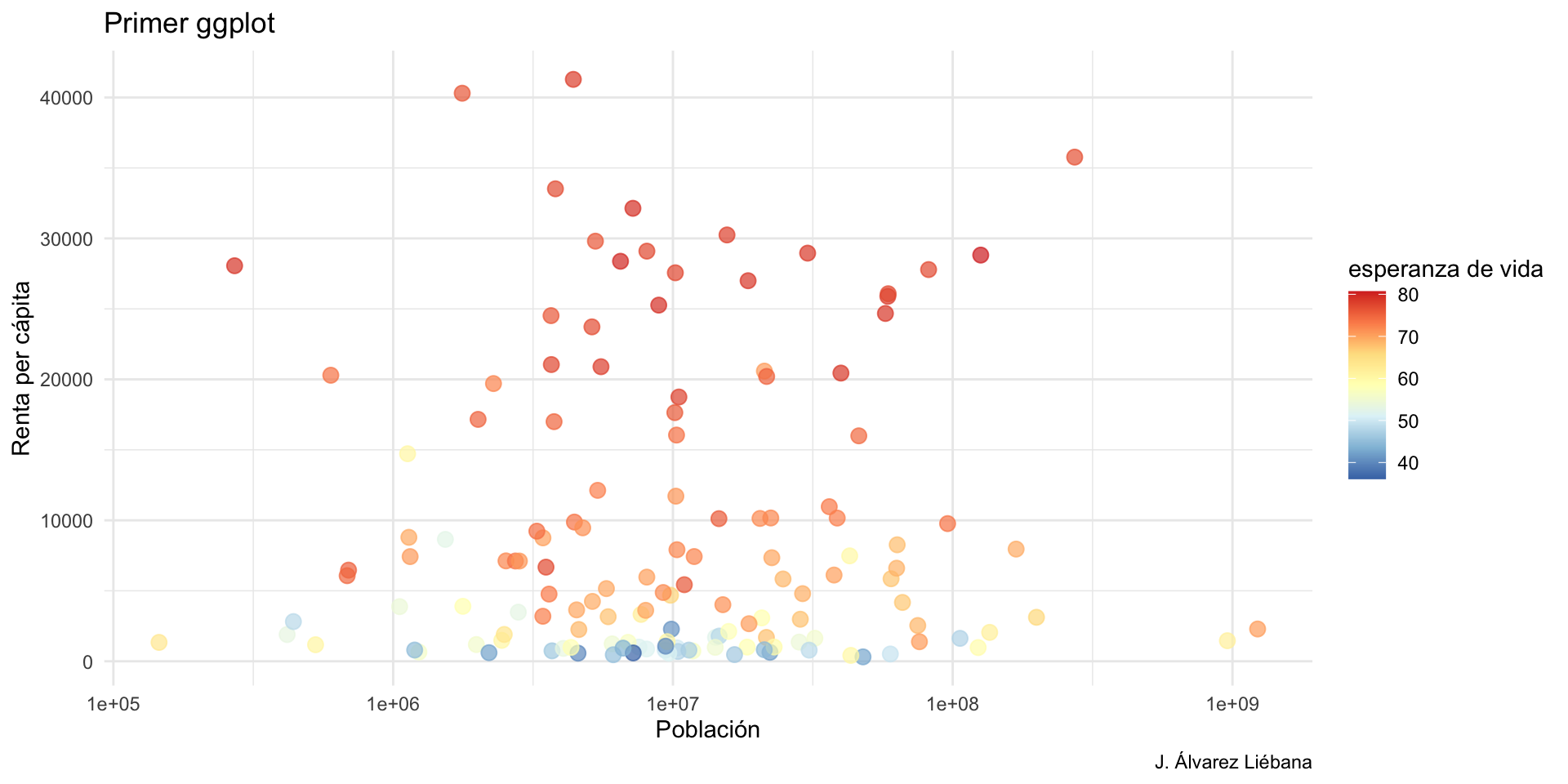
ggplot(gapminder_1997, aes(y = gdpPercap, x = pop, color = lifeExp)) +
geom_point(alpha = 0.7, size = 3) +
scale_x_log10() +
scale_color_distiller(palette = "RdYlBu") +
guides(size = "none") +
labs(x = "Población", y = "Renta per cápita", title = "Primer ggplot",
caption = "J. Álvarez Liébana", color = "esperanza de vida") +
theme_minimal()Gradiente de color manual
Tambien podemos crear un gradiente de color manual son scale_..._gradient() para dos colores, scale_..._gradient2() para tres colores (bajo, medio y alto) y scale_..._gradientn() para n colores
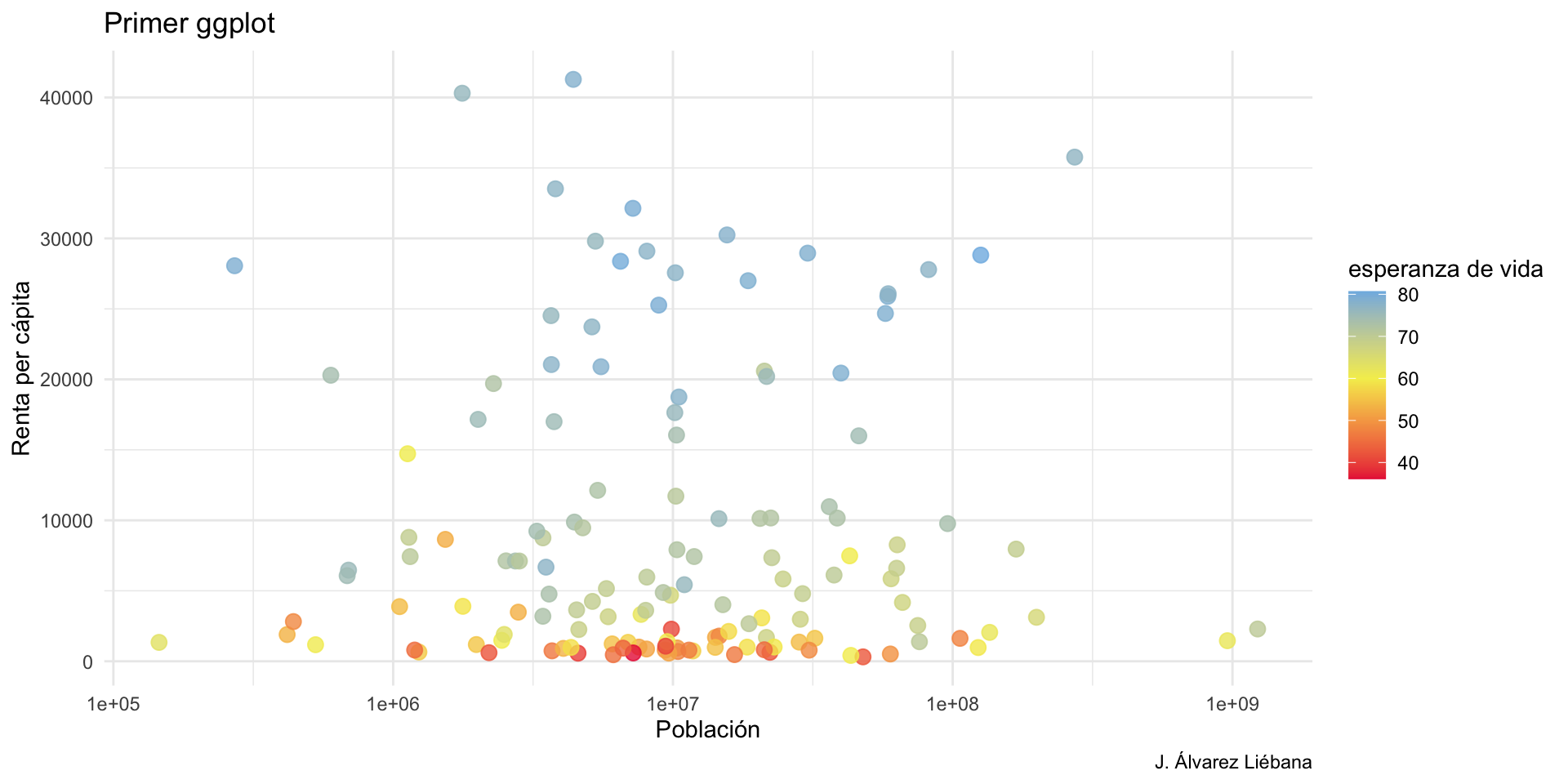
ggplot(gapminder_1997, aes(y = gdpPercap, x = pop, color = lifeExp)) +
geom_point(alpha = 0.8, size = 3) +
scale_x_log10() +
scale_color_gradient2(low = "#E92745", mid = "#F4ED5B", high = "#56B1F7", midpoint = 60) +
labs(x = "Población", y = "Renta per cápita", title = "Primer ggplot",
caption = "J. Álvarez Liébana", color = "esperanza de vida") +
theme_minimal()Capa de coordenadas
Además de escalas tenemos una capa de coordenadas con coord_... para indicar si queremos un sistema cartesiano (y sus límites), coordenadas polares (coord_polar()), si queremos coordenadas iguales (coord_equal()) o invertir su rol (coord_flip())
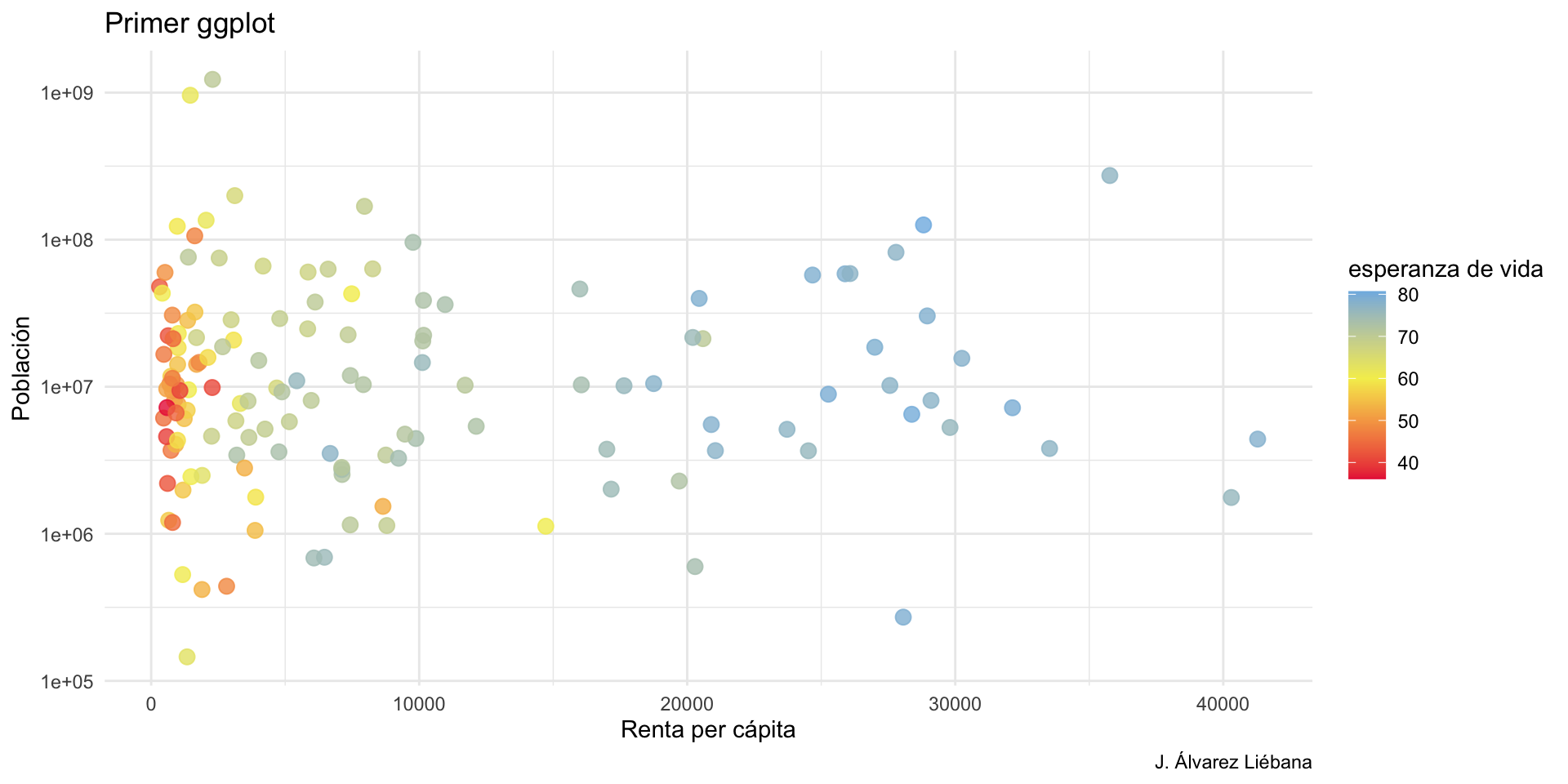
ggplot(gapminder_1997, aes(y = gdpPercap, x = pop, color = lifeExp)) +
geom_point(alpha = 0.8, size = 3) +
scale_x_log10() +
scale_color_gradient2(low = "#E92745", mid = "#F4ED5B", high = "#56B1F7", midpoint = 60) +
coord_flip() +
labs(x = "Población", y = "Renta per cápita", title = "Primer ggplot",
caption = "J. Álvarez Liébana", color = "esperanza de vida") +
theme_minimal()Capa de stats
Una capa importante es la capa de estadísticas
stat_smooth(): visualiza un ajuste suavizado de los datos (reg. lineal, glm, loess, gam, etc).
Con stat_smooth(method = "lm", se = FALSE) una recta de regresión (sin intervalos).
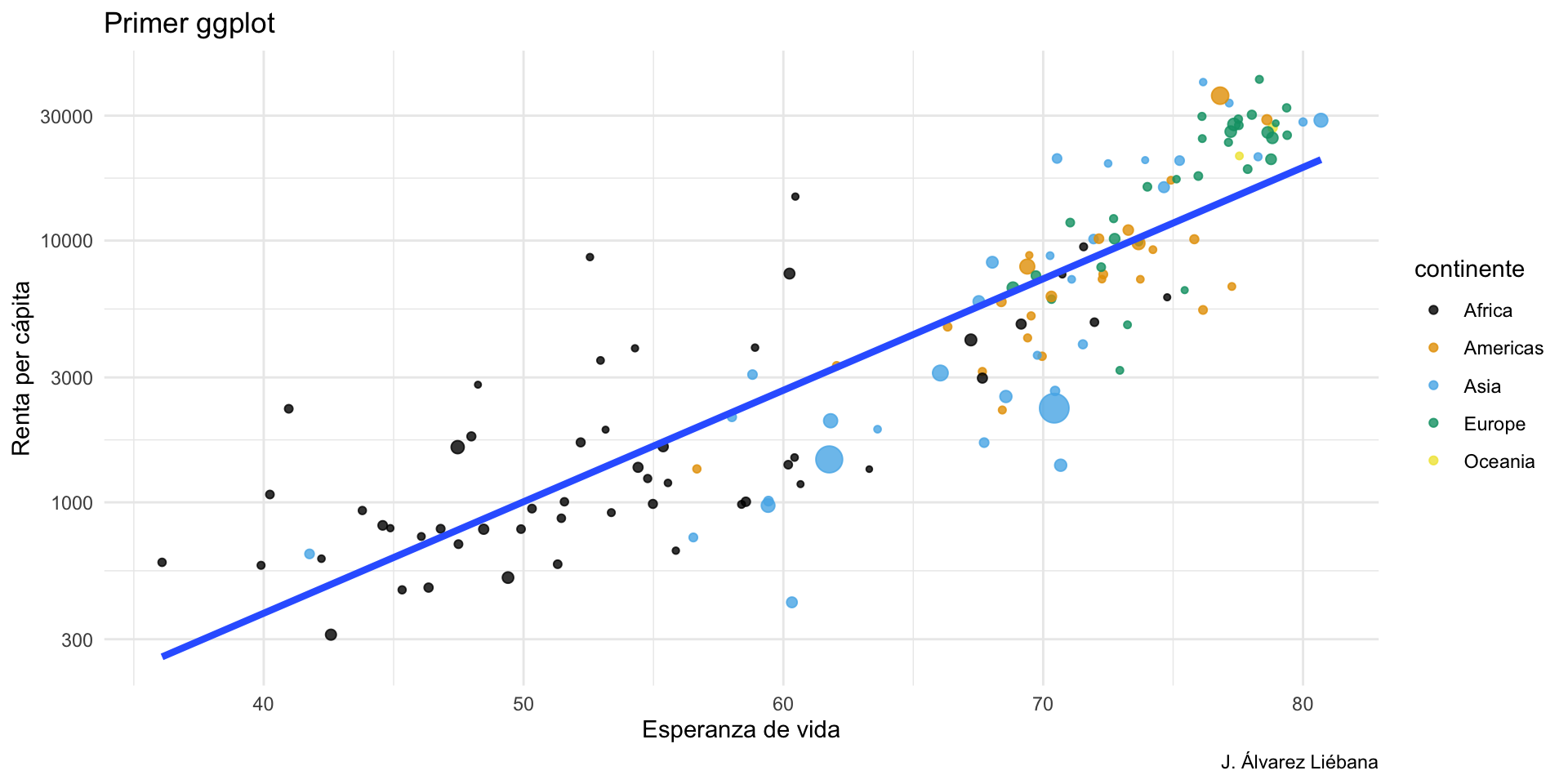
ggplot(gapminder_1997, aes(y = gdpPercap, x = lifeExp)) +
geom_point(aes(color = continent, size = pop), alpha = 0.8) +
stat_smooth(method = "lm", se = FALSE, linewidth = 1.5) +
scale_y_log10() +
guides(size = "none") +
ggthemes::scale_color_colorblind() +
labs(x = "Esperanza de vida", y = "Renta per cápita", title = "Primer ggplot",
caption = "J. Álvarez Liébana", color = "continente") +
theme_minimal()Capa de stats
Una capa importante es la capa de estadísticas que nos permite combinar en nuestro gráfico algunas funcionalidades
Fíjate que si usas en la primera capa parámetros estéticos se acaban heredando a capas posteriores, en concreto al ajuste visualizado.
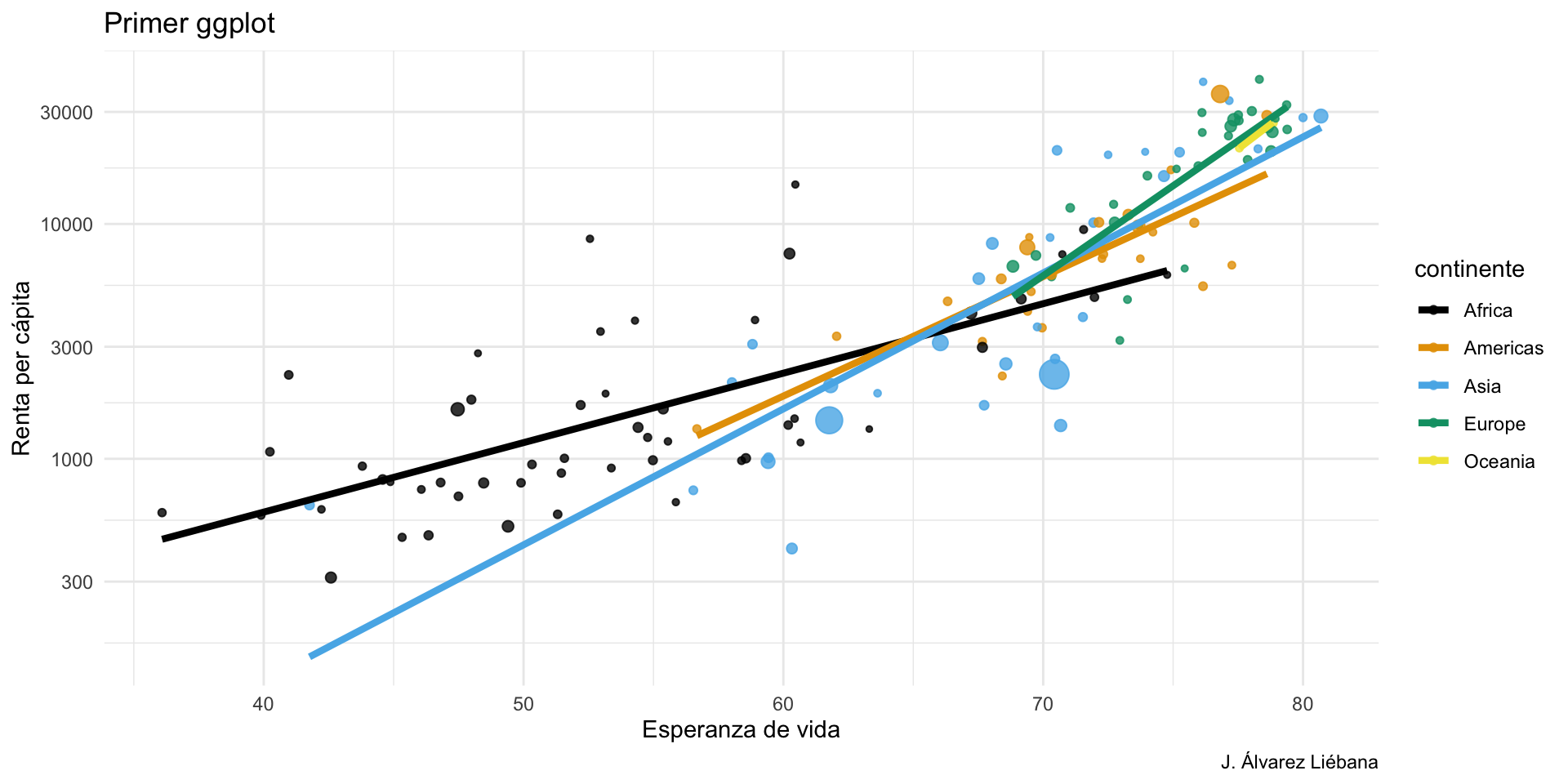
ggplot(gapminder_1997, aes(y = gdpPercap, x = lifeExp, color = continent, size = pop)) +
geom_point(alpha = 0.8) +
stat_smooth(method = "lm", se = FALSE, linewidth = 1.5) +
scale_y_log10() +
guides(size = "none") +
ggthemes::scale_color_colorblind() +
labs(x = "Esperanza de vida", y = "Renta per cápita", title = "Primer ggplot",
caption = "J. Álvarez Liébana", color = "continente") +
theme_minimal()geom_text()
Podemos añadirle textos simples con geom_text(label = ...), por ejemplo, para añadir la correlación del ajuste.
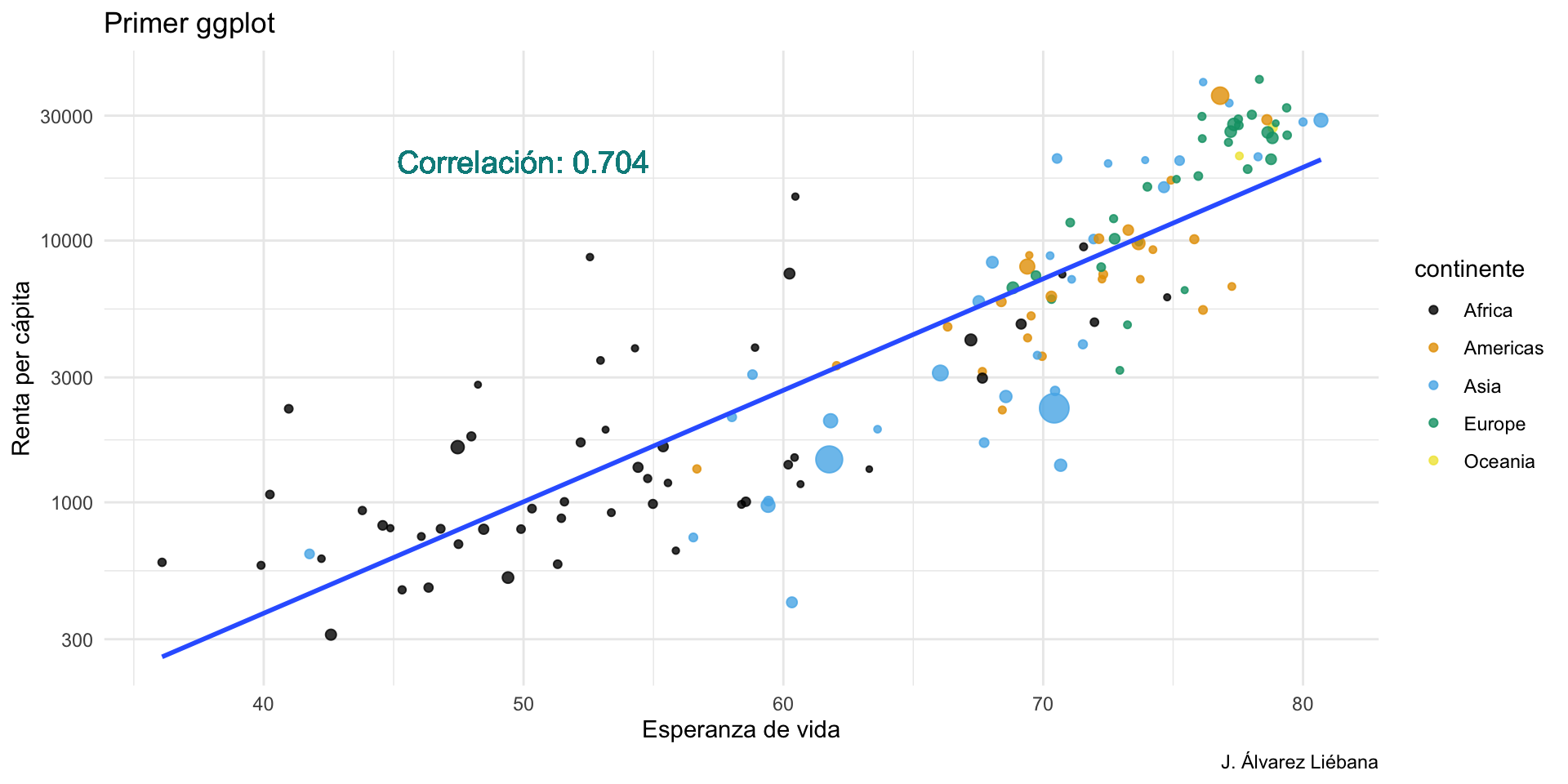
cor <- round(cor(gapminder_1997$gdpPercap, gapminder_1997$lifeExp), 3)
ggplot(gapminder_1997, aes(y = gdpPercap, x = lifeExp)) +
geom_point(aes(color = continent, size = pop), alpha = 0.8) +
stat_smooth(method = "lm", se = FALSE) +
geom_text(aes(x = 50, y = 20000, label = glue("Correlación: {cor}")),
size = 5, color = "darkcyan") +
scale_y_log10() +
guides(size = "none") +
ggthemes::scale_color_colorblind() +
labs(x = "Esperanza de vida", y = "Renta per cápita", title = "Primer ggplot",
caption = "J. Álvarez Liébana", color = "continente") +
theme_minimal()Capa de stats
Dentro de stat_smooth() podemos especificarle otro ajuste polinómico dándole expresión en formula = ...
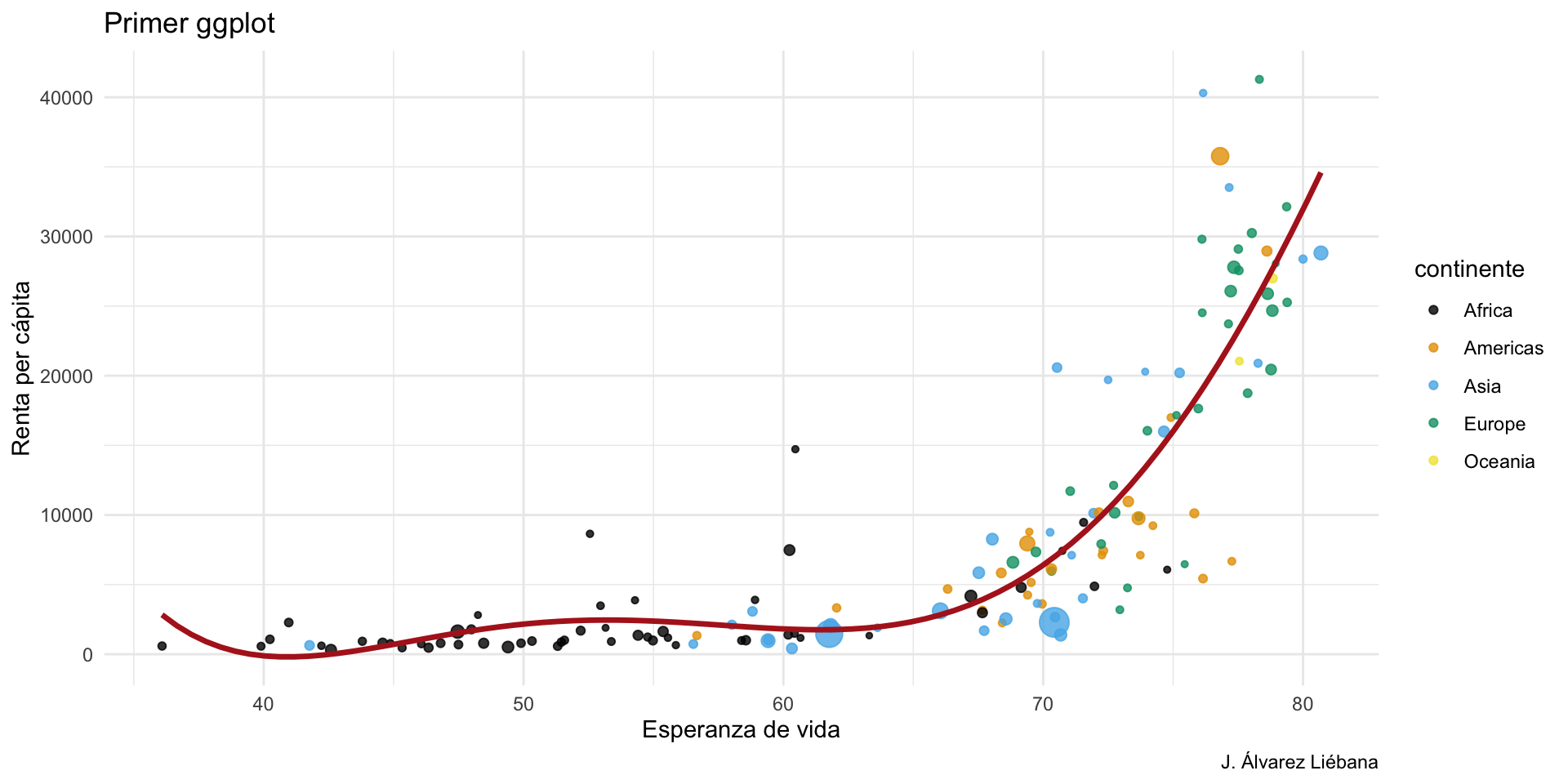
ggplot(gapminder_1997, aes(y = gdpPercap, x = lifeExp)) +
geom_point(aes(color = continent, size = pop), alpha = 0.8) +
stat_smooth(method = "lm", formula = y ~ x + I(x^2) + I(x^3) + I(x^4) + I(x^5),
color = "firebrick", se = FALSE, linewidth = 1.2) +
guides(size = "none") +
ggthemes::scale_color_colorblind() +
labs(x = "Esperanza de vida", y = "Renta per cápita", title = "Primer ggplot",
caption = "J. Álvarez Liébana", color = "continente") +
theme_minimal()Capa de stats
Sin method especificado ajuste por un LOESS (menos de 1000 puntos) o GAM (más de 1000 puntos)
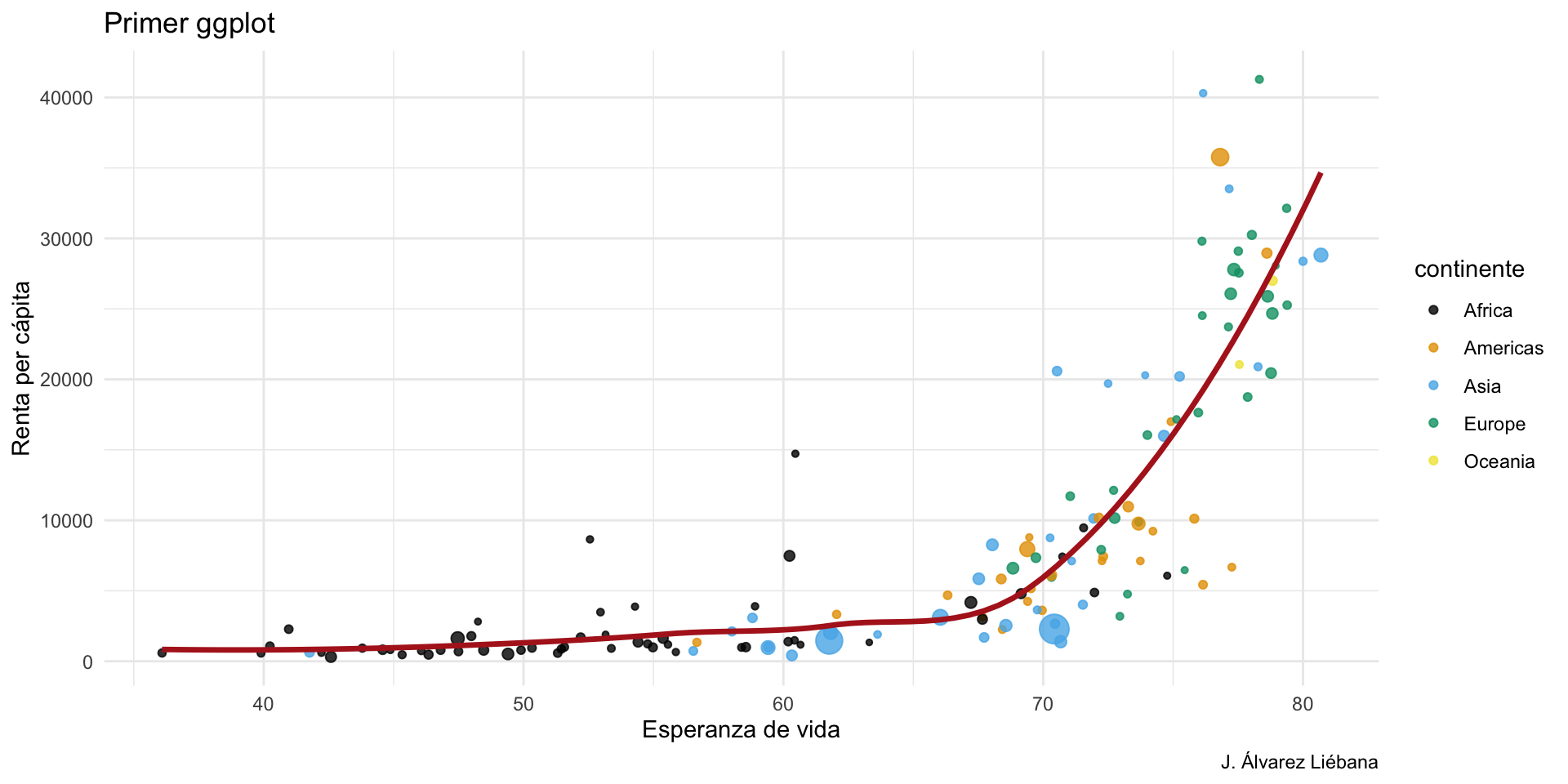
ggplot(gapminder_1997, aes(y = gdpPercap, x = lifeExp)) +
geom_point(aes(color = continent, size = pop), alpha = 0.8) +
stat_smooth(color = "firebrick", se = FALSE, linewidth = 1.2) +
guides(size = "none") +
ggthemes::scale_color_colorblind() +
labs(x = "Esperanza de vida", y = "Renta per cápita", title = "Primer ggplot",
caption = "J. Álvarez Liébana", color = "continente") +
theme_minimal()Capa de stats
Con stat_summary() podemos incluso añadir estadísticas por grupos, como la media o mediana.
ggplot(gapminder, aes(y = gdpPercap, x = year)) +
geom_point(size = 1.7, alpha = 0.2) +
stat_summary(fun = "mean", size = 0.4, color = "coral") +
stat_summary(fun = "median", size = 0.4, color = "darkcyan") +
ggthemes::scale_color_colorblind() +
labs(x = "Esperanza de vida", y = "Renta per cápita", title = "Primer ggplot",
caption = "J. Álvarez Liébana", color = "continente") +
theme_minimal()Capa de stats
Fíjate que si no tenemos una variable cuali, la media la hace con n = 1 (es decir, es el propio punto).
ggplot(gapminder, aes(y = gdpPercap, x = pop)) +
geom_point(size = 1.7, alpha = 0.2) +
stat_summary(fun = "mean", size = 0.4, color = "coral") +
stat_summary(fun = "median", size = 0.4, color = "darkcyan") +
ggthemes::scale_color_colorblind() +
labs(x = "Esperanza de vida", y = "Renta per cápita", title = "Primer ggplot",
caption = "J. Álvarez Liébana", color = "continente") +
theme_minimal()Componiendo (facet)
También podemos desagregar los gráficos (facetar) por grupos, equivalente al group_by() en tidyverse.
Por ejemplo, vamos a crear un gráfico por continente, mostrando todos los gráficos a la vez (pero por separado) con facet_wrap(~continent).
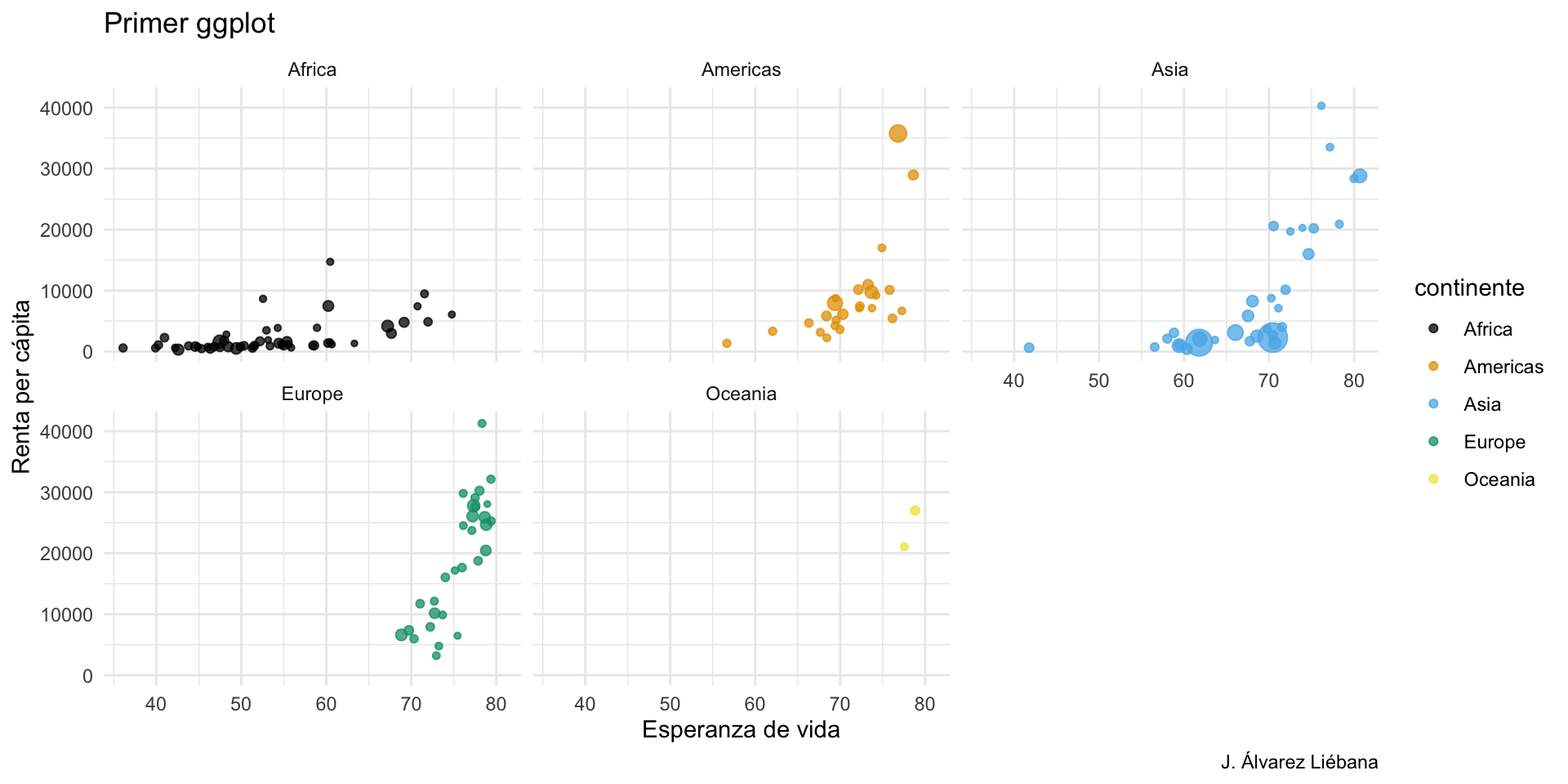
ggplot(gapminder_1997, aes(y = gdpPercap, x = lifeExp, size = pop, color = continent)) +
geom_point(alpha = 0.75) +
ggthemes::scale_color_colorblind() +
facet_wrap(~continent) +
guides(size = "none") +
labs(x = "Esperanza de vida", y = "Renta per cápita", title = "Primer ggplot",
caption = "J. Álvarez Liébana", color = "continente") +
theme_minimal()Componiendo (facet)
También podemos desagregar los gráficos (facetar) por grupos, equivalente al group_by() en tidyverse.
Por defecto las escalas en los ejes son compartidas. Si queremos que la escala de los ejes vaya por libre debemos usar scales = "free_x", scales = "free_y" o scales = "free"
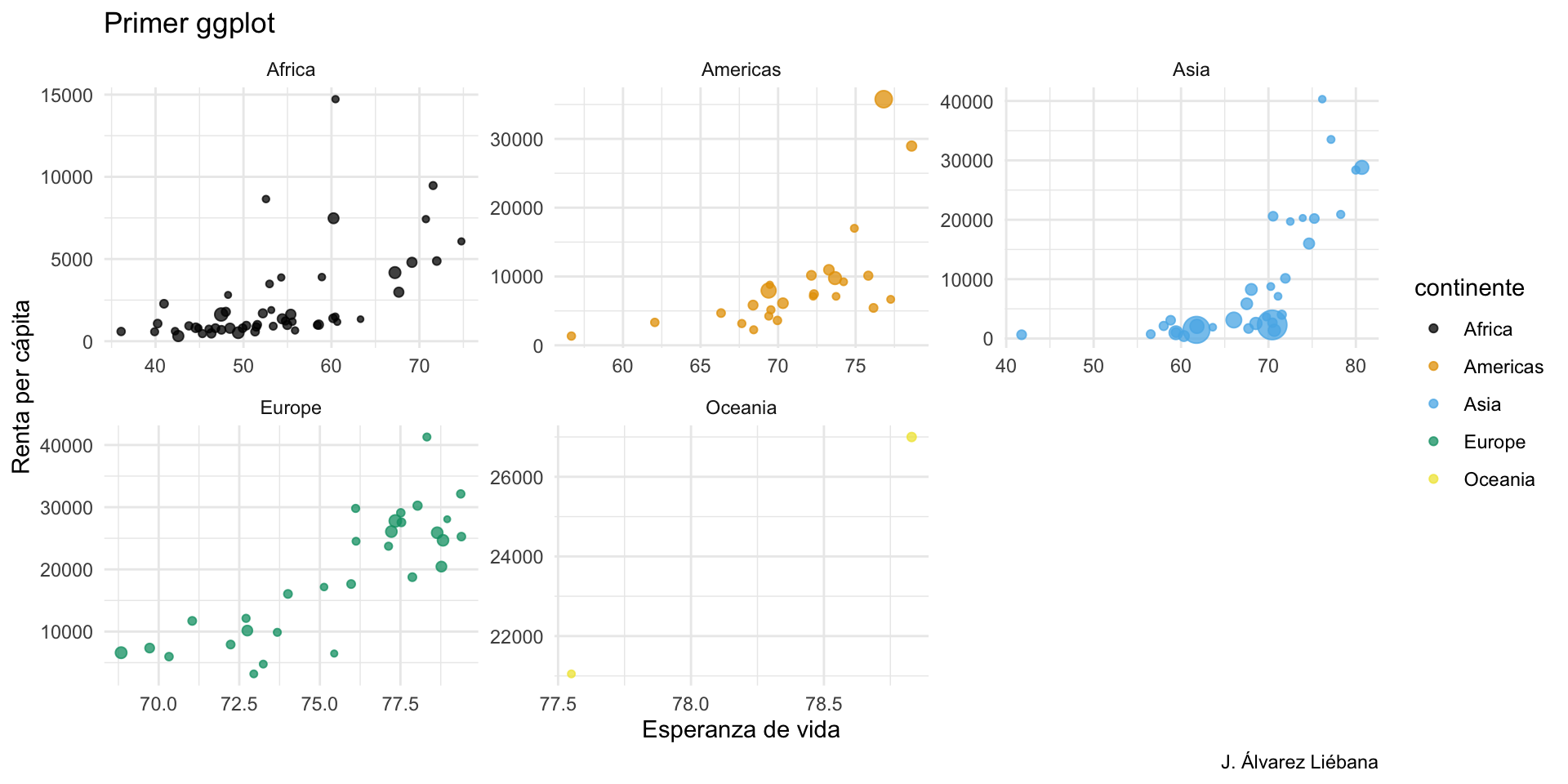
ggplot(gapminder_1997, aes(y = gdpPercap, x = lifeExp, size = pop, color = continent)) +
geom_point(alpha = 0.75) +
ggthemes::scale_color_colorblind() +
facet_wrap(~continent, scales = "free") +
guides(size = "none") +
labs(x = "Esperanza de vida", y = "Renta per cápita", title = "Primer ggplot",
caption = "J. Álvarez Liébana", color = "continente") +
theme_minimal()Componiendo (facet)
También podemos desagregar los gráficos (facetar) por grupos, equivalente al group_by() en tidyverse.
Con nrow = ... y ncol = ... podemos especificar cuantas columnas y filas tenemos en la cuadrícula de gráficas
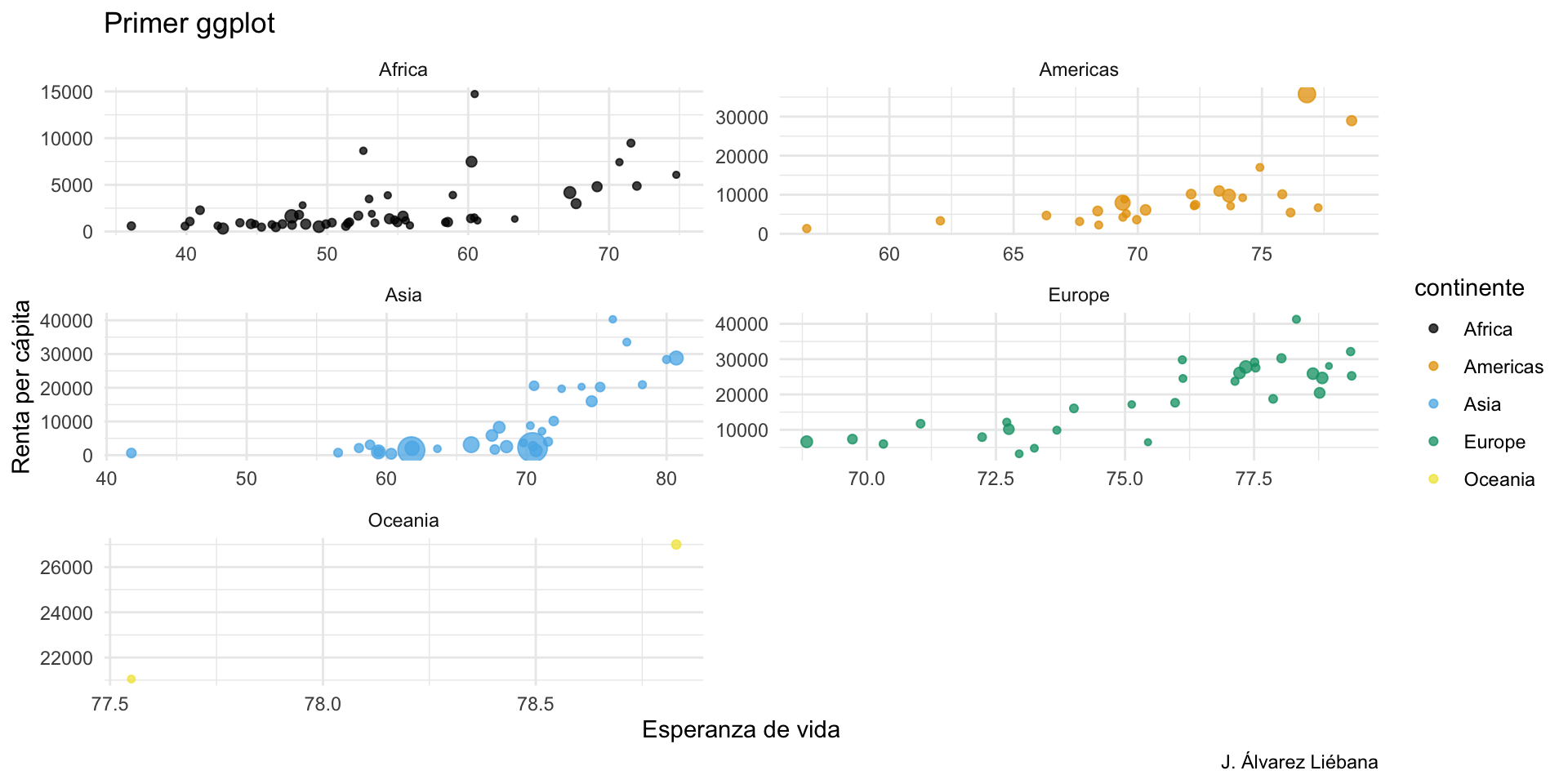
ggplot(gapminder_1997, aes(y = gdpPercap, x = lifeExp, size = pop, color = continent)) +
geom_point(alpha = 0.75) +
ggthemes::scale_color_colorblind() +
facet_wrap(~continent, scales = "free", nrow = 3) +
guides(size = "none") +
labs(x = "Esperanza de vida", y = "Renta per cápita", title = "Primer ggplot",
caption = "J. Álvarez Liébana", color = "continente") +
theme_minimal()Componiendo (facet)
También le podemos pasar dos argumentos (variables) para formar un grid de gráficas
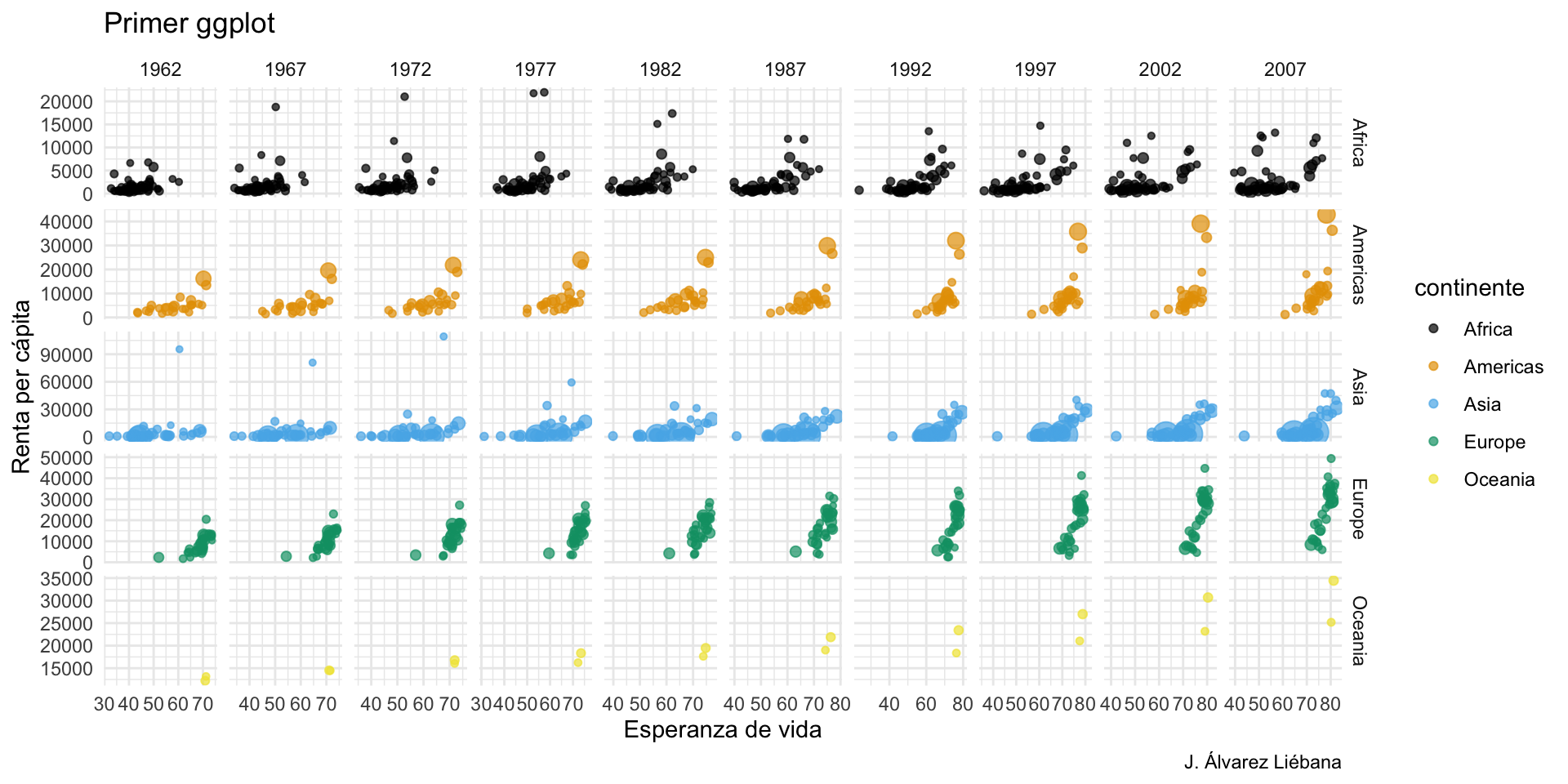
ggplot(gapminder |> filter(year >= 1962), aes(y = gdpPercap, x = lifeExp, size = pop, color = continent)) +
geom_point(alpha = 0.7) +
ggthemes::scale_color_colorblind() +
facet_grid(continent ~ year, scales = "free") +
guides(size = "none") +
labs(x = "Esperanza de vida", y = "Renta per cápita", title = "Primer ggplot",
caption = "J. Álvarez Liébana", color = "continente") +
theme_minimal()Componiendo (facet)
Aprenderemos distintas personalizaciones del tema pero con theme(legend.position = ...) podemos decidir la posición de la leyenda
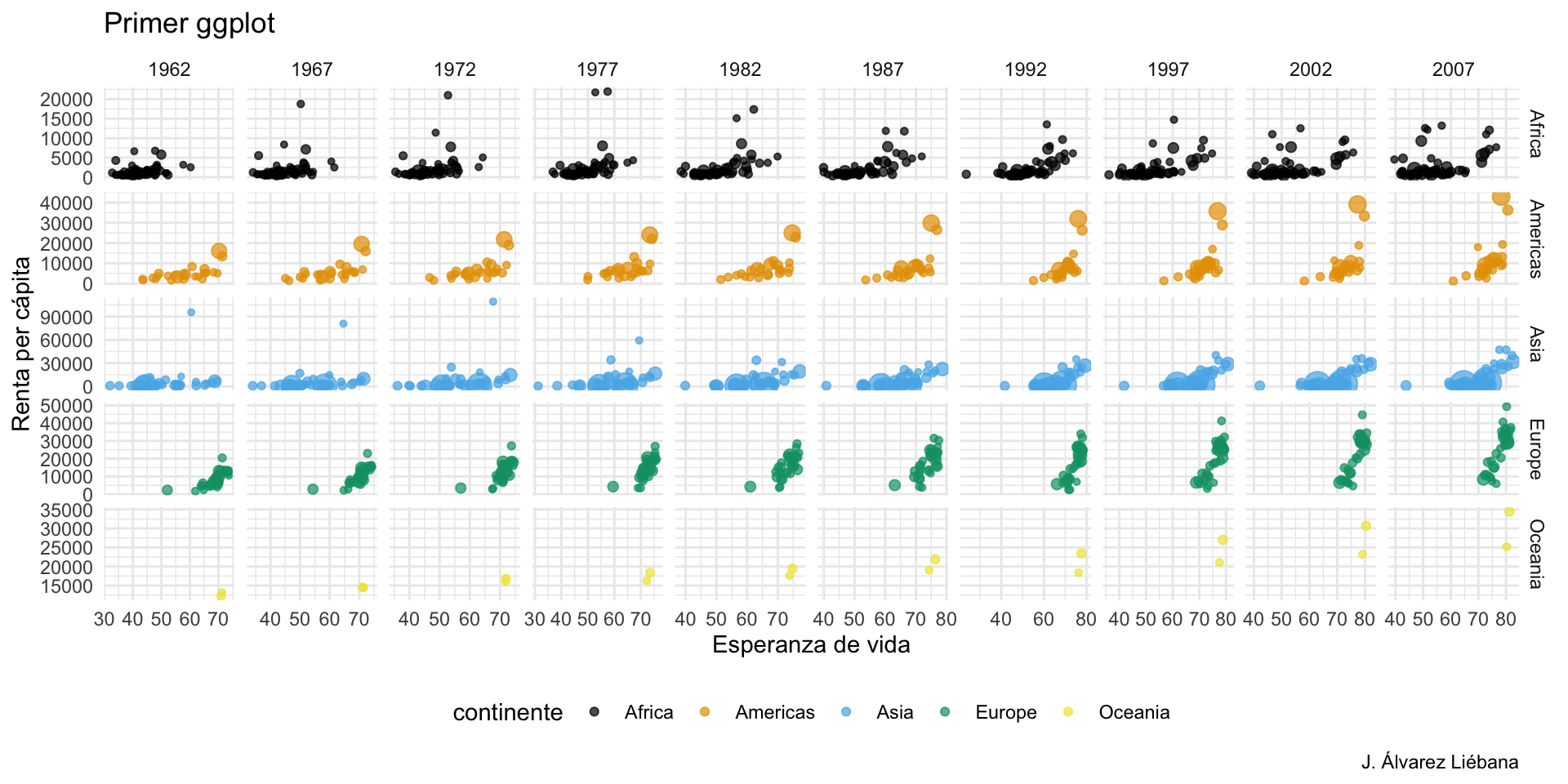
ggplot(gapminder |> filter(year >= 1962), aes(y = gdpPercap, x = lifeExp, size = pop, color = continent)) +
geom_point(alpha = 0.7) +
ggthemes::scale_color_colorblind() +
facet_grid(continent ~ year, scales = "free") +
guides(size = "none") +
labs(x = "Esperanza de vida", y = "Renta per cápita", title = "Primer ggplot",
caption = "J. Álvarez Liébana", color = "continente") +
theme_minimal() +
theme(legend.position = "bottom")Clase 6: profundizando en la visualización
Visualización de datos
Variables continuas
Hemos aprendido a realizar uno de los gráficos más famosos, un diagrama de dispersión, pero…¿qué propiedades deben cumplir las variables?
Para visualizar dos variables con un diagrama de dispersión es necesario que ambas sean variables numéricas continuas
¿Se te ocurre algúna gráfico básico para variables discretas?
Cualis: barras
¿Y si tengo variables discretas o cualitativas?
Cualis: barras
La ventaja de ggplot es que, al trabajar por capas, todo lo que hemos aprendido nos sirve: solo tenemos que cambiar la geometría.
En este caso para realizar un diagrama de barras usaremos geom_bar() en lugar de geom_point(), indicando solo la variable de grupo con x = sex (ggplot hará solo el recuento)
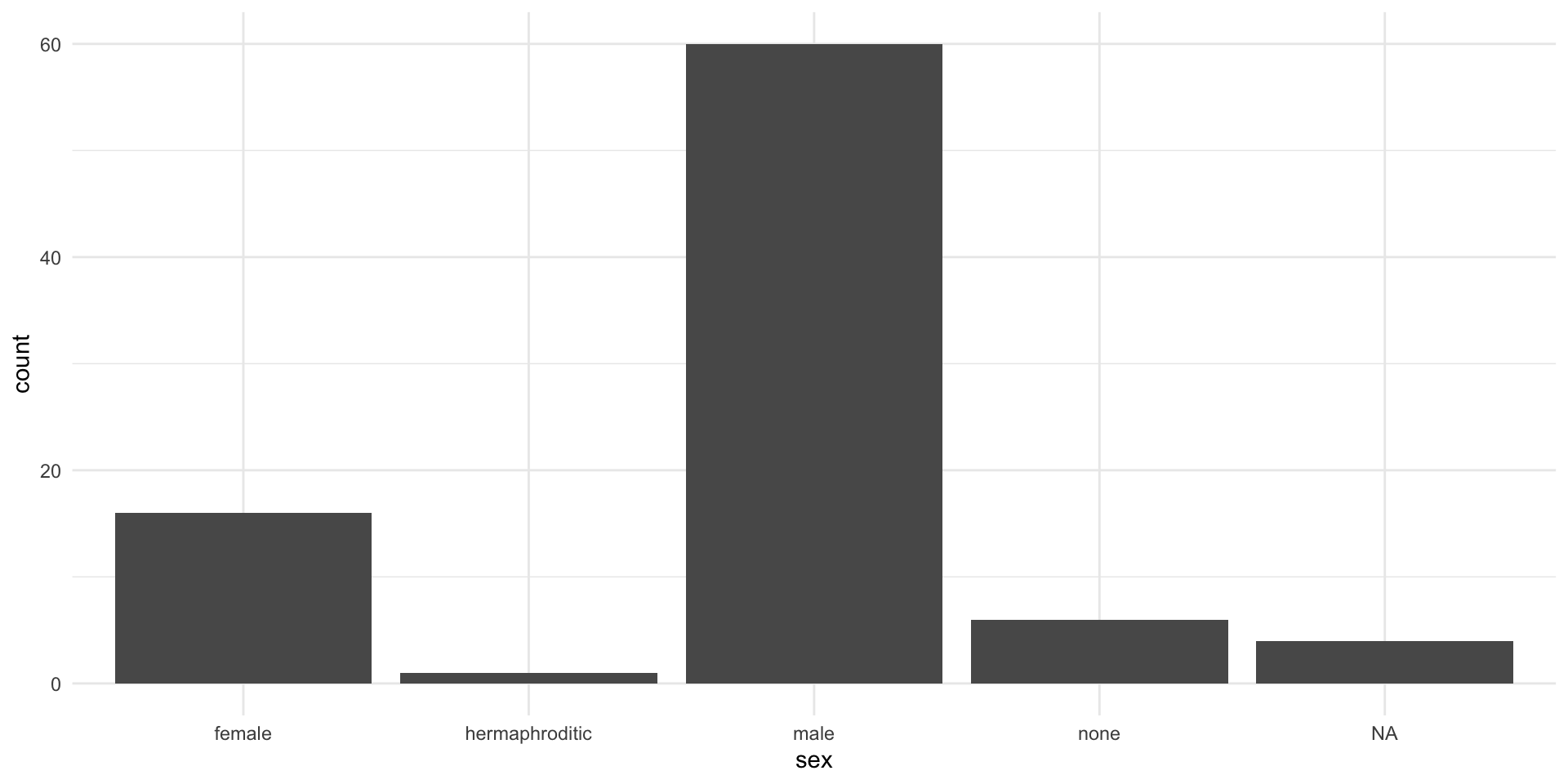
Cualis: barras
Podemos aplicar lo aprendido sobre colores para codificar la información, en este caso vamos a usar las paletas ya cargadas en scale_color_colorblind() del paquete {ggthemes}
Cualis: barras
Fíjate que ahora solo nos ha coloreado el contorno: en otras geometrías, como las barras, será importante distinguir entre color y fill
Cualis: barras
Podemos personalizar el gráfico haciendo uso de las opciones ya vistas, por ejemplo, con escalas en ejes, títulos de las variables, leyendas, etc
starwars |>
drop_na(sex) |>
ggplot(aes(x = sex)) +
geom_bar(aes(fill = sex), alpha = 0.5) +
scale_fill_colorblind() +
scale_y_continuous(breaks = seq(0, 70, by = 10)) +
labs(x = "sexo", y = "frecuencia absoluta", fill = "sexo", title = "Primer diagrama de barras",
subtitle = "Sexos: femenino, masculino, hemafrodita, ninguno y ausente",
caption = "J. Álvarez Liébana") +
theme_minimal()Cualis: barras
Para cambiar el rol de los ejes, generando un diagrama de barras horizontales, podemos dejar el gráfico igual y luego simplemente hacer coord_flip()
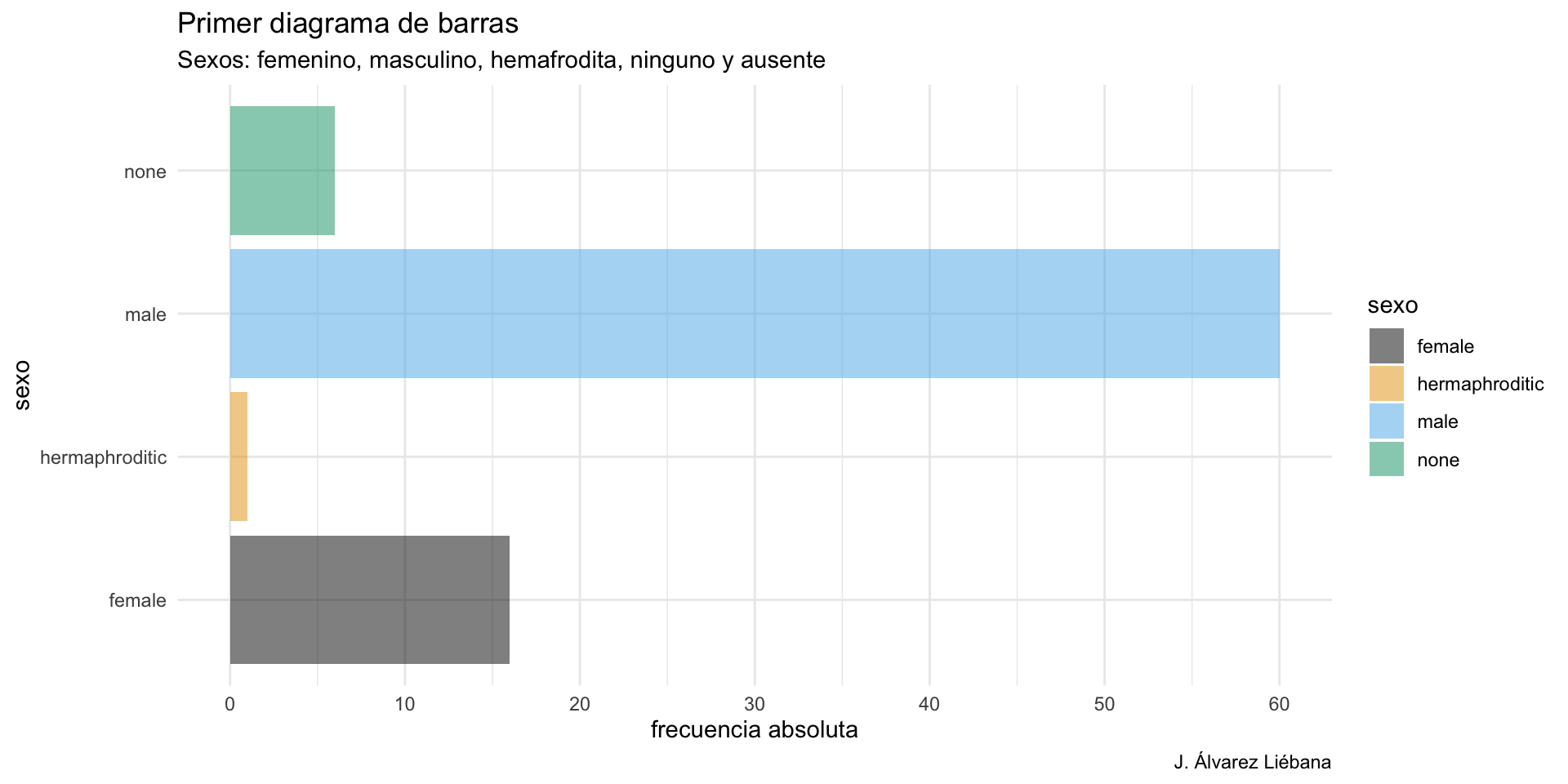
starwars |>
drop_na(sex) |>
ggplot(aes(x = sex)) +
geom_bar(aes(fill = sex), alpha = 0.5) +
scale_fill_colorblind() +
scale_y_continuous(breaks = seq(0, 70, by = 10)) +
coord_flip() +
labs(x = "sexo", y = "frecuencia absoluta", fill = "sexo", title = "Primer diagrama de barras",
subtitle = "Sexos: femenino, masculino, hemafrodita, ninguno y ausente",
caption = "J. Álvarez Liébana") +
theme_minimal()Paréntesis: temas
Vamos a hacer un paréntesis y aprender a personalizar más nuestras gráficas
theme_set(theme_minimal())fija tema basetheme_update(...)personaliza parámetros.
Por ejemplo, en plot.title vamos a indicarle el tamaño y negrita en el título, dentro de element_text()
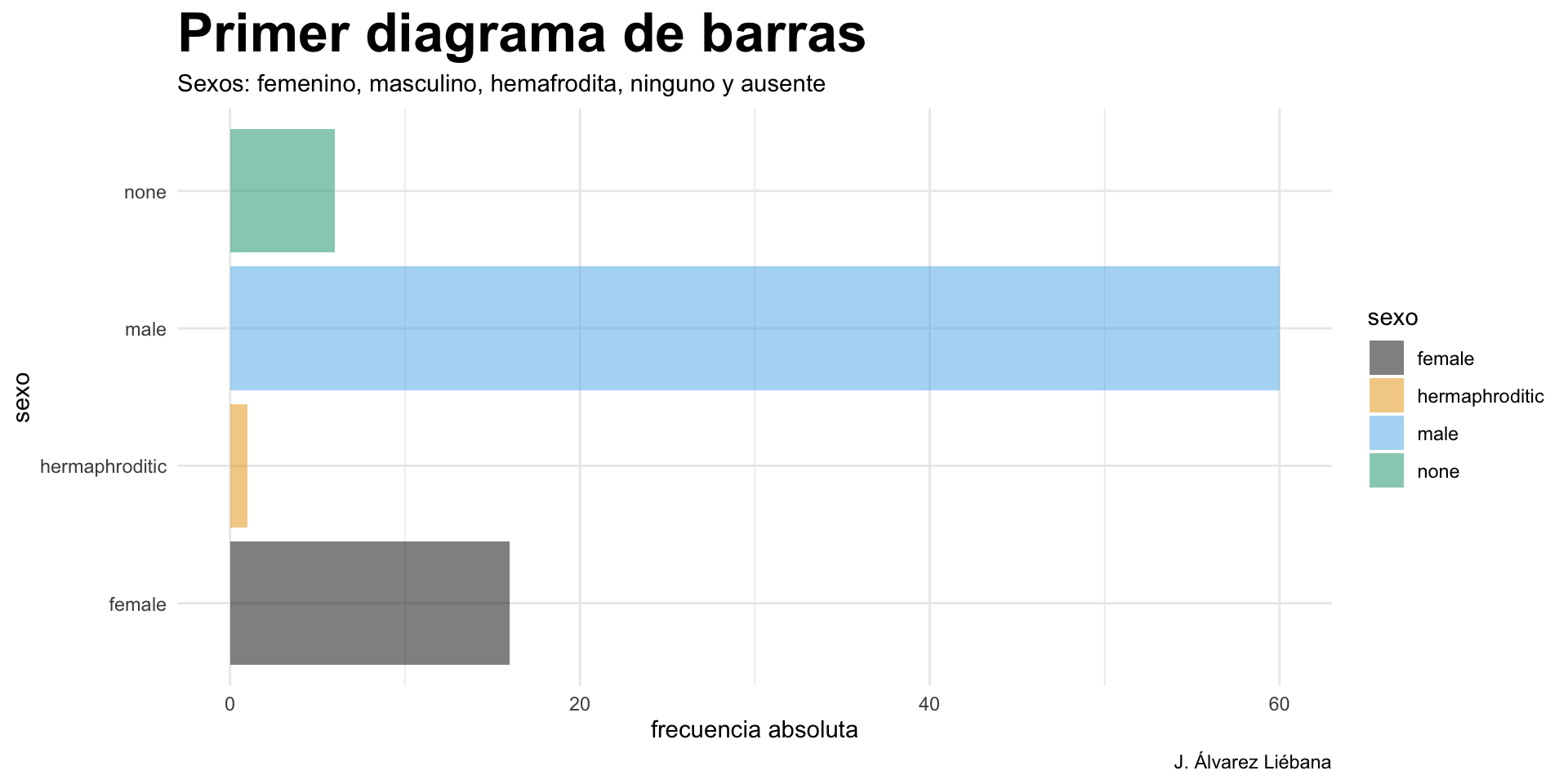
theme_set(theme_minimal())
theme_update(plot.title = element_text(size = 25, face = "bold"))
starwars |>
drop_na(sex) |>
ggplot(aes(x = sex)) +
geom_bar(aes(fill = sex), alpha = 0.5) +
scale_fill_colorblind() +
coord_flip() +
labs(x = "sexo", y = "frecuencia absoluta", fill = "sexo", title = "Primer diagrama de barras",
subtitle = "Sexos: femenino, masculino, hemafrodita, ninguno y ausente", caption = "J. Álvarez Liébana")Paréntesis: temas
Podemos hacer lo mismo con otros textos con plot.subtitle o plot.caption
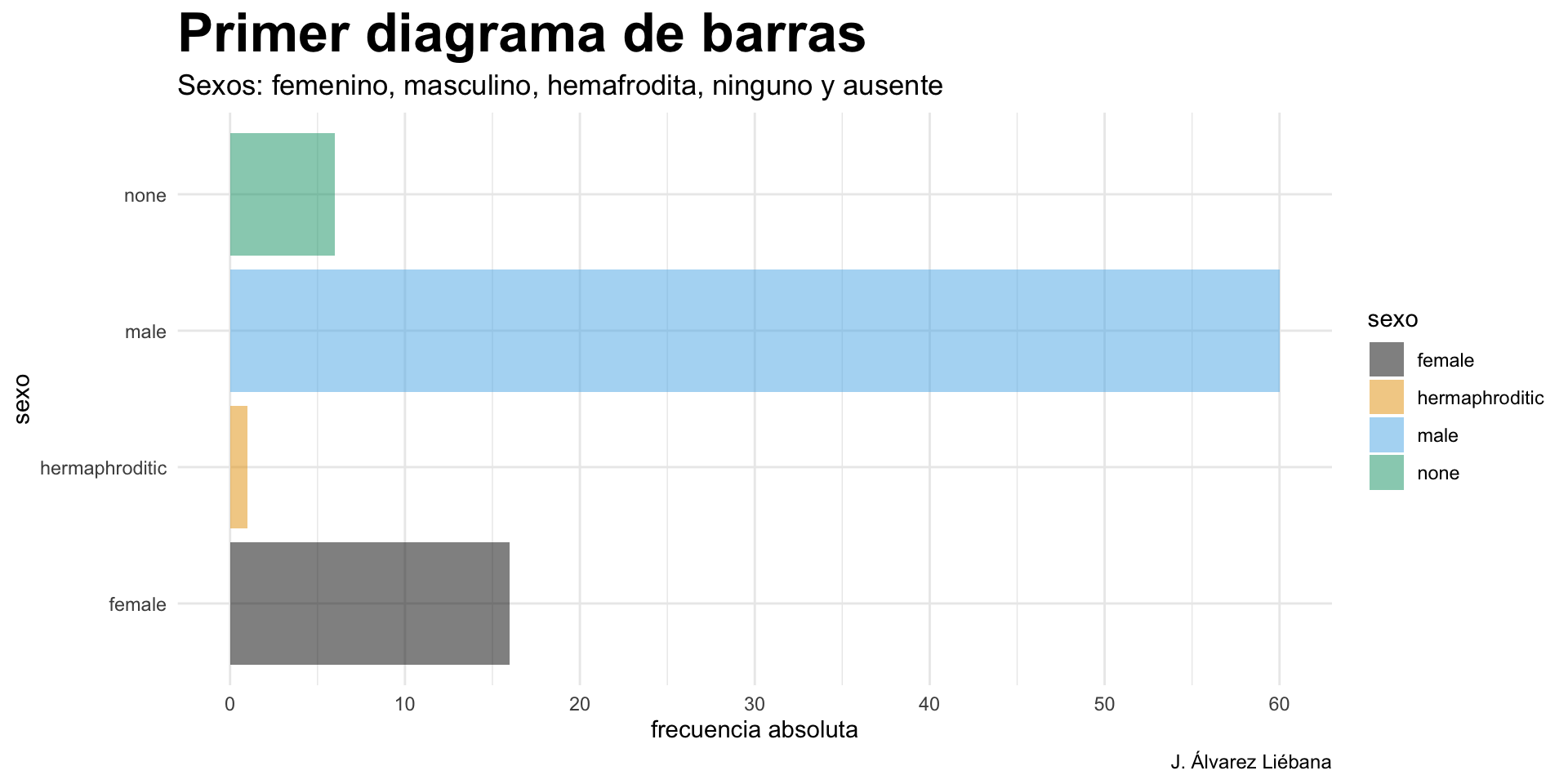
Paréntesis: temas
Vamos incluso a elegir fuente o el color
sysfonts::font_add_google(): le indicaremos la tipografía de https://fonts.google.com/showtext_auto()del paquete{showtext}nos permite su uso.
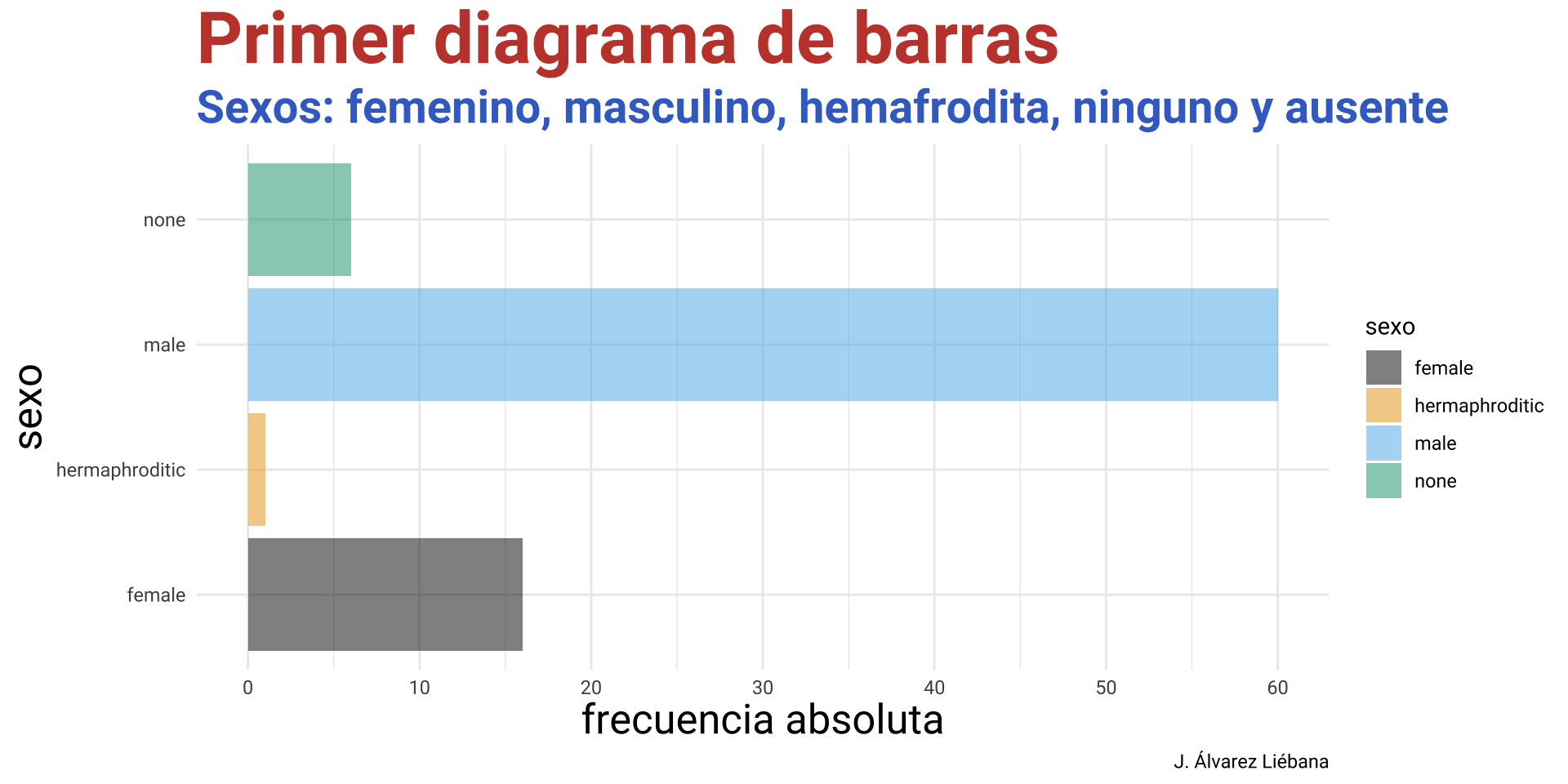
library(showtext)
library(sysfonts)
font_add_google(name = "Roboto")
showtext_auto()
theme_set(theme_minimal(base_family = "Roboto"))
# Configurar tema
theme_update(
plot.title = element_text(color = "#C34539", face = "bold", size = 33),
plot.subtitle = element_text(color = "#3E6FCB", face = "bold", size = 21),
axis.title.x = element_text(size = 19),
axis.title.y = element_text(size = 19))Cualis: barras apiladas
¿Podríamos visualizar dos variables discretas/cualis a la vez?
Podemos incluir una en x = ... y otra en fill = ..., de manera que por defecto nos visualiza barras apiladas, por ejemplo, para ver el reparto de sexos entre humanos y no humanos.
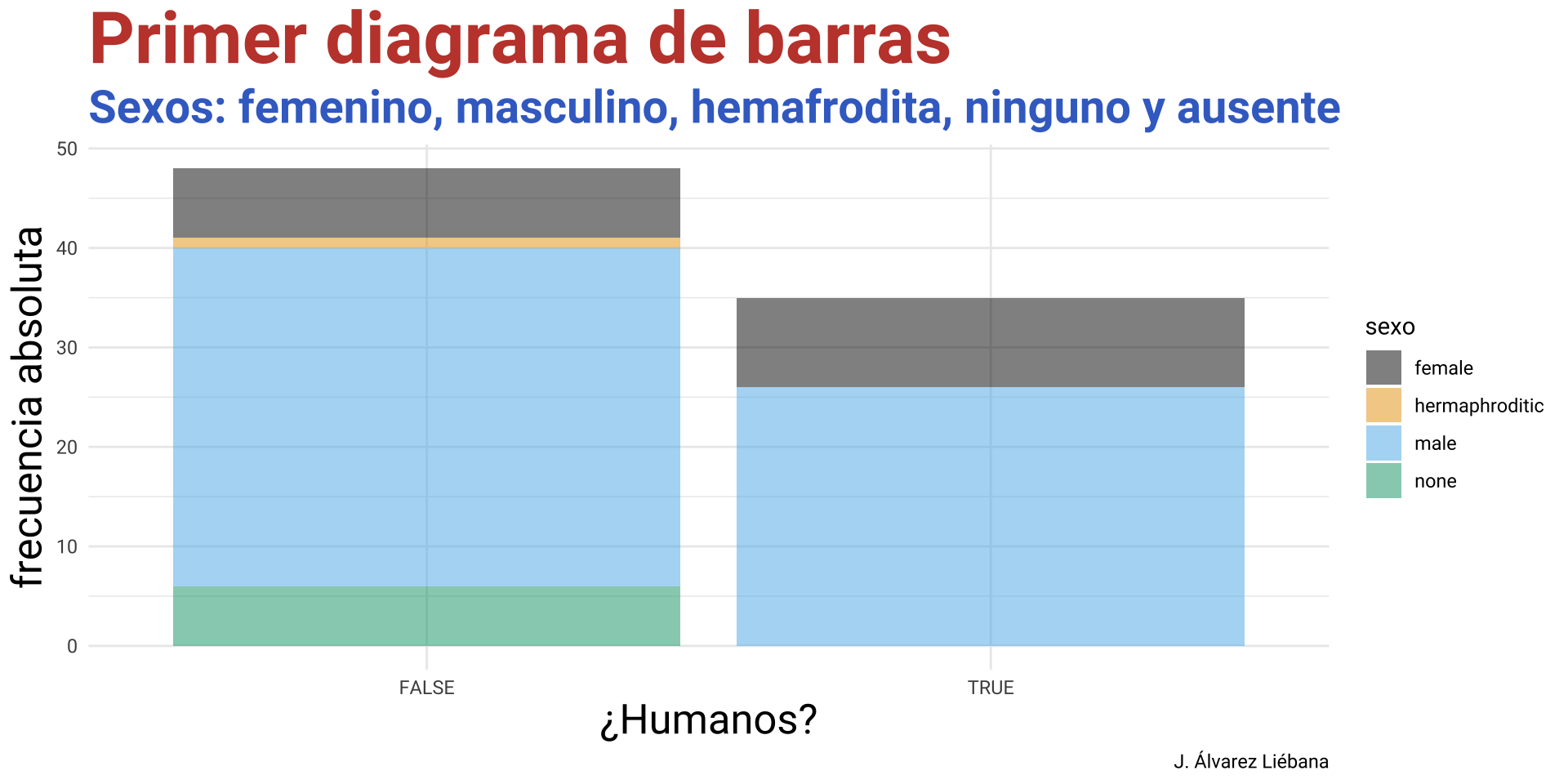
starwars |>
drop_na(sex) |>
mutate(Human = species == "Human") |>
ggplot(aes(x = Human)) +
geom_bar(aes(fill = sex), alpha = 0.5) +
scale_fill_colorblind() +
labs(x = "¿Humanos?", y = "frecuencia absoluta", fill = "sexo", title = "Primer diagrama de barras",
subtitle = "Sexos: femenino, masculino, hemafrodita, ninguno y ausente",
caption = "J. Álvarez Liébana")Cualis: barras solapas
Con position = "dodge" visualizamos las barras sin apilar, solapadas una al lado de otra
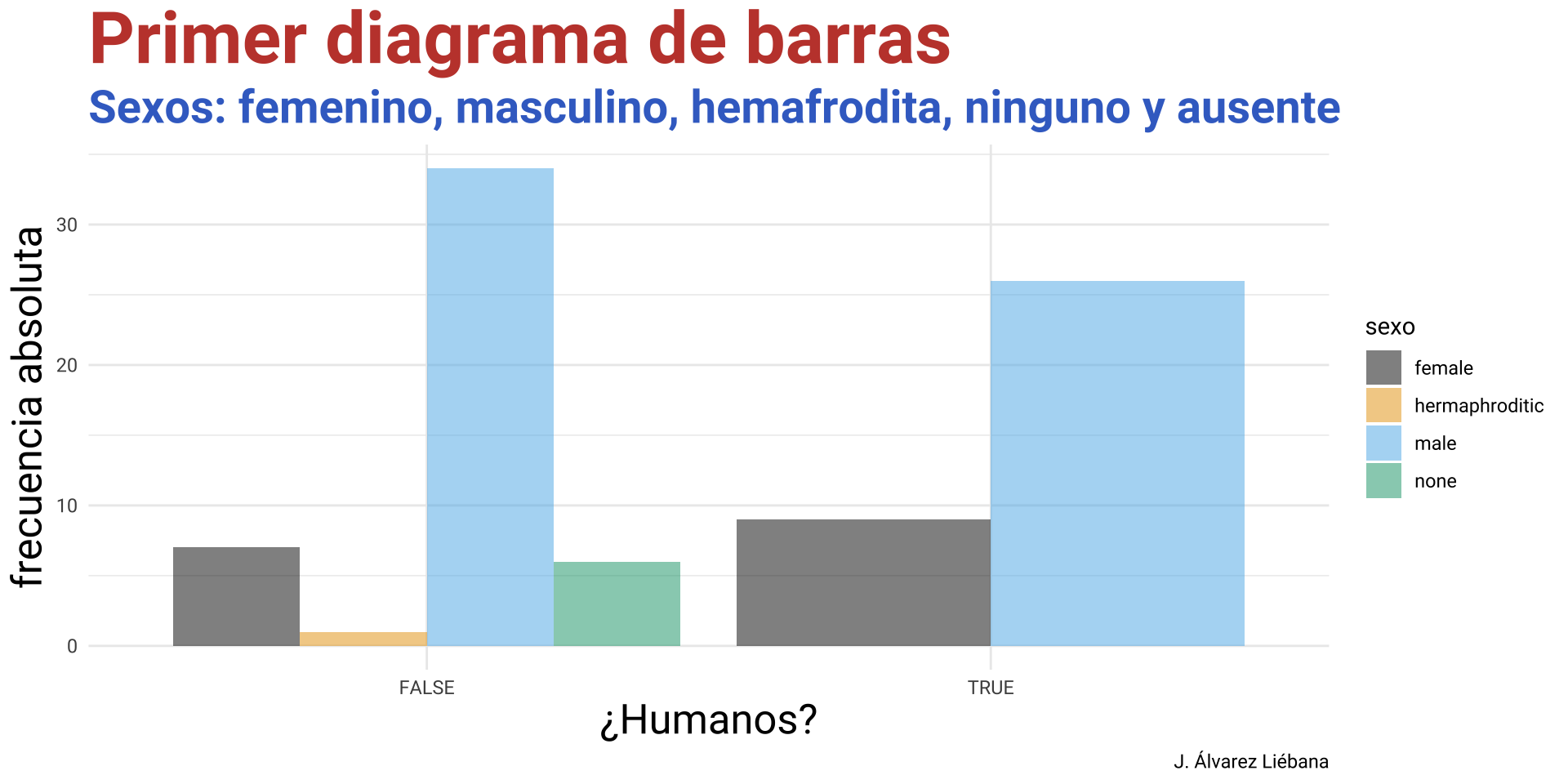
starwars |>
drop_na(sex) |>
mutate(Human = species == "Human") |>
ggplot(aes(x = Human)) +
geom_bar(aes(fill = sex), alpha = 0.5, position = "dodge") +
scale_fill_colorblind() +
labs(x = "¿Humanos?", y = "frecuencia absoluta", fill = "sexo", title = "Primer diagrama de barras",
subtitle = "Sexos: femenino, masculino, hemafrodita, ninguno y ausente",
caption = "J. Álvarez Liébana")Cualis: barras fill
Con position = "fill" visualizamos las barras en forma de frecuencia relativa, con las barras de la misma altura para facilitar la comparativa.
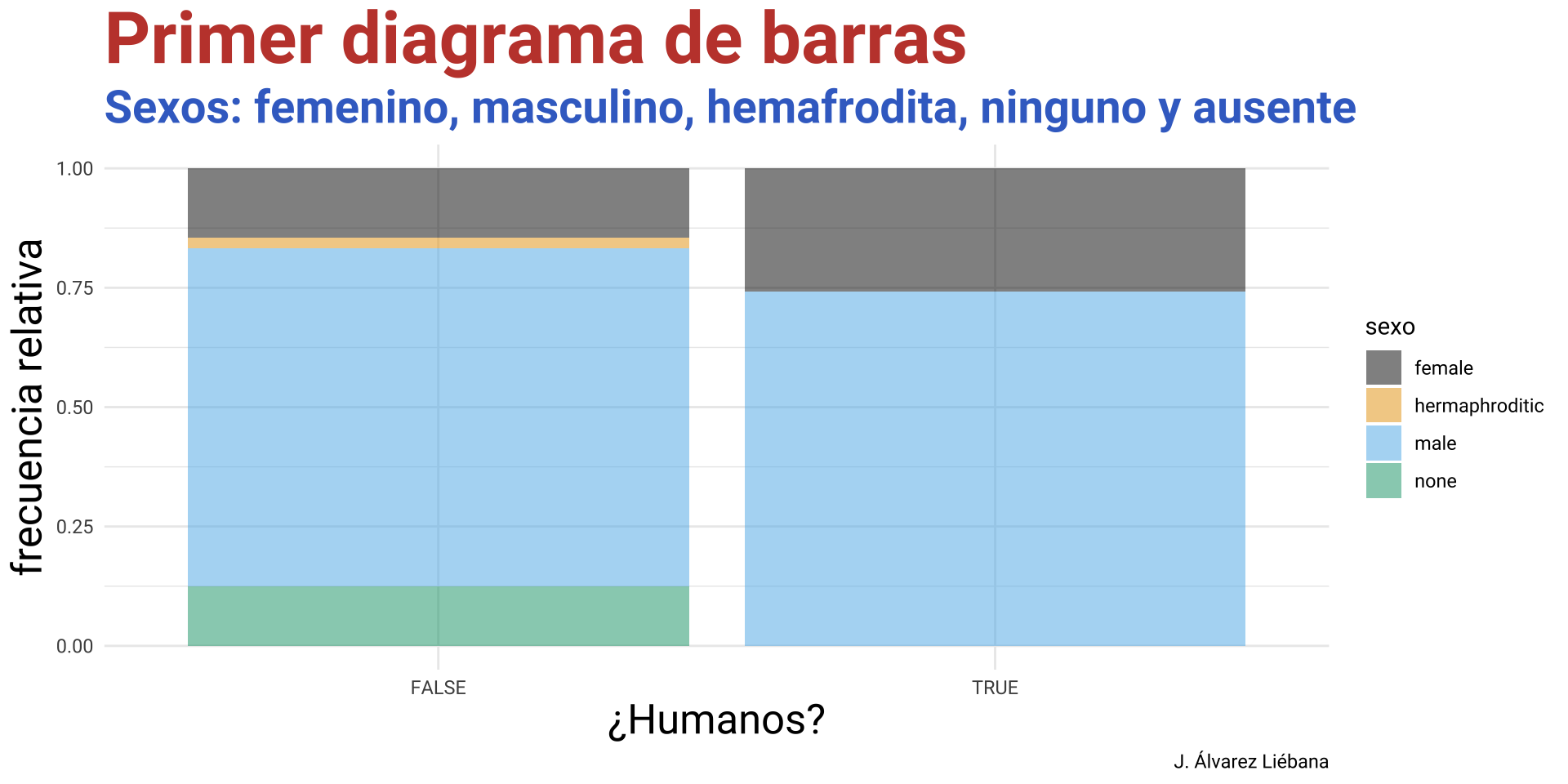
starwars |>
drop_na(sex) |>
mutate(Human = species == "Human") |>
ggplot(aes(x = Human)) +
geom_bar(aes(fill = sex), alpha = 0.5, position = "fill") +
scale_fill_colorblind() +
labs(x = "¿Humanos?", y = "frecuencia relativa", fill = "sexo", title = "Primer diagrama de barras",
subtitle = "Sexos: femenino, masculino, hemafrodita, ninguno y ausente",
caption = "J. Álvarez Liébana")Clase 6: factores
Variables cualitativas
Paréntesis: factores
En el caso de las variables cualitativas, llamaremos niveles o modalidades a los diferentes valores que pueden tomar estos datos. Por ejemplo, en el caso de la variable sex del conjunto starwars, tenemos 4 niveles permitidos: female, hermaphroditic, male y none (amén de datos ausentes).
Paréntesis: factores
Este tipo de variables se conocen en R como factores. Y el paquete fundamental para tratarlos es {forcats} (del entorno {tidyverse}).
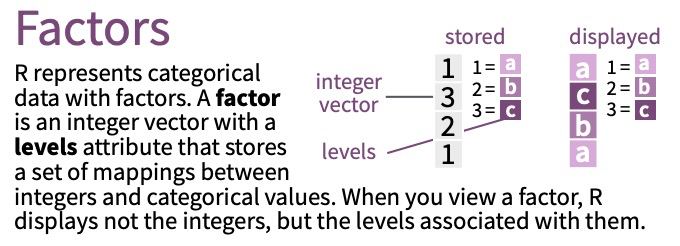
Paréntesis: factores
Este paquete nos permite fijar los niveles (guardados internamente como levels) que toma una determinada variable categórica, dándoles un tratamiento diferente a las cadena de texto normales.
Veamos un ejempo sencillo definiendo una variable estado que tome los valores "sano", "leve" y "grave" de la siguiente manera.
estado <-
c("leve", "grave", "sano", "sano", "leve", "sano", "sano", "grave",
"grave", "leve", "grave", "sano", "sano")
estado [1] "leve" "grave" "sano" "sano" "leve" "sano" "sano" "grave" "grave"
[10] "leve" "grave" "sano" "sano" La variable estado actualmente es de tipo texto, de tipo chr, algo que podemos comprobar con class(estado).
Paréntesis: factores
Desde un punto de vista estadístico y computacional, para R esta variable ahora mismo sería equivalente una variable de nombres. Pero estadísticamente no es lo mismo una variable con nombres (que identifican muchas veces el registro) que una variable categórica como estado que solo puede tomar esos 3 niveles. ¿Cómo convertir a factor?
Haciendo uso de la función as_factor() del paquete {forcats}.
Paréntesis: factores
No solo ha cambiado la clase de la variable sino que ahora, debajo del valor guardado, nos aparece la frase Levels: grave leve sano: son las modalidades o niveles de nuestra cualitativa.
Imagina que ese día en el hospital no tuviésemos a nadie en estado grave: aunque ese día nuestra variable no tome dicho valor, el estado grave es un nivel permitido en la base de datos, así que aunque lo eliminemos, por ser un factor, el nivel permanece (no lo tenemos ahora pero es un nivel permitido).
Paréntesis: factores
Con factor() podemos especificar explícitamente los nombres de las modalidades, incluso si son nominales u ordinales
Paréntesis: factores
Con levels = ... podemos indicarle explícitamente el orden de las modalidades
Paréntesis: factores
Si queremos indicarle que elimine un nivel no usado en ese momento (y que queremos excluir de la definición) podemos hacerlo con fct_drop()
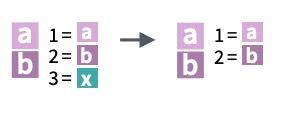
Paréntesis: factores
Al igual que podemos eliminar niveles podemos ampliar los niveles existentes (aunque no existan datos de ese nivel en ese momento) con fct_expand()
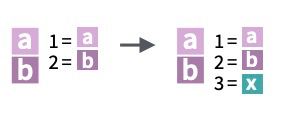
Paréntesis: factores
Además con fct_explicit_na() podemos asignar un nivel a los valores para que sea incluido dicho nivel en los análisis y visualizaciones.
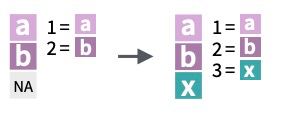
Paréntesis: factores
Incluso una vez definidos podemos reordenar los níveles con fct_relevel()
estado_fct_expand <-
estado_fct |>
mutate(estado = fct_expand(estado, c("UCI", "fallecido"))) |>
pull(estado)
estado_fct_expand |>
fct_relevel(c("fallecido", "leve", "sano", "grave", "UCI")) [1] leve grave sano sano leve sano sano grave grave leve grave sano
[13] sano
Levels: fallecido < leve < sano < grave < UCIParéntesis: factores
Esta forma de trabajar con variables cualitativas nos permite dar una definición teórica de nuestra base de datos, pudiendo incluso contar valores que aún no existen (pero que podrían), haciendo uso de fct_count()
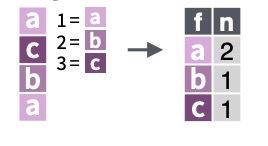
Paréntesis: factores
Los níveles también podemos ordenarlos por frecuencia con fct_infreq()
Paréntesis: factores
A veces querremos agrupar niveles, por ejemplo, no permitiendo niveles que no sucedan un mínimo de veces con fct_lump_min(.., min = ..) (las observaciones que no lo cumplan irán a un nivel genérico llamado Other, aunque se puede cambiar con el argumento other_level).
Paréntesis: factores
Podemos hacer algo equivalente pero en función de su frecuencia relativa con fct_lump_prop().
Paréntesis: factores
Esto lo podemos aplicar a nuestros conjuntos de datos para recategorizar variables de forma muy rápida.
Paréntesis: factores
Con fct_reorder() podemos también indicar que queremos ordenar los factores en función de una función aplicada a otra variable.
[1] Human Droid Droid Human Human Human Human Droid Human Human
[11] Human Human Otras Human Otras Otras Human Otras Human Human
[21] Droid Otras Human Human Otras Human Otras Otras Human Otras
[31] Human Human Gungan Gungan Gungan Human Otras Otras Human Human
[41] Otras Otras Otras Otras Otras Otras Otras Human Otras Otras
[51] Otras Otras Otras Otras Otras Otras Human Otras Otras Otras
[61] Human Human Human Human Otras Otras Otras Otras Human Droid
[71] Otras Otras Otras Otras Otras Human Otras
Levels: Droid Gungan Human Otras [1] Human Droid Droid Human Human Human Human Droid Human Human
[11] Human Human Otras Human Otras Otras Human Otras Human Human
[21] Droid Otras Human Human Otras Human Otras Otras Human Otras
[31] Human Human Gungan Gungan Gungan Human Otras Otras Human Human
[41] Otras Otras Otras Otras Otras Otras Otras Human Otras Otras
[51] Otras Otras Otras Otras Otras Otras Human Otras Otras Otras
[61] Human Human Human Human Otras Otras Otras Otras Human Droid
[71] Otras Otras Otras Otras Otras Human Otras
Levels: Droid Otras Human Gungan💻 Tu turno
Intenta realizar los siguientes ejercicios sin mirar las soluciones
📝 Dada la variable meses definida debajo (definida como un vector de caracteres), convierte dicha variable a factor (solo eso)
📝 Dada la variable meses definida debajo convierte dicha variable a factor pero indicando los niveles de forma correcta.
Código
meses <- c(NA, "Abr", "Ene", "Oct", "Jul", "Ene", "Sep", NA, "Feb", "Dic",
"Jul", "Mar", "Ene", "Mar", "Feb", "Abr", "May", "Oct", "Sep", NA,
"Dic", "Jul", "Nov", "Feb", "Oct", "Jun", "Sep", "Oct", "Oct", "Sep")
# Orden de niveles correcto e incluimos agosto aunque no haya
meses_fct <-
factor(meses,
levels = c("Ene", "Feb", "Mar", "Abr", "May", "Jun", "Jul", "Ago", "Sep", "Oct", "Nov", "Dic"))
meses_fct📝 Cuenta cuantos valores hay de cada mes pero teniendo en cuenta que son factores (quizás haya niveles sin ser usados y de los que debería obtener un 0).
📝 Dado que hay ausentes, indica que los ausentes sea un decimotercer nivel etiquetado como “ausente”.
Cualis: barras ordenadas
Haciendo uso de los que sabemos sobre factores podemos indicarle que nos ordene las columnas de manera personalizada definiendo la variable cuali como un factor.
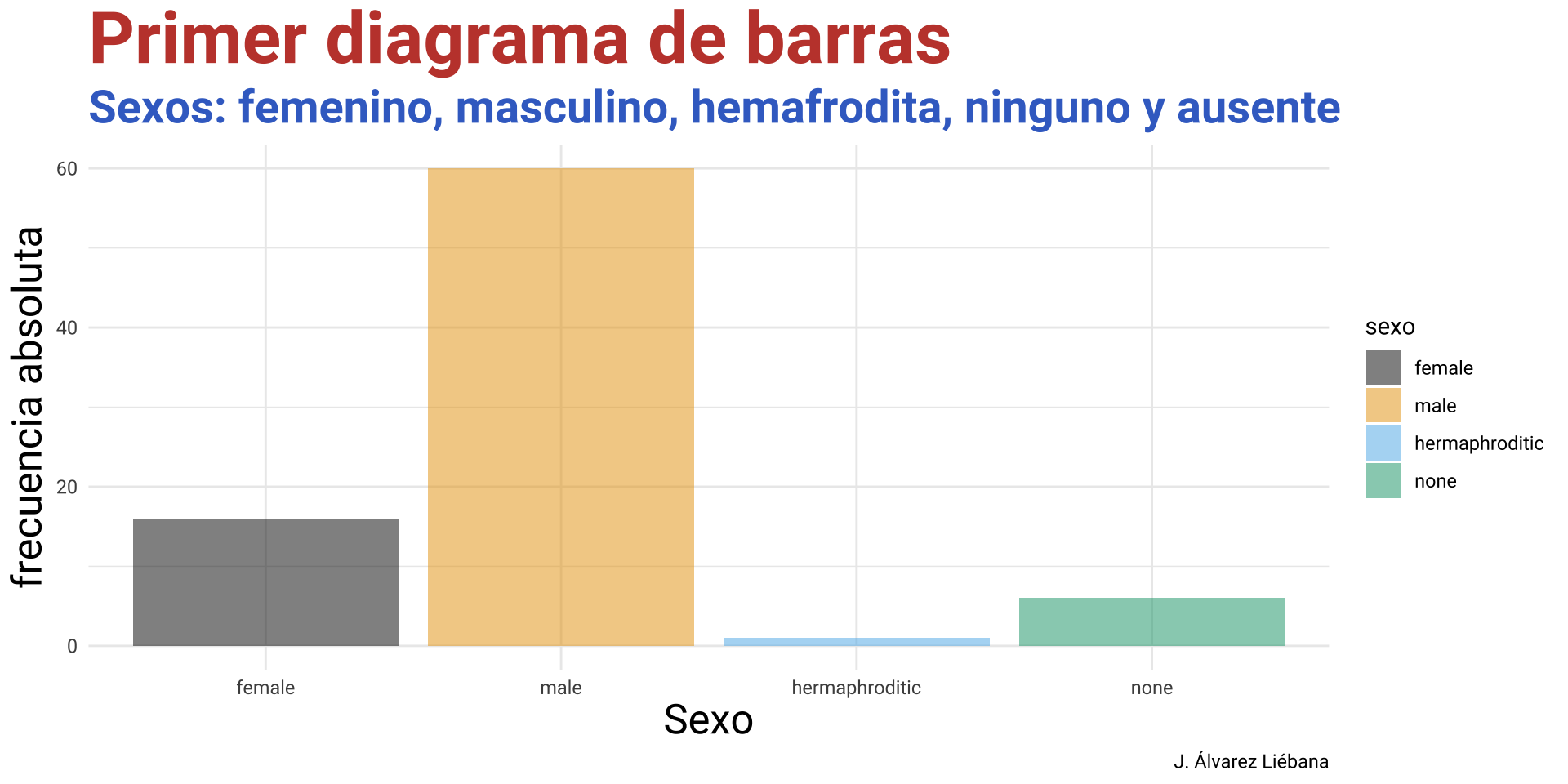
starwars |>
drop_na(sex) |>
mutate(sex = factor(sex, levels = c("female", "male", "hermaphroditic", "none"))) |>
ggplot(aes(x = sex)) +
geom_bar(aes(fill = sex), alpha = 0.5) +
scale_fill_colorblind() +
labs(x = "Sexo", y = "frecuencia absoluta", fill = "sexo", title = "Primer diagrama de barras",
subtitle = "Sexos: femenino, masculino, hemafrodita, ninguno y ausente",
caption = "J. Álvarez Liébana")Cualis: barras ordenadas
También podemos indicarle que nos ordene las columnas de mayor a menor frecuencia usando simplemente fct_infreq()
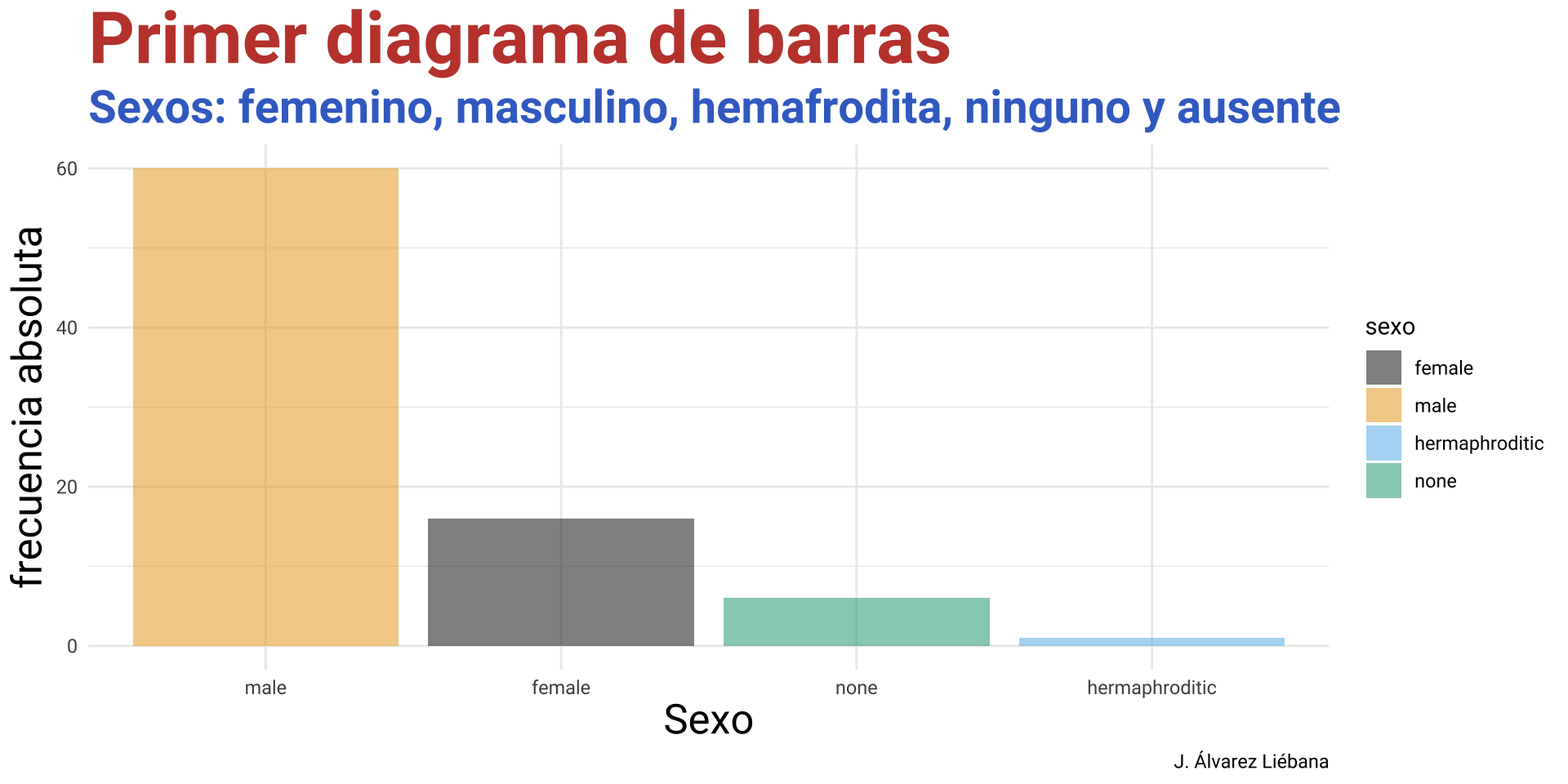
starwars |>
drop_na(sex) |>
mutate(sex = factor(sex, levels = c("female", "male", "hermaphroditic", "none"))) |>
ggplot(aes(x = fct_infreq(sex))) +
geom_bar(aes(fill = sex), alpha = 0.5) +
scale_fill_colorblind() +
labs(x = "Sexo", y = "frecuencia absoluta", fill = "sexo", title = "Primer diagrama de barras",
subtitle = "Sexos: femenino, masculino, hemafrodita, ninguno y ausente",
caption = "J. Álvarez Liébana")Cualis: barras ordenadas
Para invertir el orden de los factores basta usar fct_rev()
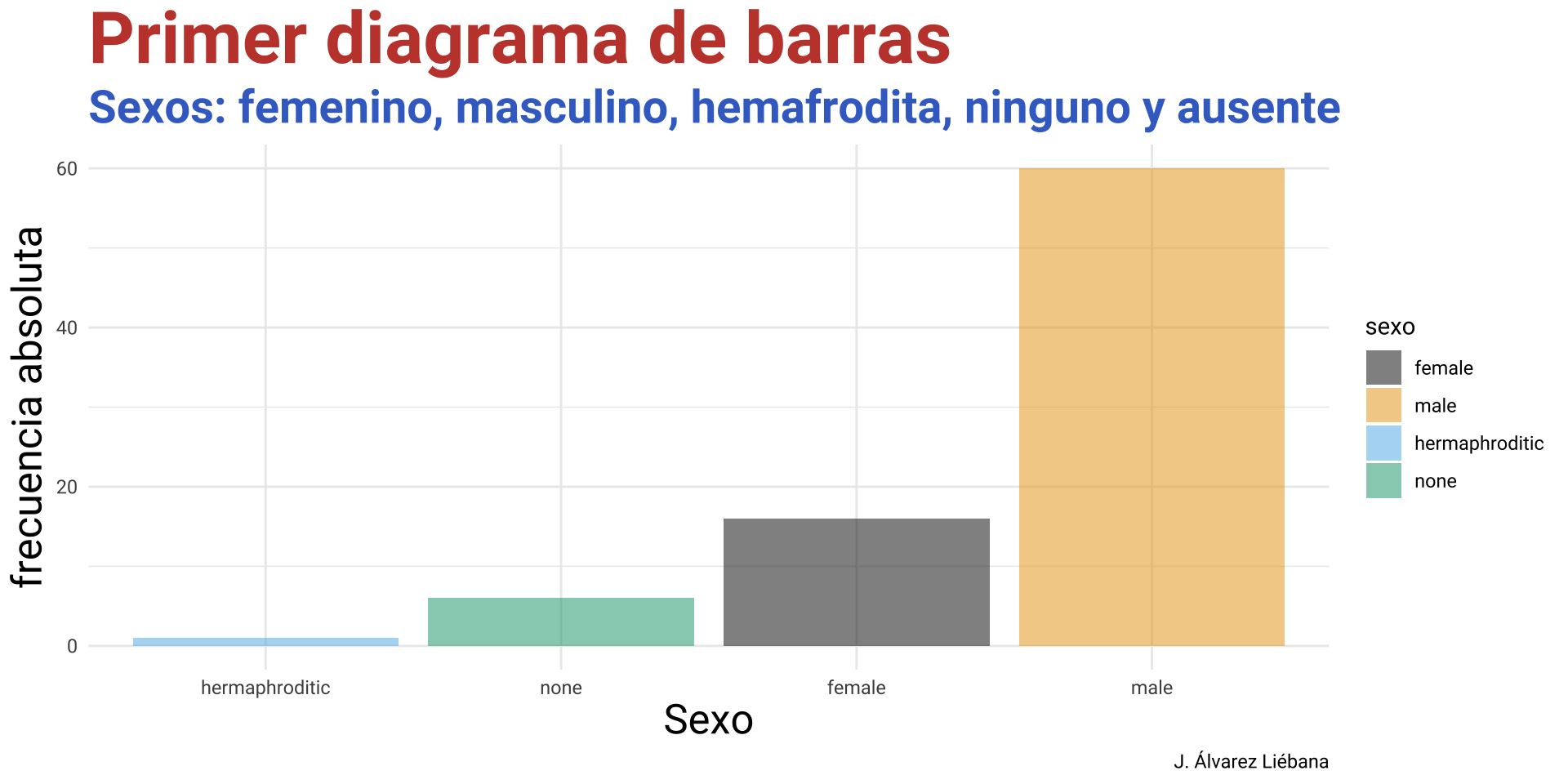
starwars |>
drop_na(sex) |>
mutate(sex = factor(sex, levels = c("female", "male", "hermaphroditic", "none"))) |>
ggplot(aes(x = fct_rev(fct_infreq(sex)))) +
geom_bar(aes(fill = sex), alpha = 0.5) +
scale_fill_colorblind() +
labs(x = "Sexo", y = "frecuencia absoluta", fill = "sexo", title = "Primer diagrama de barras",
subtitle = "Sexos: femenino, masculino, hemafrodita, ninguno y ausente",
caption = "J. Álvarez Liébana")Cualis: geom_col()
La capa geom_bar() está solo pensada para conteos de variables discretas o cualitativas. ¿Y si queremos visualizar en el peso por sexo?
Usaremos geom_col() (ahora si necesitamos x,y)
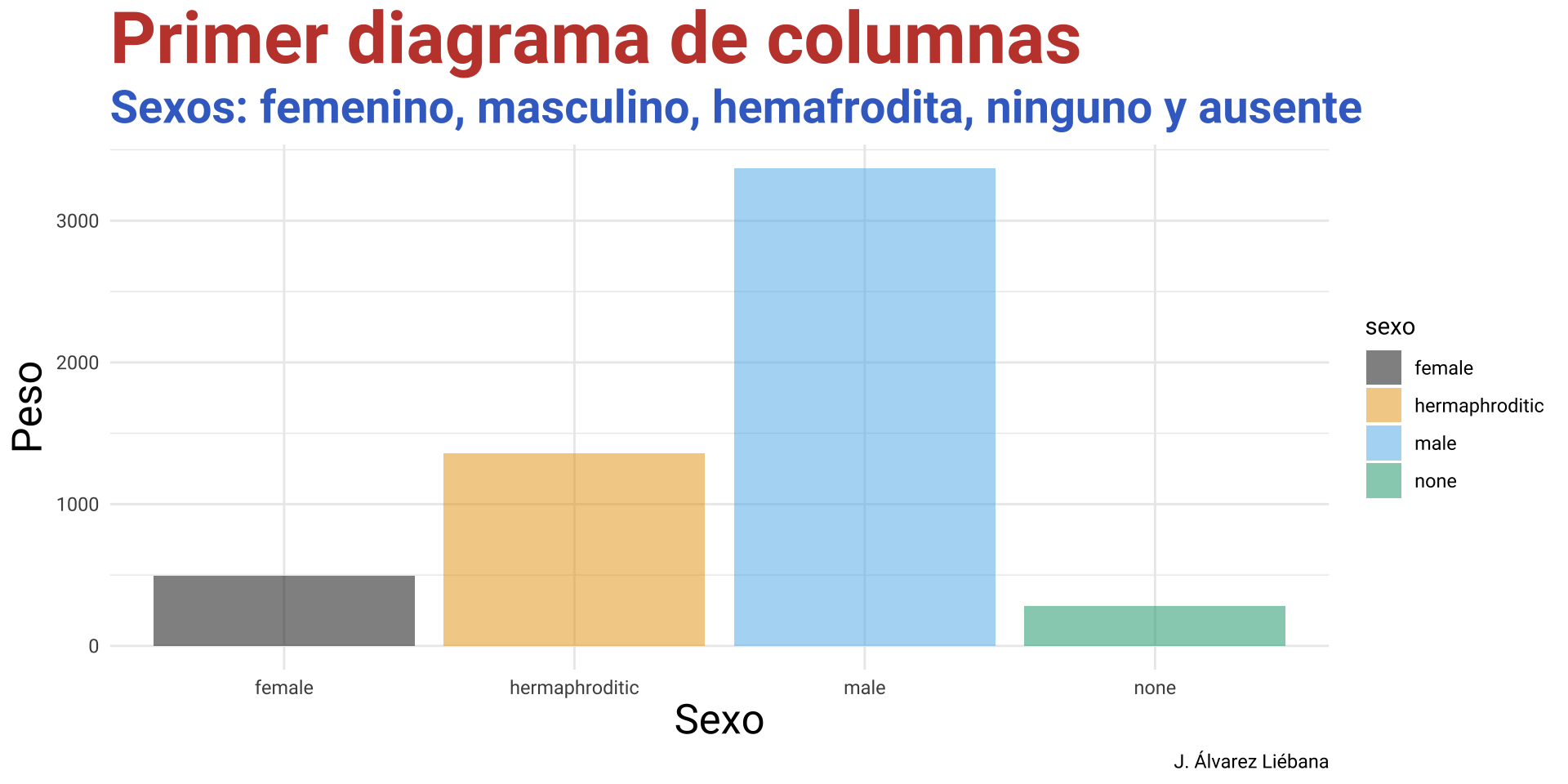
starwars |>
drop_na(mass, sex) |>
ggplot(aes(x = sex, y = mass)) +
geom_col(aes(fill = sex), alpha = 0.5) +
scale_fill_colorblind() +
labs(x = "Sexo", y = "Peso", fill = "sexo", title = "Primer diagrama de columnas",
subtitle = "Sexos: femenino, masculino, hemafrodita, ninguno y ausente", caption = "J. Álvarez Liébana")Cualis: geom_col()
Fíjate que por defecto lo que hace es sumar la variable continua. ¿Cómo pedir que visualice, por ejemplo, la media por grupos?
La forma más inmediata es hacer un geom_col() pero en lugar de a la tabla original a un resumen de la misma.
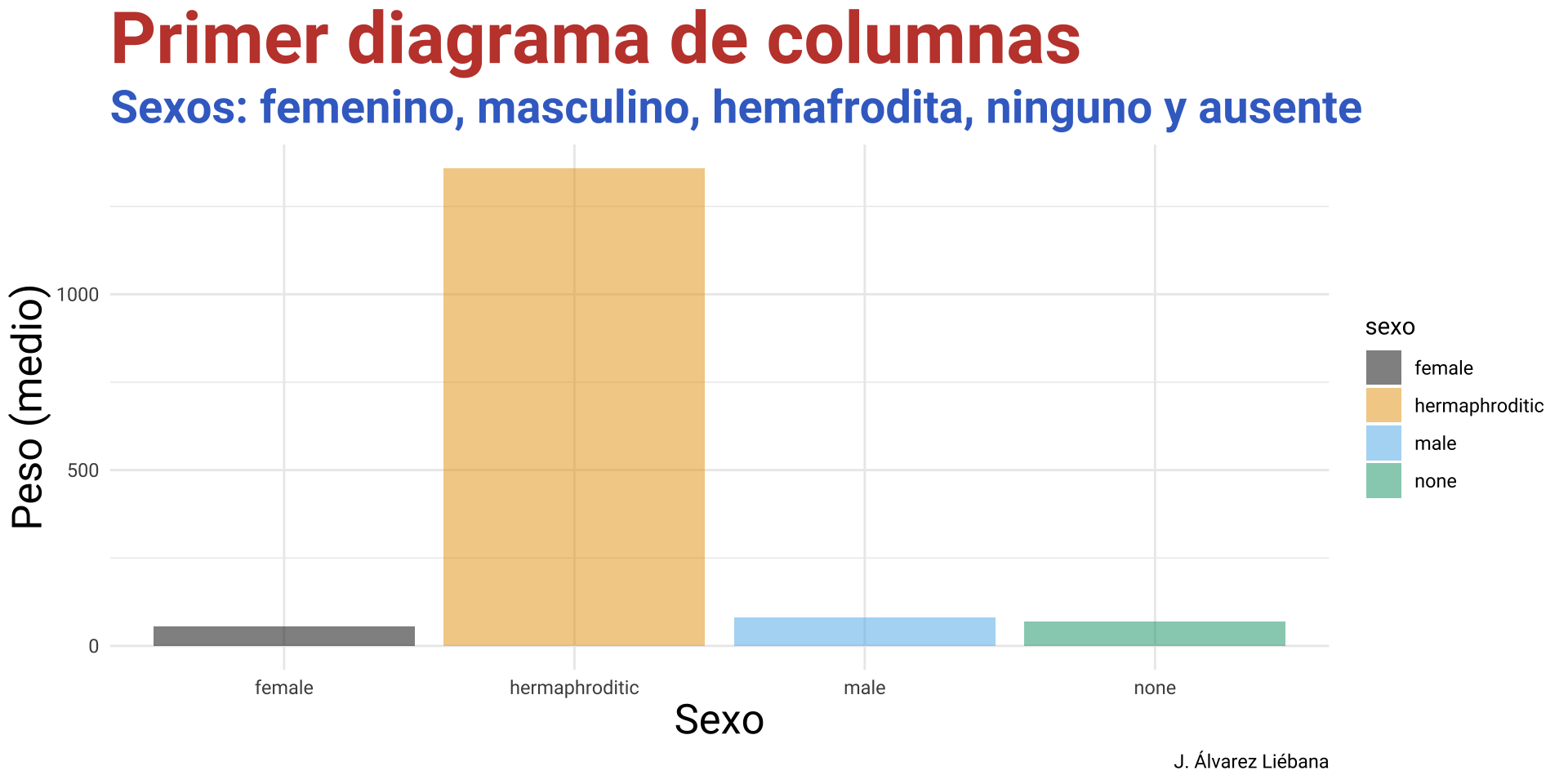
starwars |>
drop_na(mass, sex) |>
summarise(mean_mass = mean(mass), .by = sex) |>
ggplot(aes(x = sex, y = mean_mass)) +
geom_col(aes(fill = sex), alpha = 0.5) +
scale_fill_colorblind() +
labs(x = "Sexo", y = "Peso (medio)", fill = "sexo", title = "Primer diagrama de columnas",
subtitle = "Sexos: femenino, masculino, hemafrodita, ninguno y ausente", caption = "J. Álvarez Liébana")Cualis: geom_col()
Otra opción es no usar la capa geométrica sino la capa estadística, con stat_summary() e indicándole la función a visualizar y el geometría
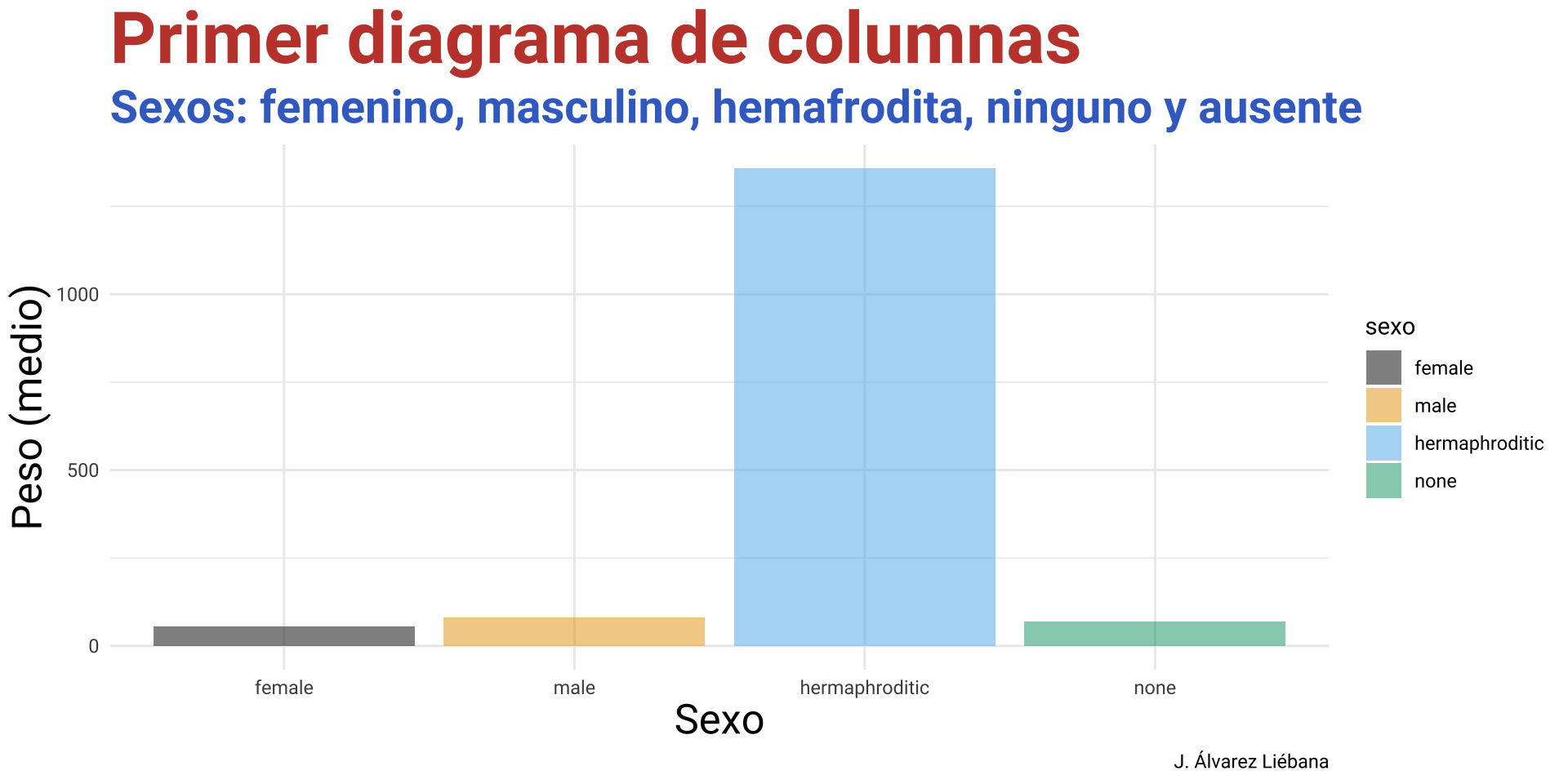
starwars |>
drop_na(mass, sex) |>
mutate(sex = factor(sex, levels = c("female", "male", "hermaphroditic", "none"))) |>
ggplot(aes(x = sex, y = mass, fill = sex)) +
stat_summary(geom = "col", fun = mean, alpha = 0.5) +
scale_fill_colorblind() +
labs(x = "Sexo", y = "Peso (medio)", fill = "sexo",
title = "Primer diagrama de columnas",
subtitle = "Sexos: femenino, masculino, hemafrodita, ninguno y ausente",
caption = "J. Álvarez Liébana")Cualis: geom_col()
Fíjate que ambas formas nos permiten visualizar cualquier otro estadístico, por ejempo, la mediana
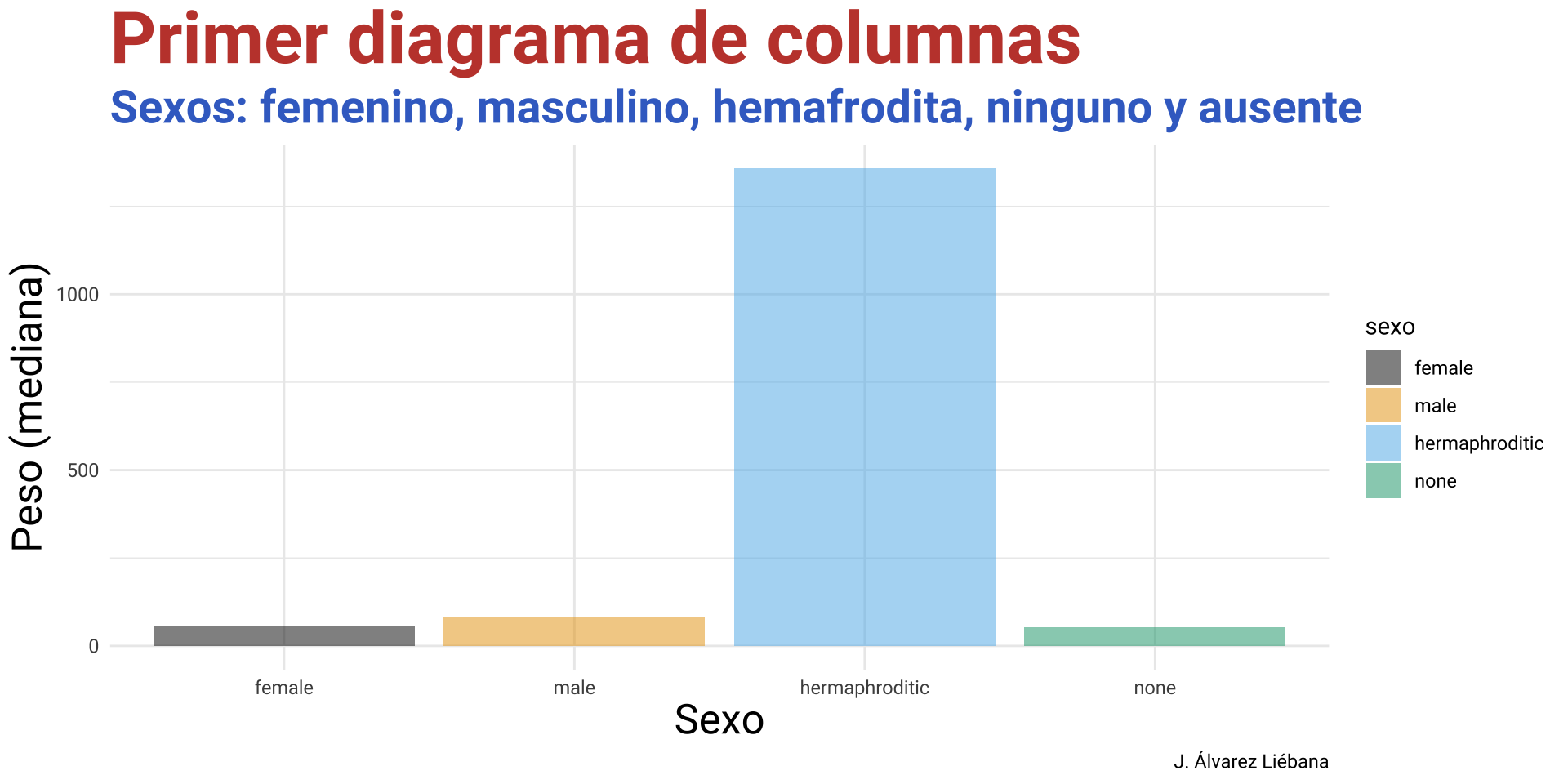
starwars |>
drop_na(mass, sex) |>
mutate(sex = factor(sex, levels = c("female", "male", "hermaphroditic", "none"))) |>
ggplot(aes(x = sex, y = mass, fill = sex)) +
stat_summary(geom = "col", fun = median, alpha = 0.5) +
scale_fill_colorblind() +
labs(x = "Sexo", y = "Peso (mediana)", fill = "sexo",
title = "Primer diagrama de columnas",
subtitle = "Sexos: femenino, masculino, hemafrodita, ninguno y ausente",
caption = "J. Álvarez Liébana")Cualis: geom_col()
La última opción es volver a nuestra conocida geom_bar(), indicándole stat = "summary", fun = "mean", por ejemplo (por defecto stat = "count") con ahora sí dos variables
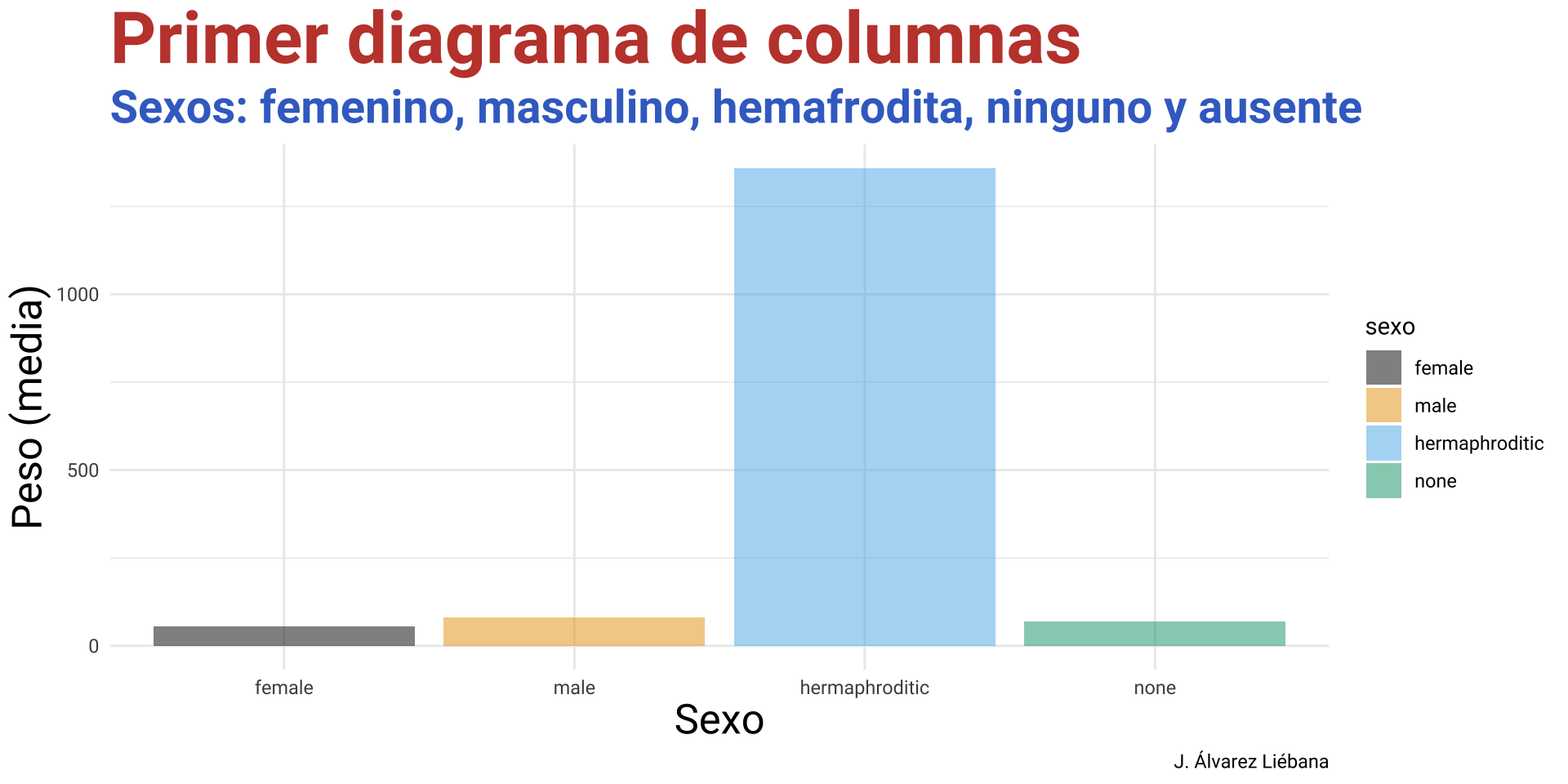
starwars |>
drop_na(mass, sex) |>
mutate(sex = factor(sex, levels = c("female", "male", "hermaphroditic", "none"))) |>
ggplot(aes(x = sex, y = mass)) +
geom_bar(aes(fill = sex), alpha = 0.5,
stat = "summary", fun = "mean") +
scale_fill_colorblind() +
labs(x = "Sexo", y = "Peso (media)", fill = "sexo",
title = "Primer diagrama de columnas",
subtitle = "Sexos: femenino, masculino, hemafrodita, ninguno y ausente",
caption = "J. Álvarez Liébana")🐣 Caso práctico: ggplot
Usa el dataset gapminder y visualiza en un gráfico la media de la variable gdpPercap por continente y año mediante un diagrama de barras en 3 formas:
- barras solapadas
- barras apiladas
- barras fill
🐣 Caso práctico: ggplot
El objetivo es analizar un conjunto de datos que contiene las respuestas a las pregunta «¿Qué probabilidad (%) asignarías al término (entre otros) …
- «almost no chance»
- «probable»
- «almost certainly»
…con el objetivo de comprender cómo la gente percibe el vocabulario de la probabilidad.
datos <-
read_csv("https://raw.githubusercontent.com/zonination/perceptions/master/probly.csv")
datos# A tibble: 46 × 17
`Almost Certainly` `Highly Likely` `Very Good Chance` Probable Likely
<dbl> <dbl> <dbl> <dbl> <dbl>
1 95 80 85 75 66
2 95 75 75 51 75
3 95 85 85 70 75
4 95 85 85 70 75
5 98 95 80 70 70
6 95 99 85 90 75
7 85 95 65 80 40
8 97 95 75 70 70
9 95 95 80 70 65
10 90 85 90 70 75
# ℹ 36 more rows
# ℹ 12 more variables: Probably <dbl>, `We Believe` <dbl>,
# `Better Than Even` <dbl>, `About Even` <dbl>, `We Doubt` <dbl>,
# Improbable <dbl>, Unlikely <dbl>, `Probably Not` <dbl>,
# `Little Chance` <dbl>, `Almost No Chance` <dbl>, `Highly Unlikely` <dbl>,
# `Chances Are Slight` <dbl>🐣 Caso práctico: ggplot
datos <-
read_csv("https://raw.githubusercontent.com/zonination/perceptions/master/probly.csv")
datos# A tibble: 46 × 17
`Almost Certainly` `Highly Likely` `Very Good Chance` Probable Likely
<dbl> <dbl> <dbl> <dbl> <dbl>
1 95 80 85 75 66
2 95 75 75 51 75
3 95 85 85 70 75
4 95 85 85 70 75
5 98 95 80 70 70
6 95 99 85 90 75
7 85 95 65 80 40
8 97 95 75 70 70
9 95 95 80 70 65
10 90 85 90 70 75
# ℹ 36 more rows
# ℹ 12 more variables: Probably <dbl>, `We Believe` <dbl>,
# `Better Than Even` <dbl>, `About Even` <dbl>, `We Doubt` <dbl>,
# Improbable <dbl>, Unlikely <dbl>, `Probably Not` <dbl>,
# `Little Chance` <dbl>, `Almost No Chance` <dbl>, `Highly Unlikely` <dbl>,
# `Chances Are Slight` <dbl>Solo haciendo uso de los gráficos aprendidos hasta ahora, ¿cómo visualizarías dicho dataset? Realiza las tranformaciones que consideres para una correcta preparación de los datos.
Clase 6: variables continuas
Variables continuas
Variables continuas
Solo haciendo uso de los gráficos aprendidos hasta ahora, ¿cómo visualizarías dicho dataset? Realiza las tranformaciones que consideres para una correcta preparación de los datos.
Lo primero que deberemos hacer es preparar nuestros datos para la posterior visualización en formato tidy
datos_tidy <-
datos |>
pivot_longer(cols = everything(),
names_to = "termino", values_to = "prob")
datos_tidy# A tibble: 782 × 2
termino prob
<chr> <dbl>
1 Almost Certainly 95
2 Highly Likely 80
3 Very Good Chance 85
4 Probable 75
5 Likely 66
6 Probably 75
7 We Believe 66
8 Better Than Even 55
9 About Even 50
10 We Doubt 40
# ℹ 772 more rowsVariables continuas
¿Cómo podemos visualizar estos datos?
Tenemos dos variables:
termino: cualitativa ordinalprob: cuantitativa continua
Por lo que de momento no tenemos herramienta para visualizarlo ya que
- scatter plot: dos variables continuas.
- diagrama de barras: variables discretas o cualitativas
La única manera será realizar un resumen de los datos visualizando, por ejemplo, la media de probabilidad asignada
Variables continuas
La única manera será realizar un resumen de los datos visualizando, por ejemplo, la media de probabilidad asignada
# A tibble: 17 × 2
termino mean_prob
<chr> <dbl>
1 Almost Certainly 92.6
2 Highly Likely 86.2
3 Very Good Chance 79.8
4 Probable 71.5
5 Likely 72
6 Probably 71.5
7 We Believe 68.5
8 Better Than Even 58.4
9 About Even 49.6
10 We Doubt 27.9
11 Improbable 18.0
12 Unlikely 19.9
13 Probably Not 29.5
14 Little Chance 16.0
15 Almost No Chance 5.63
16 Highly Unlikely 10.1
17 Chances Are Slight 14.1 Variables continuas
Con scale_fill_gradient2() vamos a crear un gradiente de color, y con scale_y_continuous() incorporamos % en el eje Y.
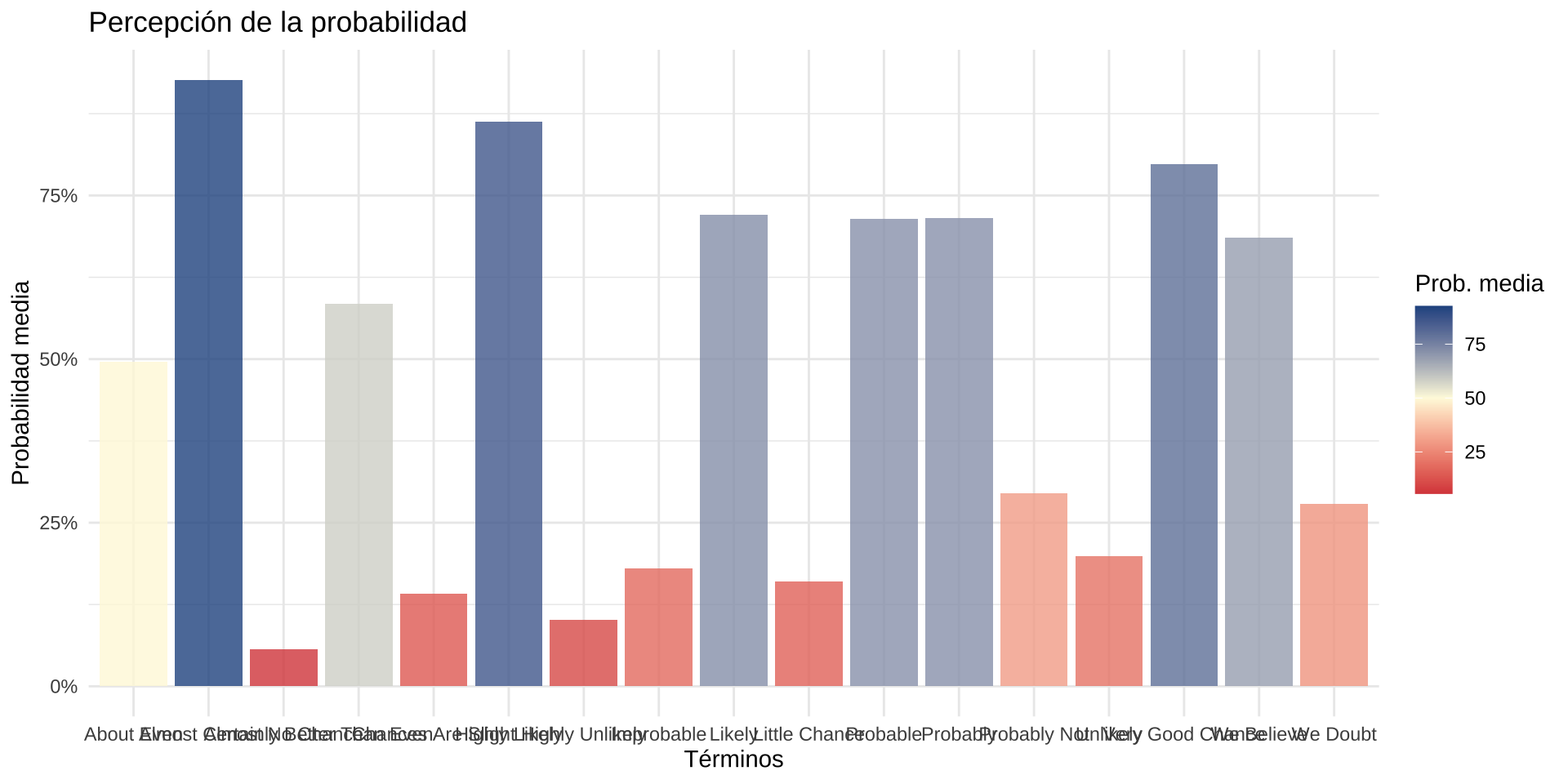
ggplot(resumen, aes(x = termino, y = mean_prob, fill = mean_prob)) +
geom_col(alpha = 0.8) +
scale_fill_gradient2(low = "#DA4A4A", mid = "#FEFADF", high = "#144F8D", midpoint = 50) +
scale_y_continuous(labels = scales::label_number(suffix = "%")) +
labs(fill = "Prob. media", x = "Términos", y = "Probabilidad media",
title = "Percepción de la probabilidad") +
theme_minimal()Variables continuas
Para mejorar la legibilidad vamos a reducir el tamaño de las etiquetas del eje X.
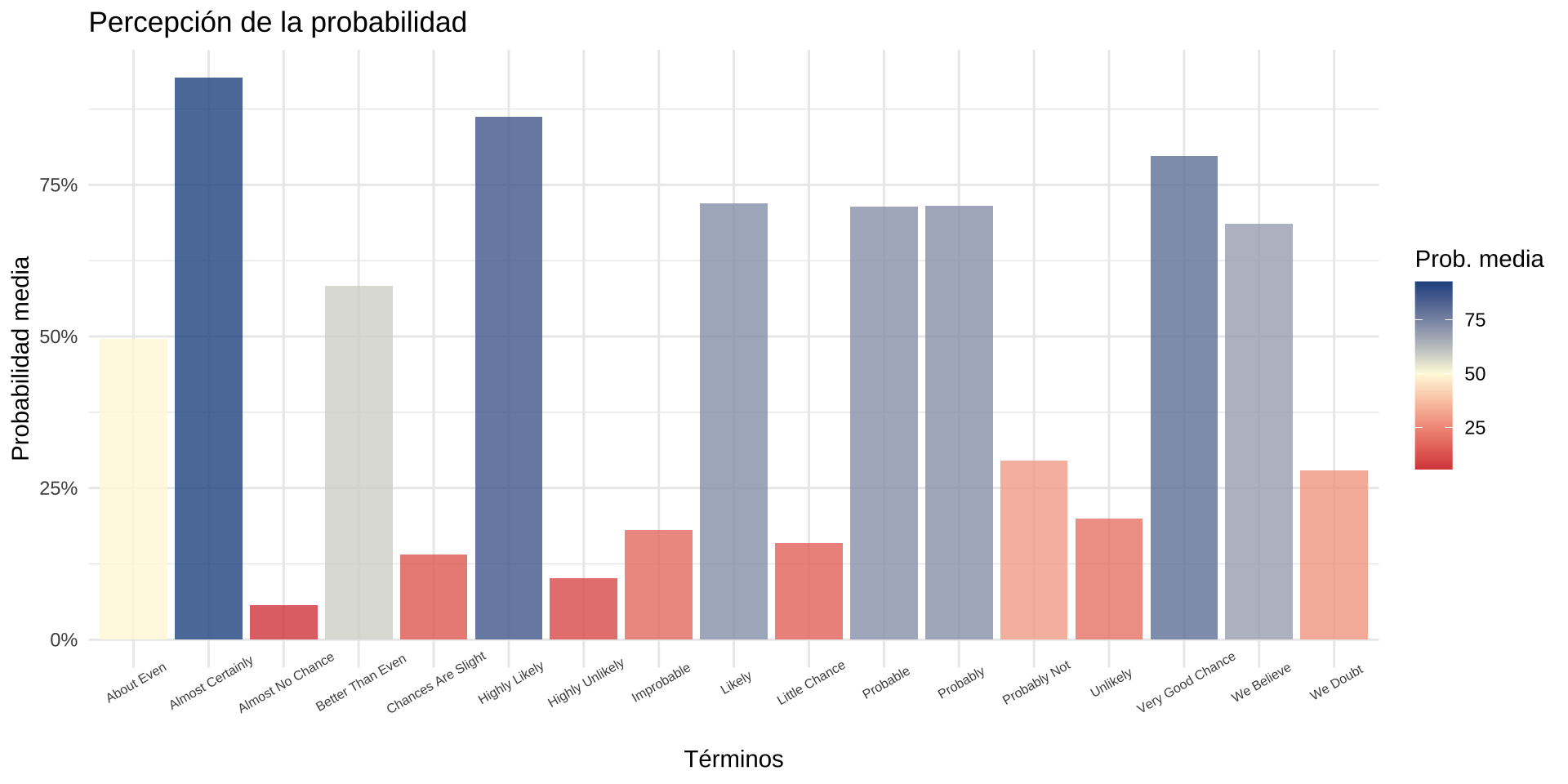
ggplot(resumen, aes(x = termino, y = mean_prob, fill = mean_prob)) +
geom_col(alpha = 0.8) +
scale_fill_gradient2(low = "#DA4A4A", mid = "#FEFADF", high = "#144F8D", midpoint = 50) +
scale_y_continuous(labels = scales::label_number(suffix = "%")) +
labs(fill = "Prob. media", x = "Términos", y = "Probabilidad media",
title = "Percepción de la probabilidad") +
theme_minimal() +
theme(axis.text.x = element_text(size = 6, angle = 30))Variables continuas
Por último, vamos a ordenar las barras de más a menos
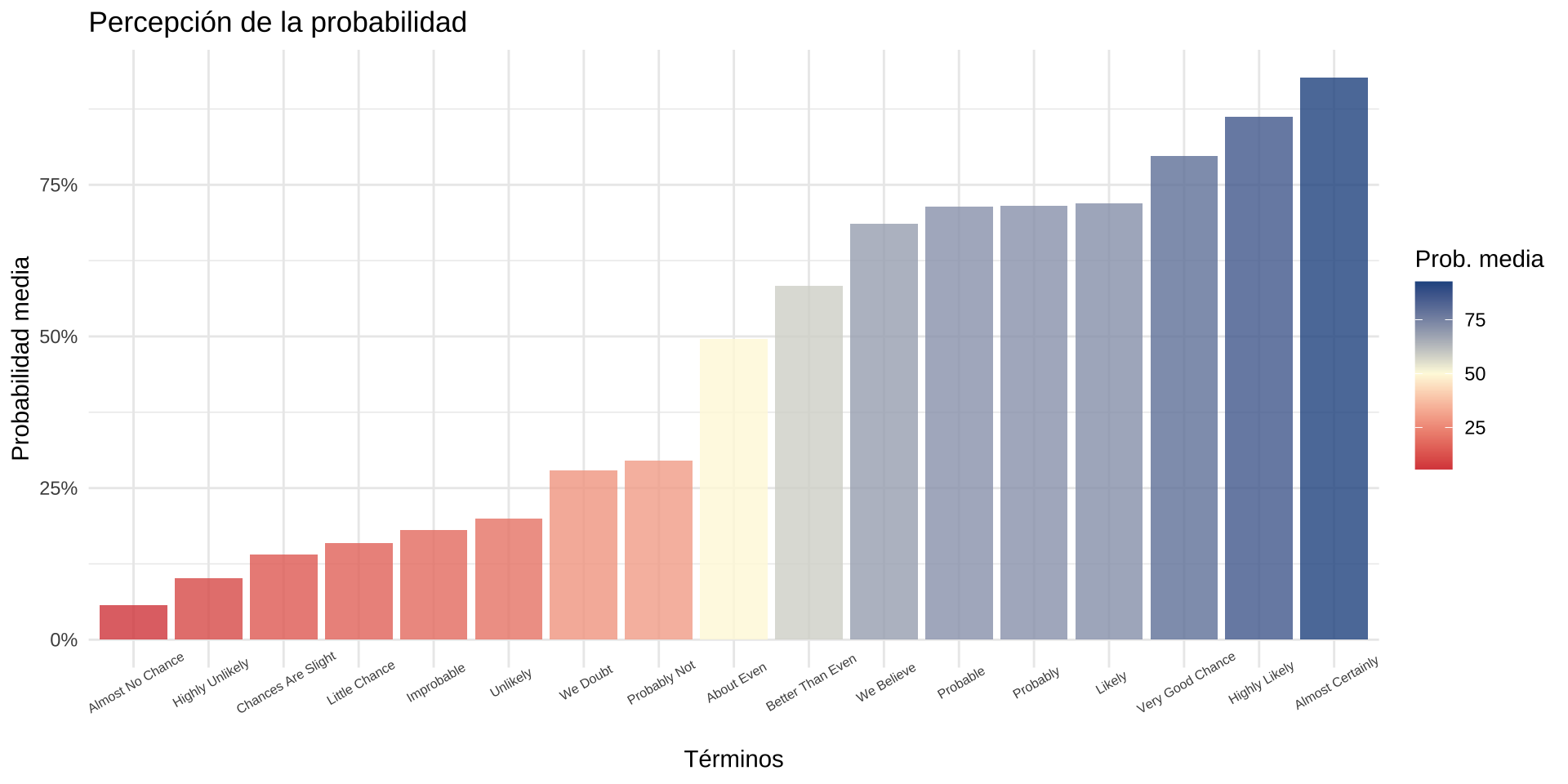
ggplot(resumen |> mutate(termino = fct_reorder(termino, mean_prob)), aes(x = termino, y = mean_prob, fill = mean_prob)) +
geom_col(alpha = 0.8) +
scale_fill_gradient2(low = "#DA4A4A", mid = "#FEFADF", high = "#144F8D", midpoint = 50) +
scale_y_continuous(labels = scales::label_number(suffix = "%")) +
labs(fill = "Prob. media", x = "Términos", y = "Probabilidad media",
title = "Percepción de la probabilidad") +
theme_minimal() +
theme(axis.text.x = element_text(size = 6, angle = 30))Variables continuas
Aun así al haber sumarizado, estamos perdiendo información…
¿Qué otros gráficos se te ocurren para hacer con variables continuas?
Algunos de los más habituales son:
- Distribuciones: histogramas, densidades (ridgeline), boxplot (alternativas: violín, raincloud plots)
- Evolución: gráficos de líneas, gráficos de área, series temporales, etc
- Correlaciones: mapas de calor, correlograma, grafos, etc
Distribuciones: histogramas
Nuestra primera alternativa será el conocido como histograma con geom_histogram()
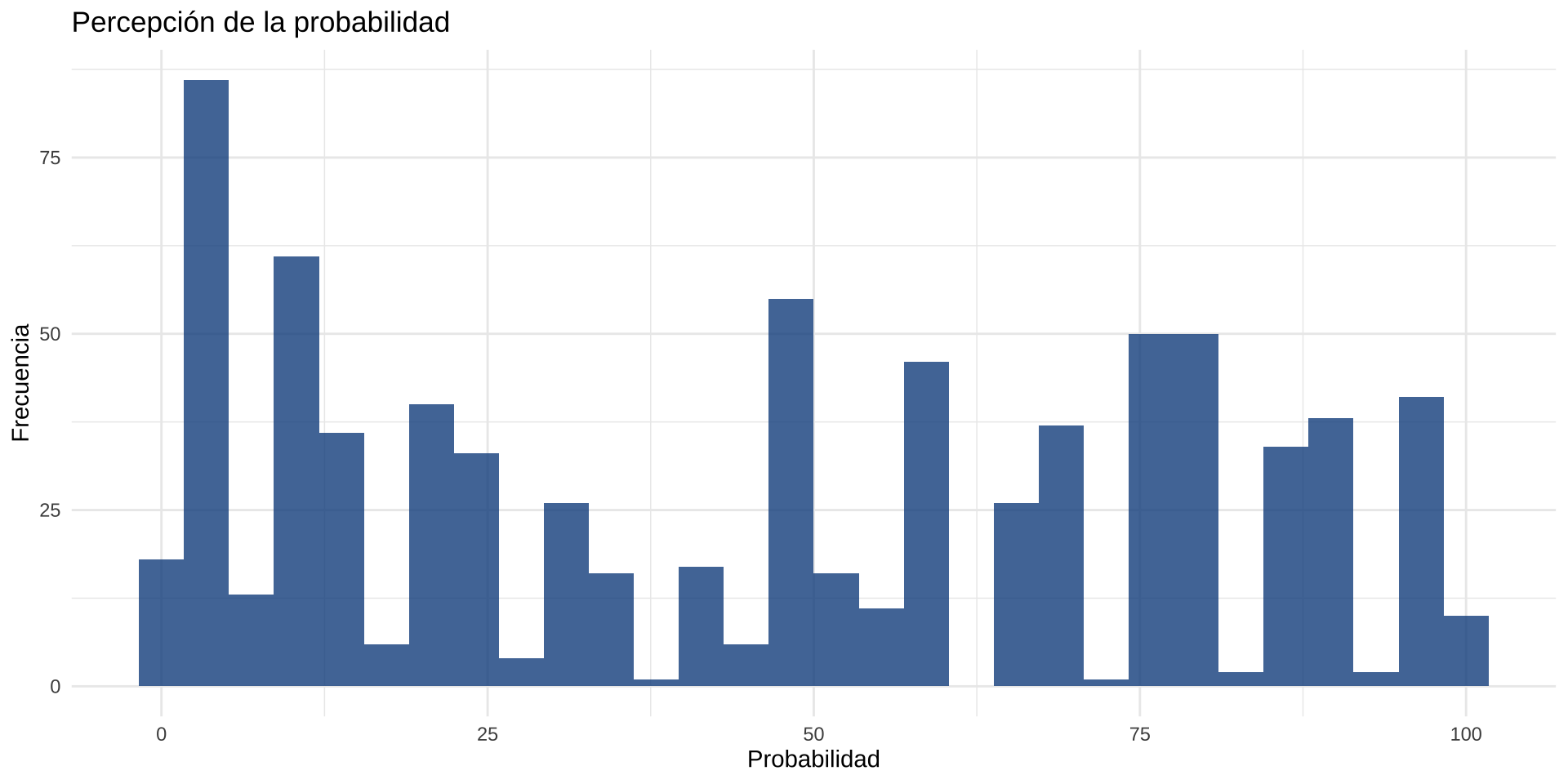
Fíjate que está realizando el histograma de todo el dataset.
Distribuciones: histogramas
El argumento bins = ... nos servirá para personalizar el nímero de barras que queremos. Fíjate que el gráfico es una proximación discreta de un gráfico de densidad.
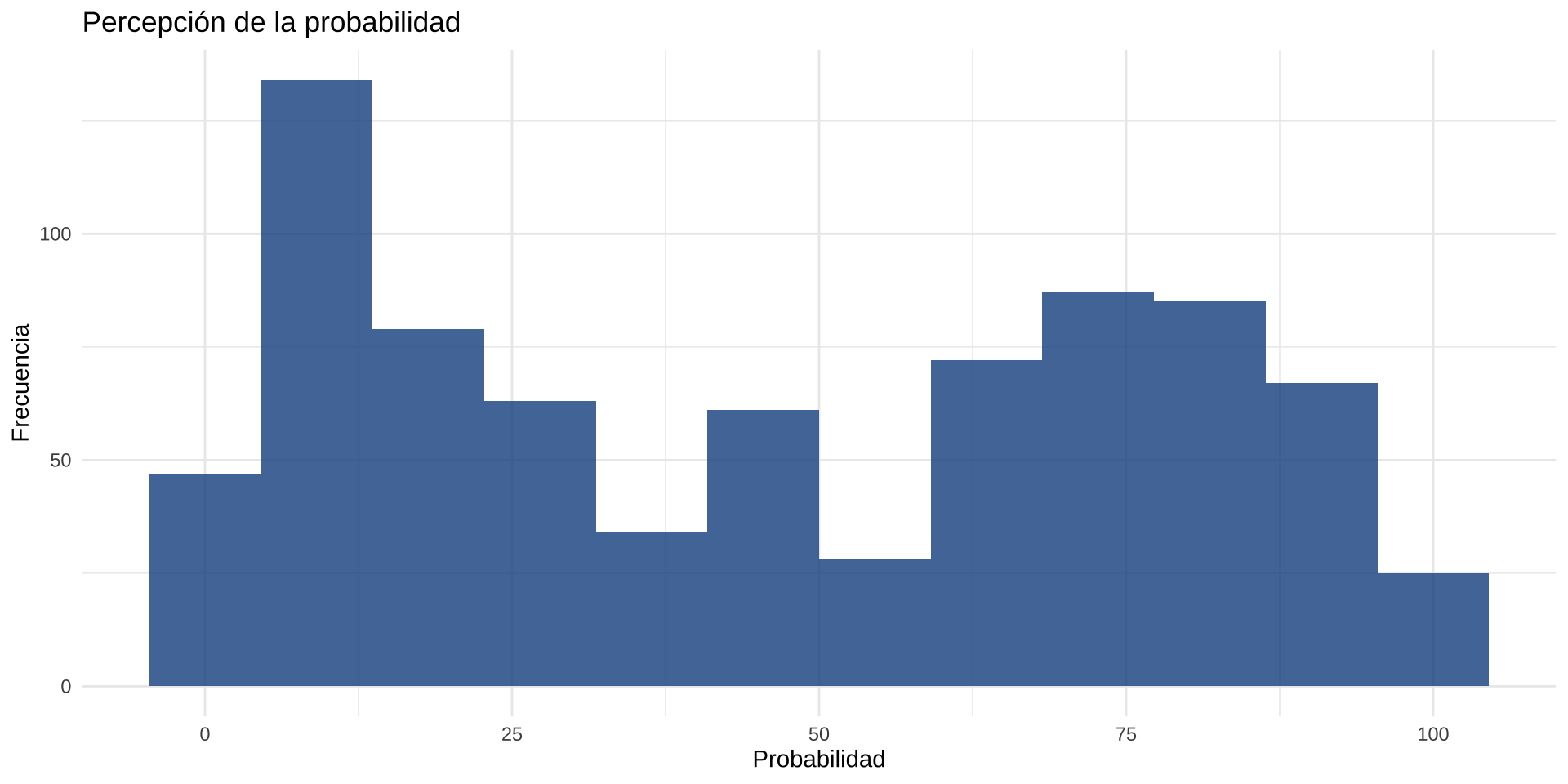
Distribuciones: histogramas
Si queremos hacer uno por término, basta con añadir a nuestro gráfico un facet_wrap() para componer
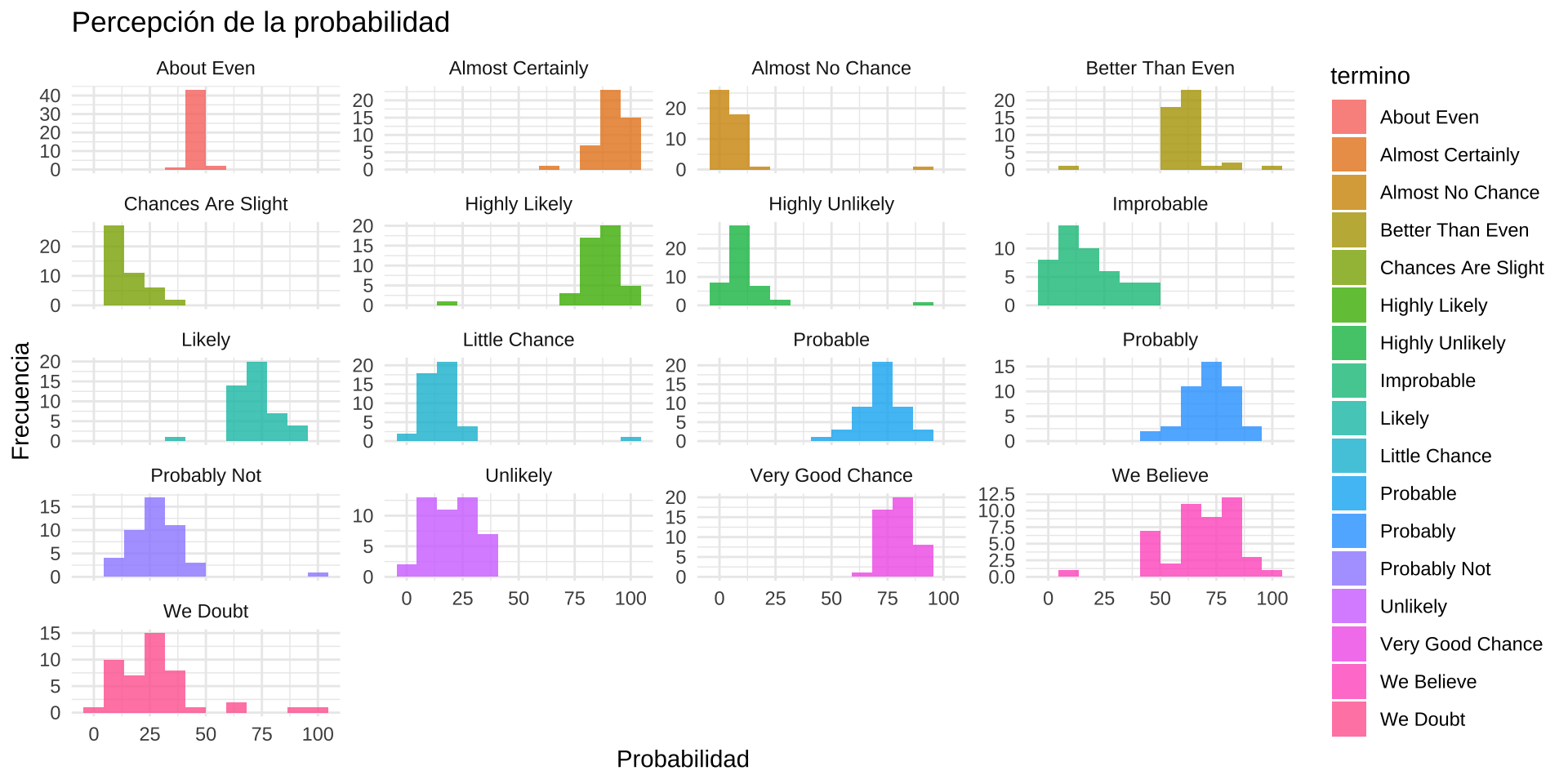
Distribuciones: histogramas
Vamos a filtrar solo algunos términos para poder usar la paleta scale_fill_brewer()
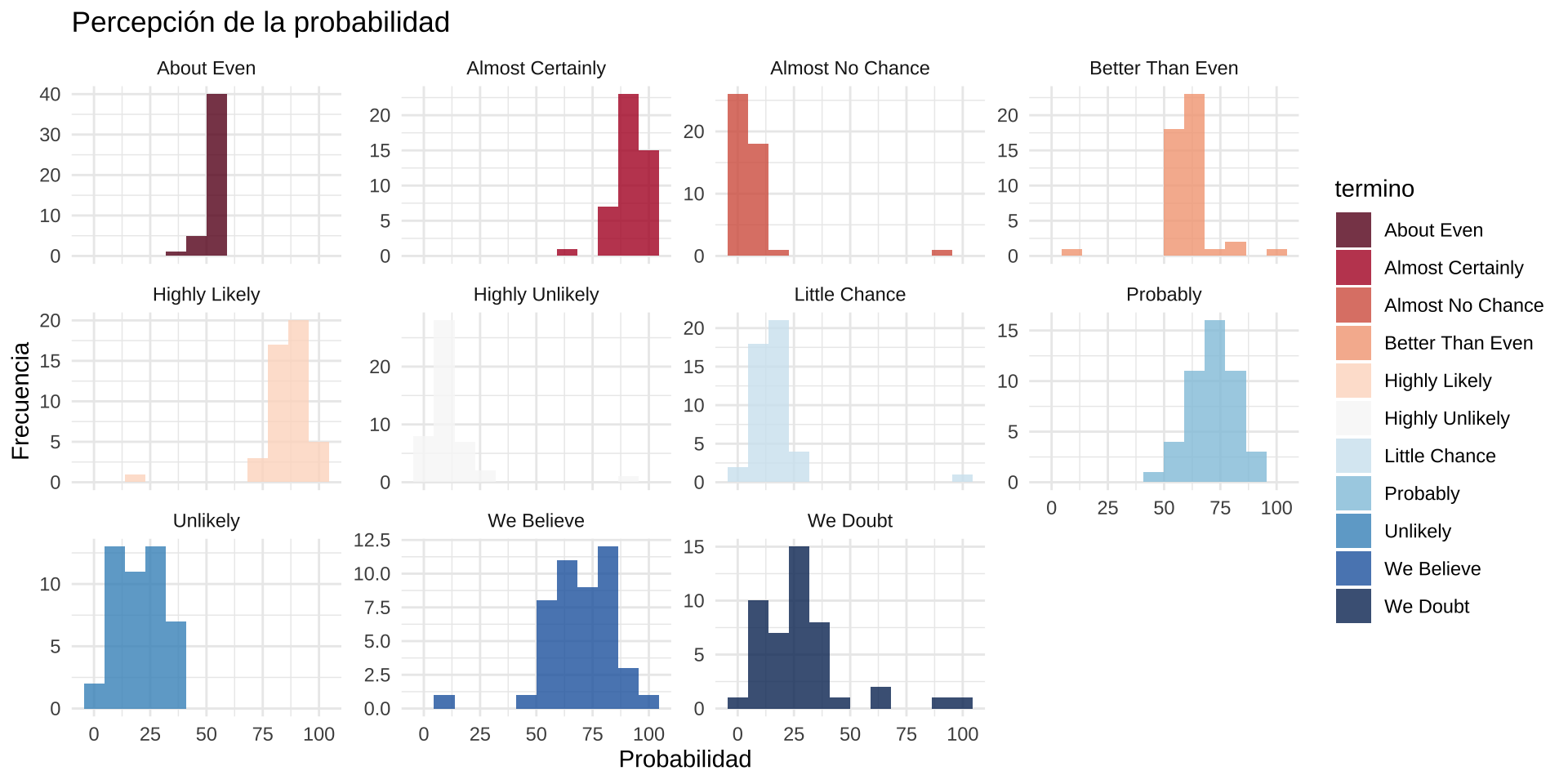
datos_tidy <-
datos_tidy |>
filter(!(termino %in% c("Chances Are Slight", "Improbable", "Probably Not", "Probable", "Likely", "Very Good Chance")))
ggplot(datos_tidy, aes(x = prob, fill = termino)) +
geom_histogram(bins = 12, alpha = 0.8) +
scale_fill_brewer(palette = "RdBu") +
facet_wrap(~termino, scale = "free_y", ncol = 4) +
labs(x = "Probabilidad", y = "Frecuencia",
title = "Percepción de la probabilidad") +
theme_minimal()Distribuciones: histogramas
Nos aparecen desordenadas así que de nuevo podemos hacer uso del paquete {forcats}
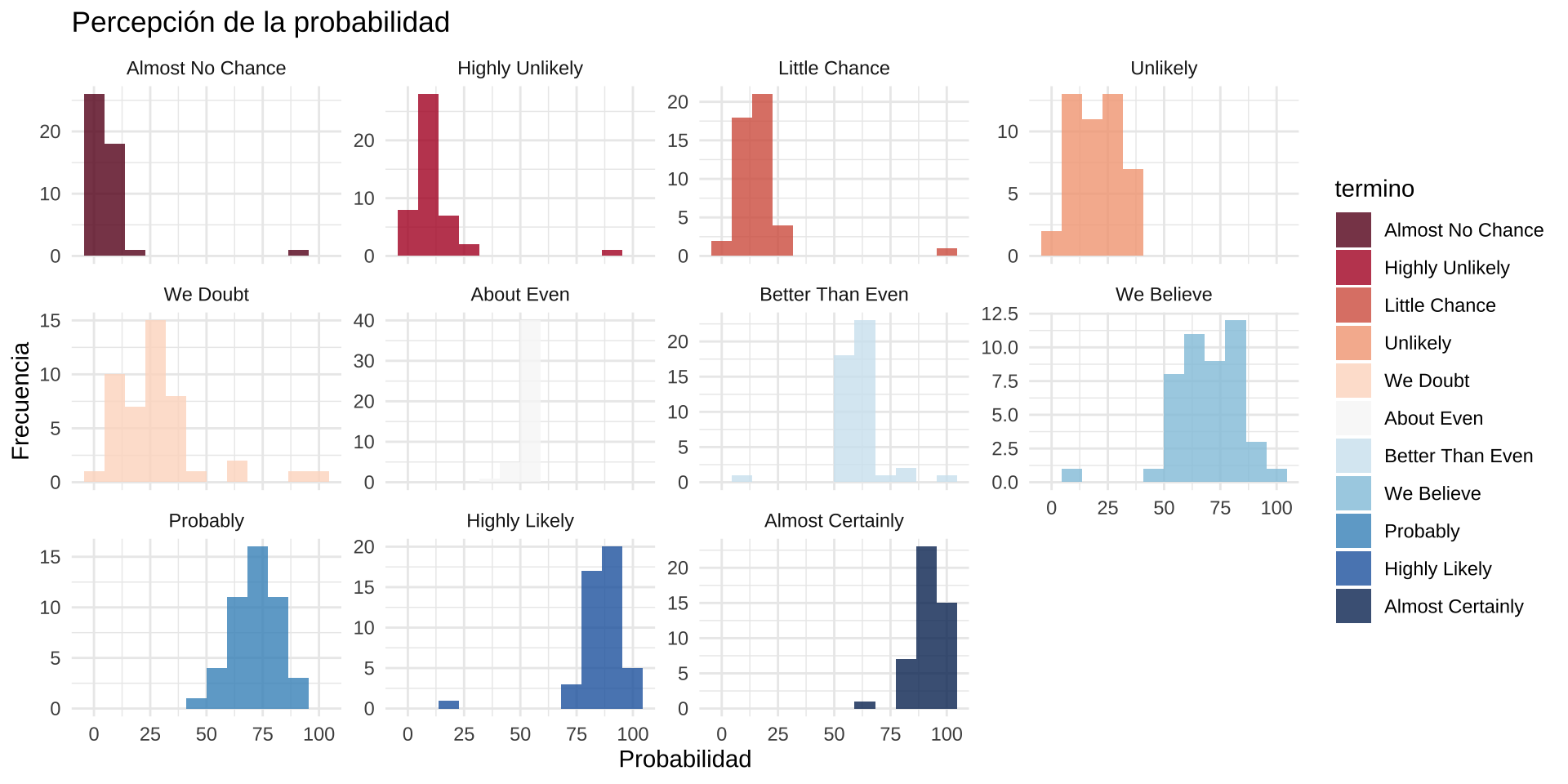
ggplot(datos_tidy |>
mutate(termino = fct_reorder(termino, prob, .fun = mean)), aes(x = prob, fill = termino)) +
geom_histogram(bins = 12, alpha = 0.8) +
scale_fill_brewer(palette = "RdBu") +
facet_wrap(~termino, scale = "free_y", ncol = 4) +
labs(x = "Probabilidad", y = "Frecuencia",
title = "Percepción de la probabilidad") +
theme_minimal()Paréntesis: tema y fuente
Vamos a añadir una fuente personaliza al gráfico anterior.
Con el paquete {showtext} podemos cargar fuentes de https://fonts.google.com/: con font_add_google() añadimos la fuente y con showtext_auto() habilitamos su uso.
Con theme_set() podemos fijar un tema base (en nuestro caso theme_minimal(base_family = ...)) y con theme_update() añadimos el resto de personalizaciones
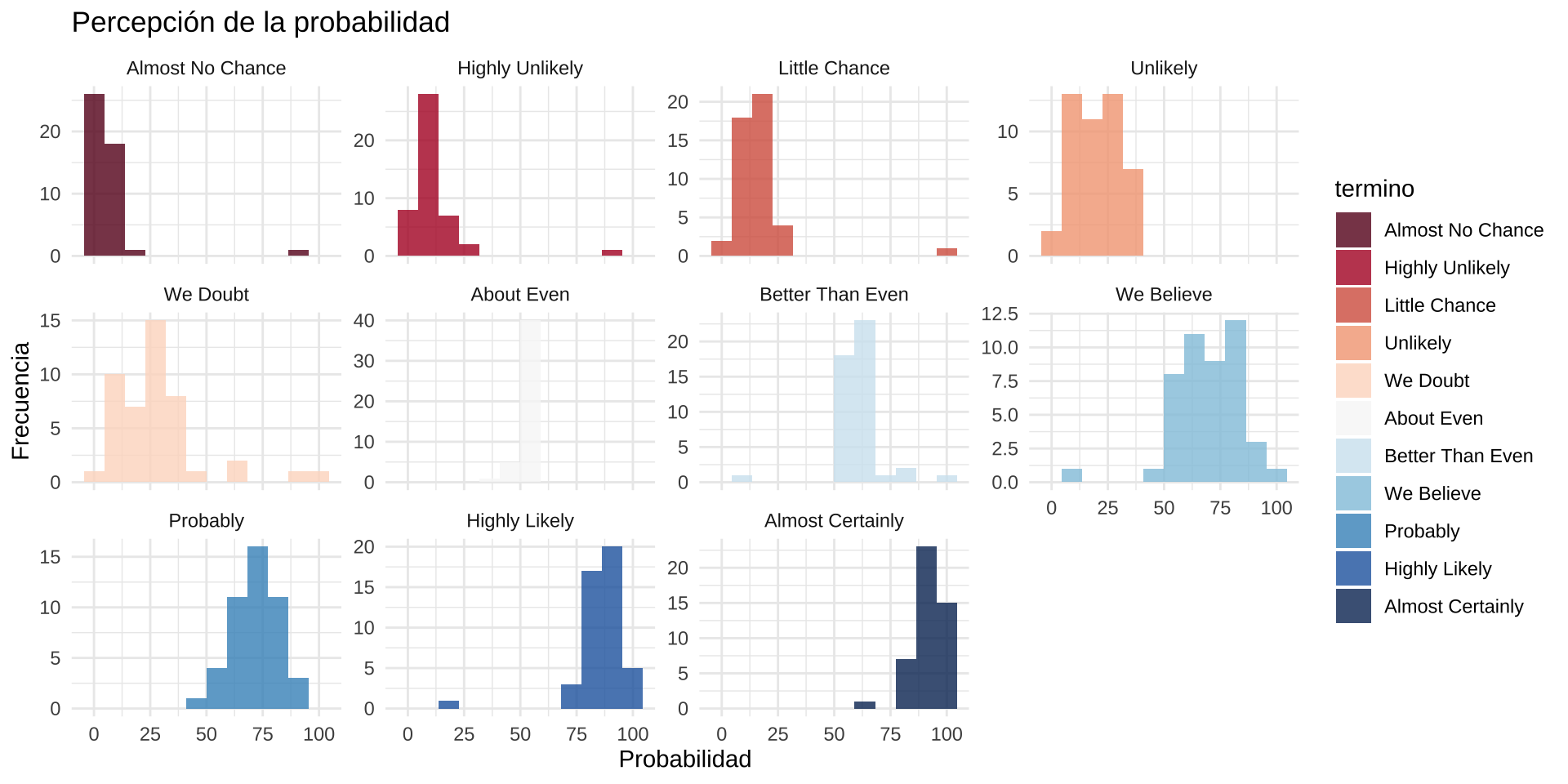
Distribuciones: densidades
Los histogramas en realidad son una aproximación discreta de los gráficos de densidad (asumiendo que los intervalos se pudieran ir haciendo tan pequeños como queramos).
Las densidades mejoran la robustez al histograma. Para ello usaremos geom_density()
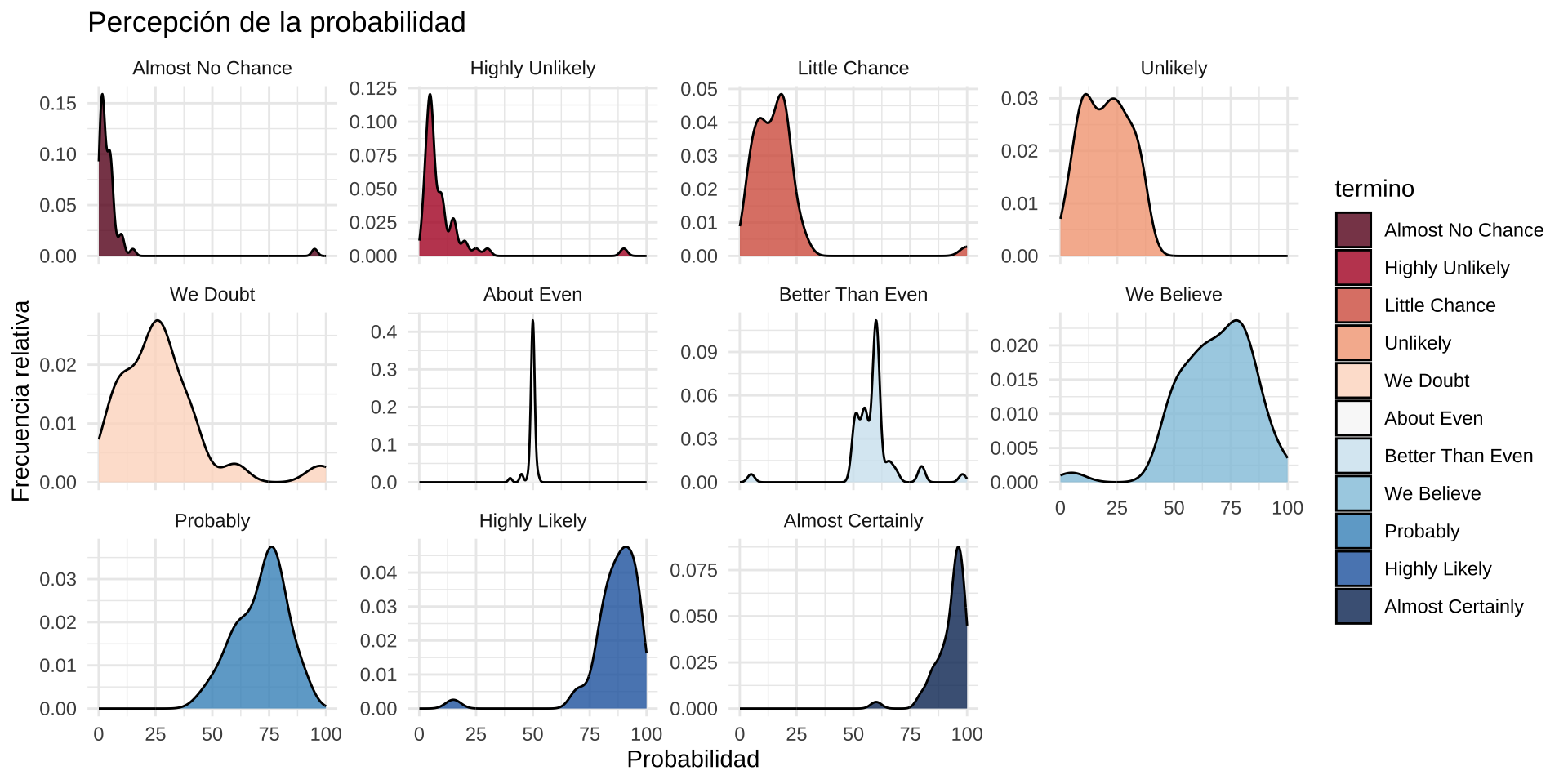
ggplot(datos_tidy |>
mutate(termino = fct_reorder(termino, prob, .fun = mean)), aes(x = prob, fill = termino)) +
geom_density(alpha = 0.8) +
scale_fill_brewer(palette = "RdBu") +
facet_wrap(~termino, scale = "free_y", ncol = 4) +
labs(x = "Probabilidad", y = "Frecuencia relativa",
title = "Percepción de la probabilidad") +
theme_minimal()Distribuciones: densidades
A veces puede ser interesante [superponer las densidades], lo cual lo podemos hacer con geom_density_ridges() del paquete {ggridges} (ahora sí necesitamos indicarle un y = ...)
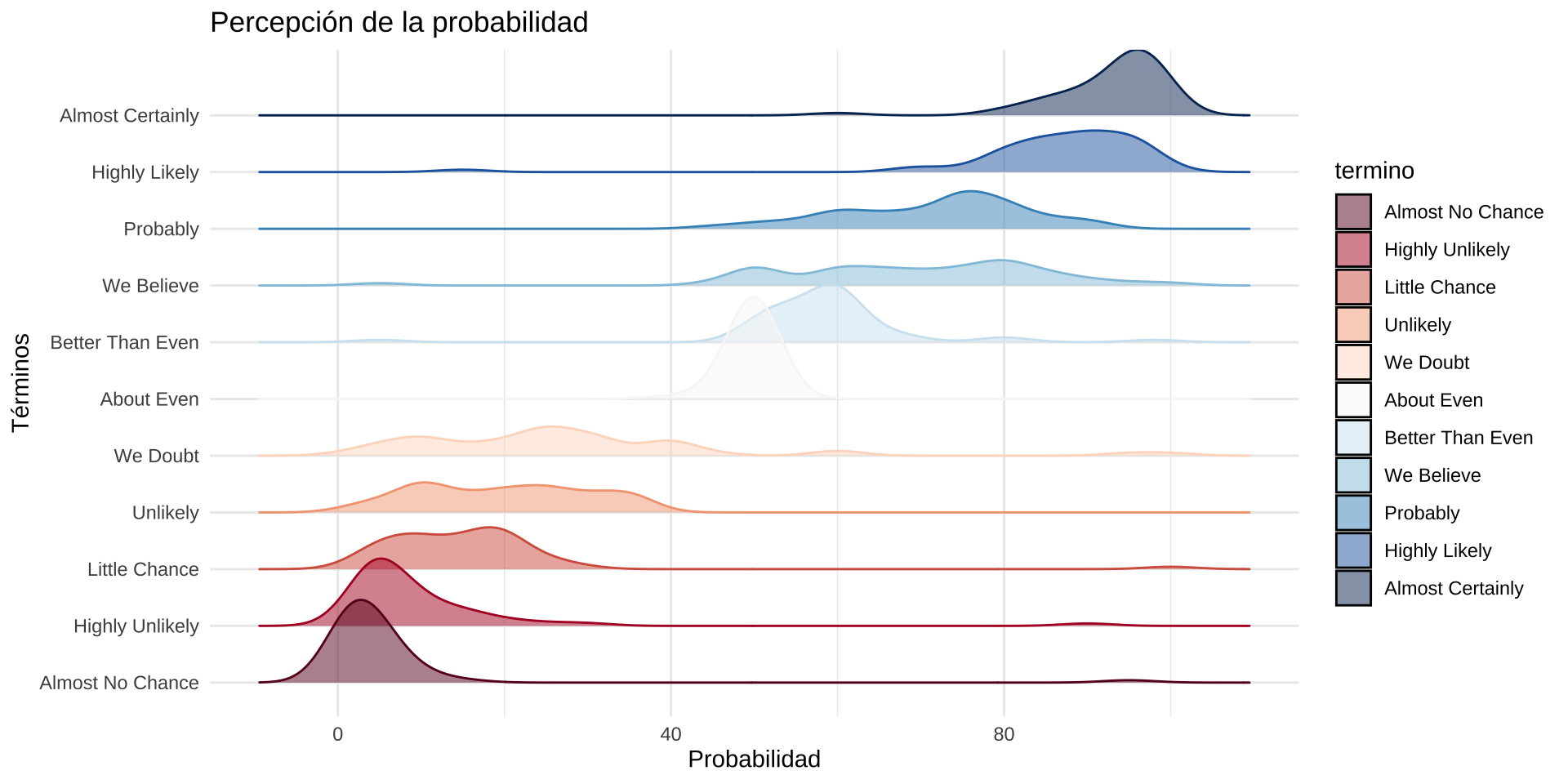
library(ggridges)
ggplot(datos_tidy |>
mutate(termino = fct_reorder(termino, prob, .fun = mean)),
aes(x = prob, y = termino, fill = termino, color = termino)) +
geom_density_ridges(alpha = 0.5) +
scale_fill_brewer(palette = "RdBu") +
scale_color_brewer(palette = "RdBu") +
guides(color = "none") +
labs(x = "Probabilidad", y = "Términos",
title = "Percepción de la probabilidad") +
theme_minimal()Distribuciones: boxplot
Una opción muy habitual en variables continuas son los gráficos de cajas y bigotes o boxplots
Para realizar estos gráficos debemos usar la geometría geom_boxplot()
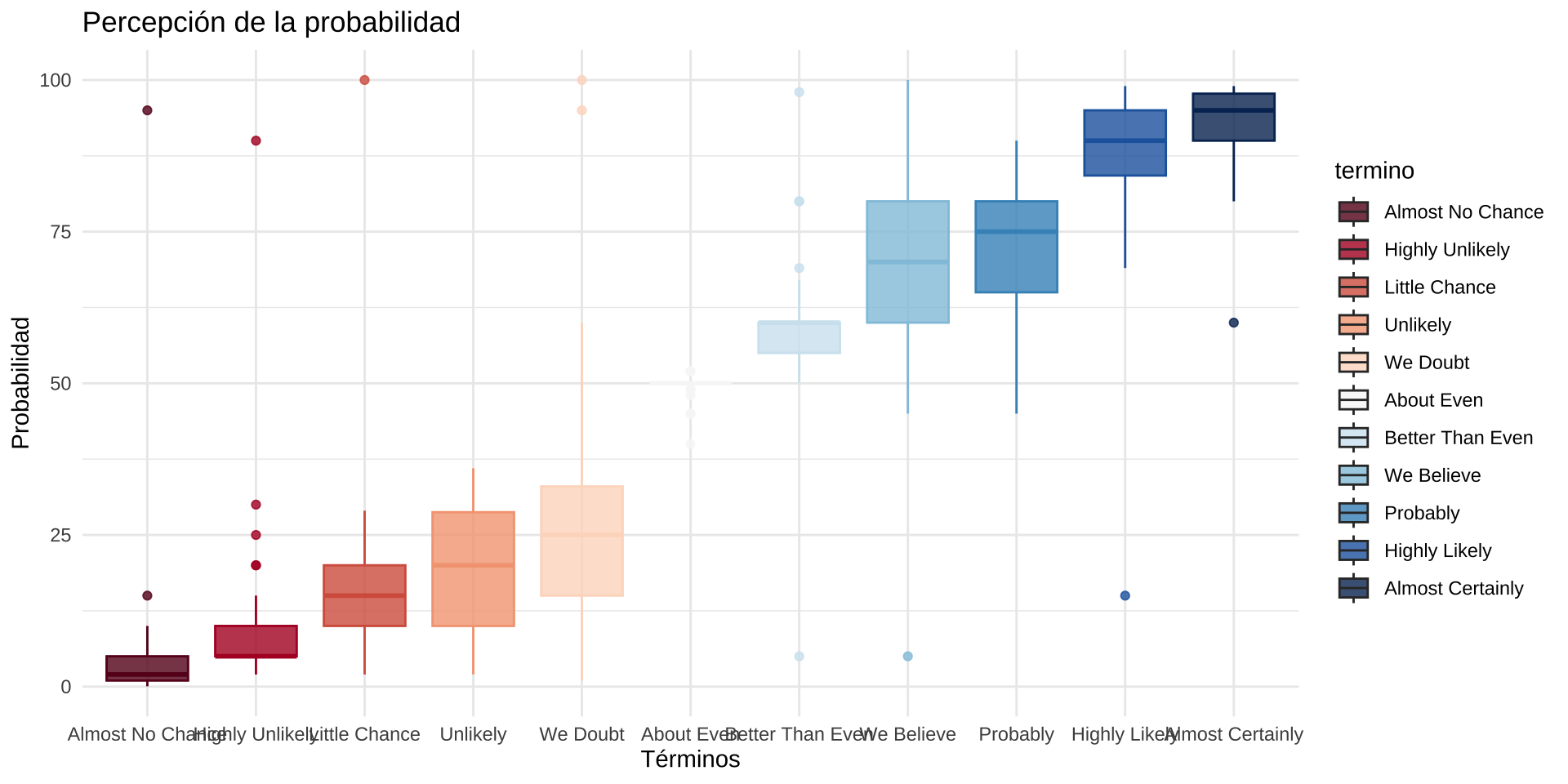
ggplot(datos_tidy |>
mutate(termino = fct_reorder(termino, prob, .fun = mean)),
aes(x = termino, y = prob, fill = termino, color = termino)) +
geom_boxplot(alpha = 0.8) +
scale_fill_brewer(palette = "RdBu") +
scale_color_brewer(palette = "RdBu") +
guides(color = "none") +
labs(x = "Términos", y = "Probabilidad",
title = "Percepción de la probabilidad") +
theme_minimal()Distribuciones: jitter
Los boxplot a veces pueden ser insuficiente y podemos usar geom_jitter() que nos añadirá puntos como un «gotelé aleatorio» (ver https://hausetutorials.netlify.app/posts/2019-02-22-why-we-should-never-use-barplots-use-geomquasirandom-instead/)
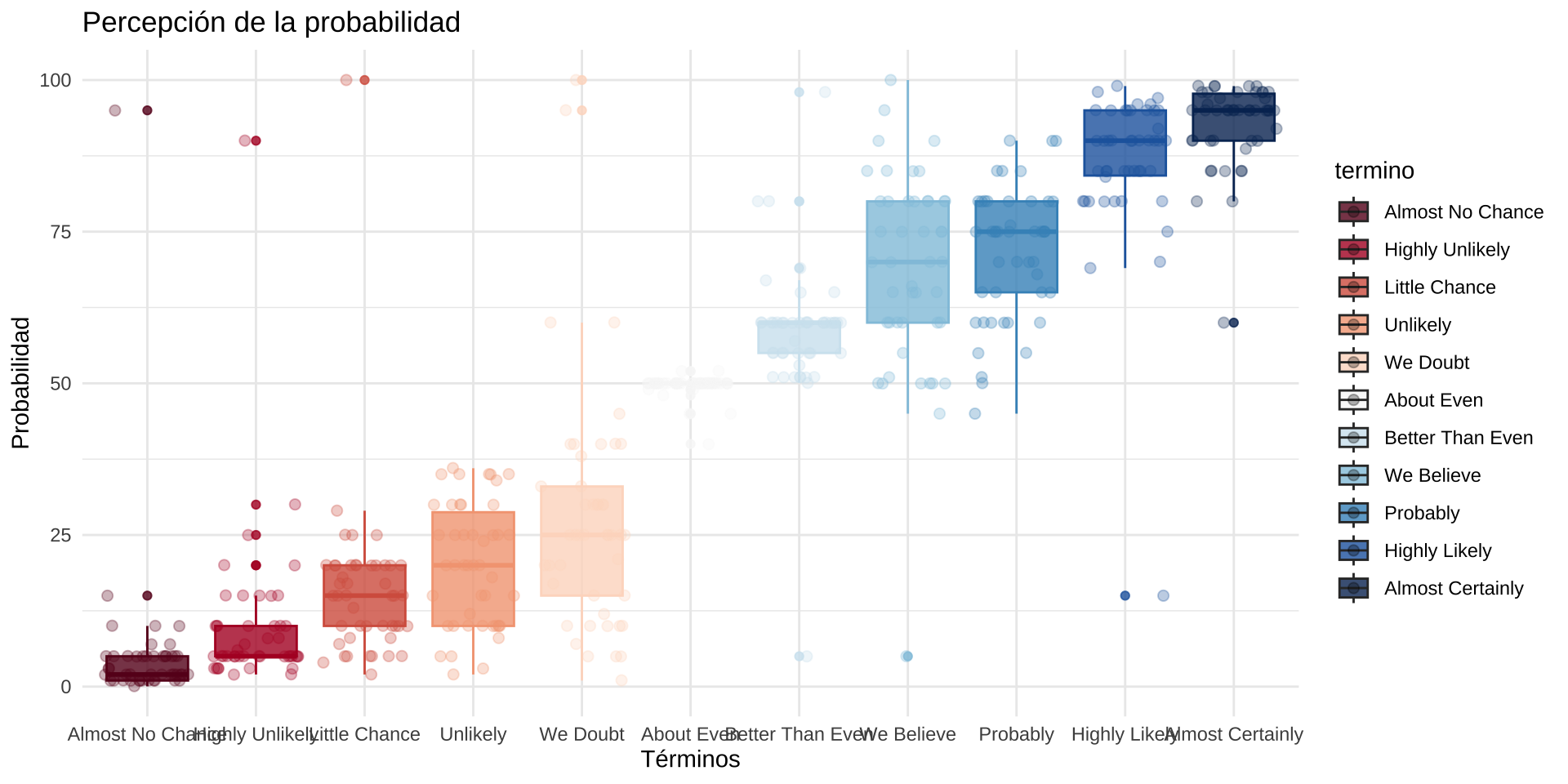
ggplot(datos_tidy |>
mutate(termino = fct_reorder(termino, prob, .fun = mean)),
aes(x = termino, y = prob, fill = termino, color = termino)) +
geom_boxplot(alpha = 0.8) +
geom_jitter(alpha = 0.3, size = 2) +
scale_fill_brewer(palette = "RdBu") +
scale_color_brewer(palette = "RdBu") +
guides(color = "none") +
labs(x = "Términos", y = "Probabilidad",
title = "Percepción de la probabilidad") +
theme_minimal()Distribuciones: jitter
Si te fijas los outliers aparecen dos veces ya que el boxplot los marca. Dentro de geom_boxplot() podemos indicarle la forma, color y alpha de los atípicos.
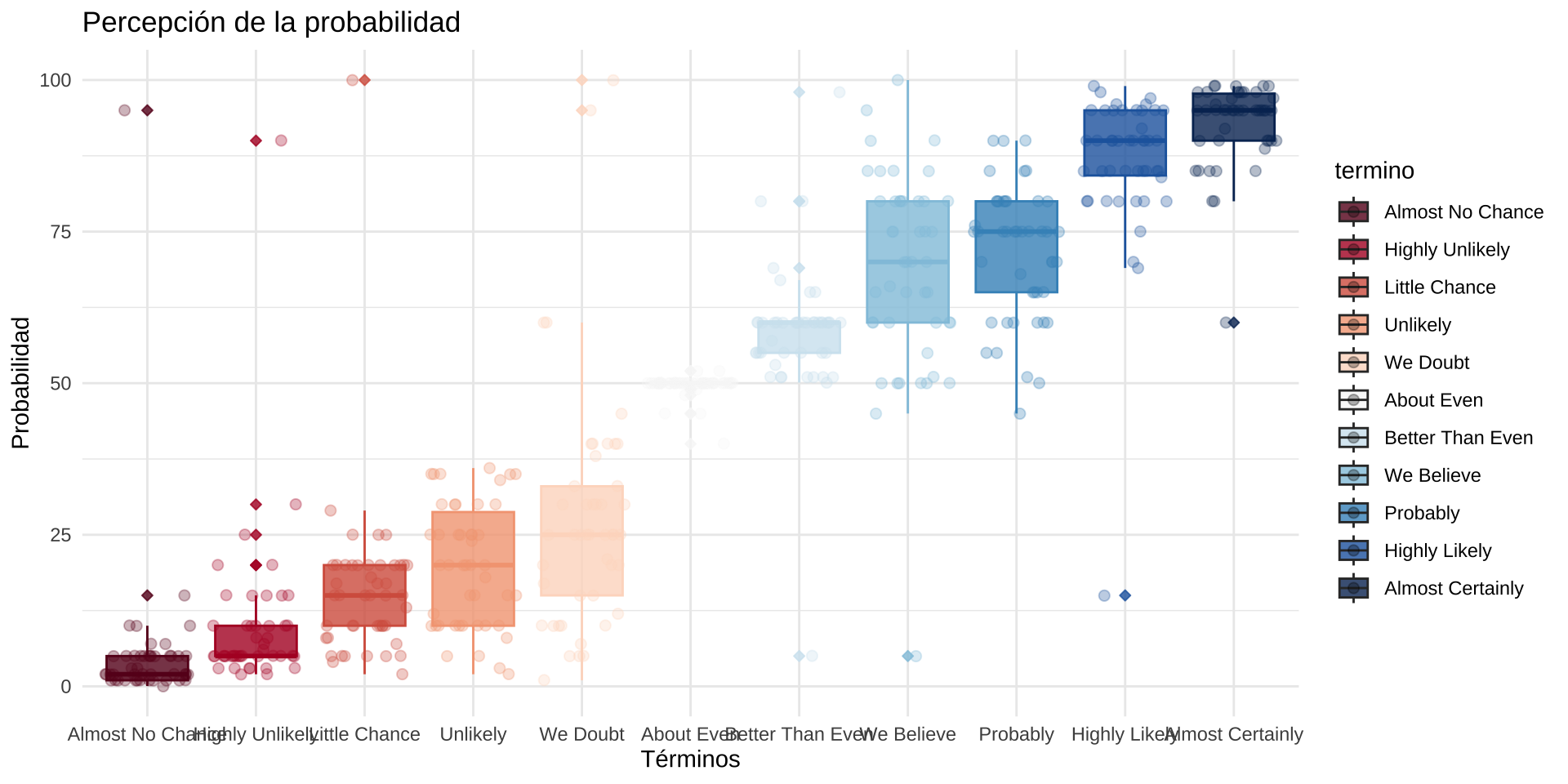
ggplot(datos_tidy |>
mutate(termino = fct_reorder(termino, prob, .fun = mean)),
aes(x = termino, y = prob, fill = termino, color = termino)) +
geom_boxplot(alpha = 0.8, outlier.shape = 23) +
geom_jitter(alpha = 0.3, size = 2) +
scale_fill_brewer(palette = "RdBu") +
scale_color_brewer(palette = "RdBu") +
guides(color = "none") +
labs(x = "Términos", y = "Probabilidad",
title = "Percepción de la probabilidad") +
theme_minimal()Distribuciones: jitter
Si te fijas los outliers aparecen dos veces ya que el boxplot los marca. Dentro de geom_boxplot() podemos indicarle la forma, color y alpha de los atípicos.
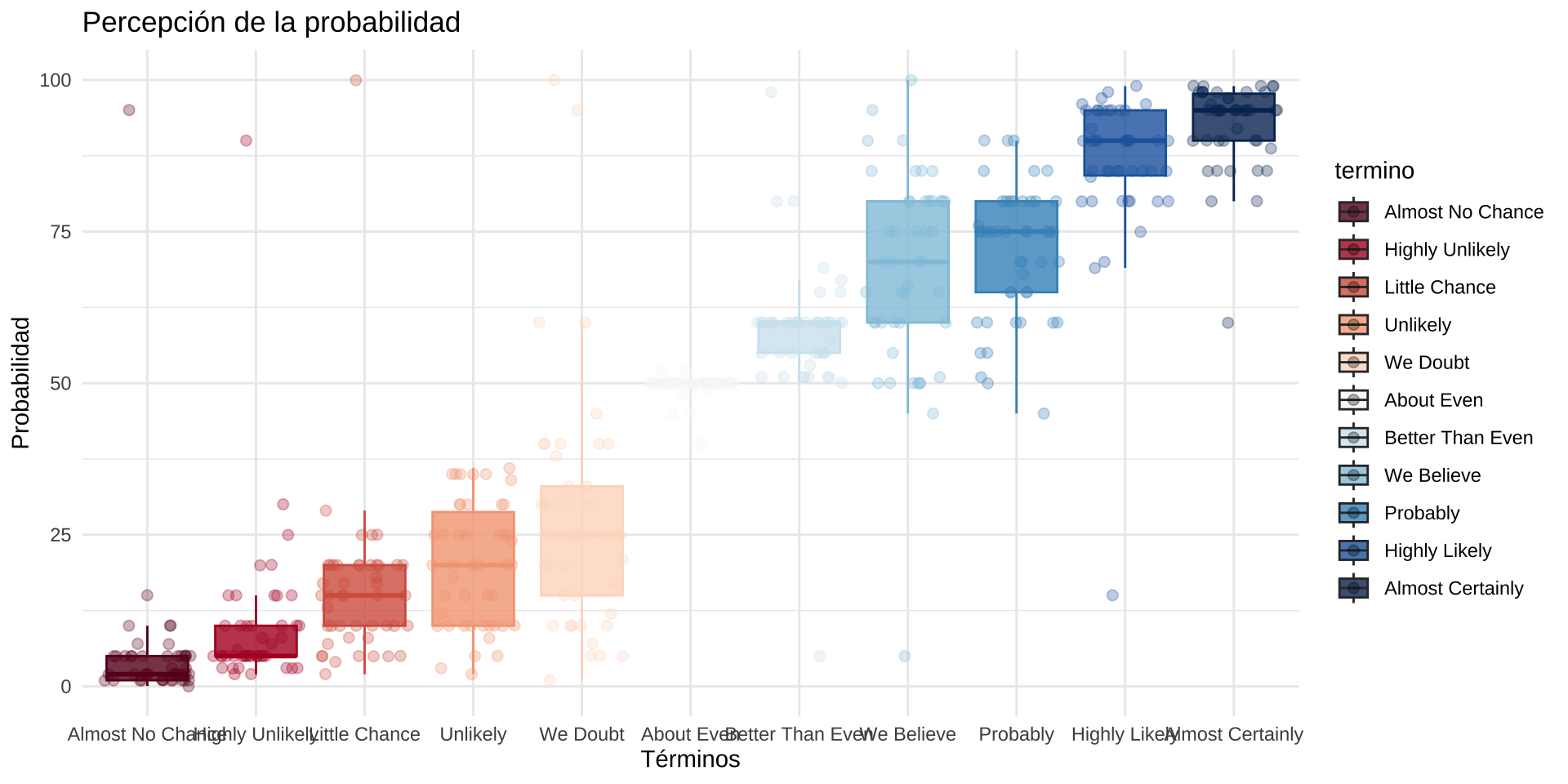
ggplot(datos_tidy |>
mutate(termino = fct_reorder(termino, prob, .fun = mean)),
aes(x = termino, y = prob, fill = termino, color = termino)) +
geom_boxplot(alpha = 0.8, outlier.alpha = 0) +
geom_jitter(alpha = 0.3, size = 2) +
scale_fill_brewer(palette = "RdBu") +
scale_color_brewer(palette = "RdBu") +
guides(color = "none") +
labs(x = "Términos", y = "Probabilidad",
title = "Percepción de la probabilidad") +
theme_minimal()Distribuciones: geom_quasirandom
Podemos mejorar el «gotelé aleatorio» con geom_quasirandom() del paquete {ggbeeswarm} (con width = ... controlamos la anchura de lo aleatorio)
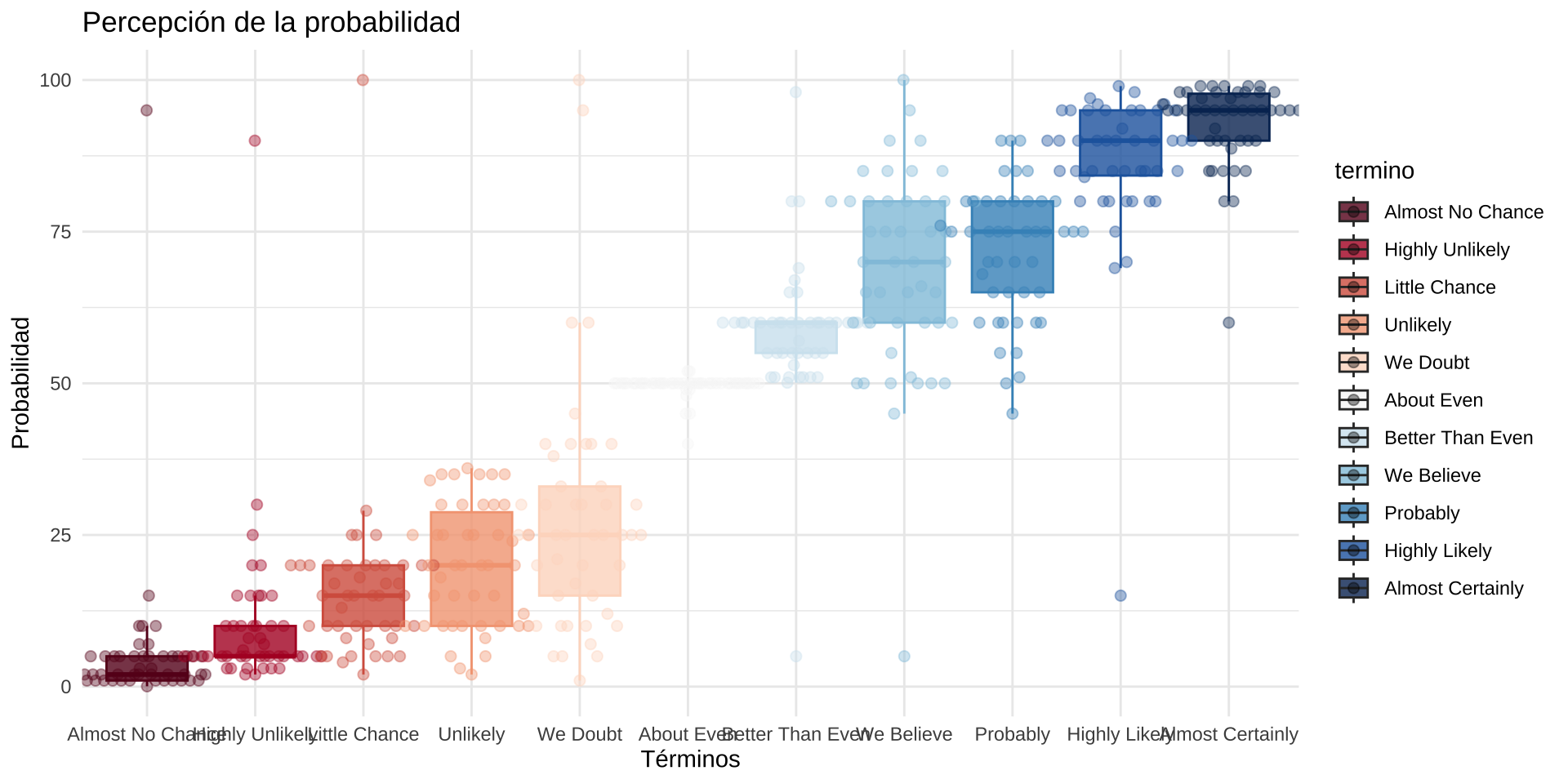
ggplot(datos_tidy |>
mutate(termino = fct_reorder(termino, prob, .fun = mean)),
aes(x = termino, y = prob, fill = termino, color = termino)) +
geom_boxplot(alpha = 0.8, outlier.alpha = 0) +
geom_quasirandom(size = 2, alpha = 0.4, width = 0.7) +
scale_fill_brewer(palette = "RdBu") +
scale_color_brewer(palette = "RdBu") +
guides(color = "none") +
labs(x = "Términos", y = "Probabilidad",
title = "Percepción de la probabilidad") +
theme_minimal()Distribuciones: violin
Para solventar los problemas de los box-plots, una alternativa muy popular son los gráficos de violín (en realidad es una densidad reflejada)
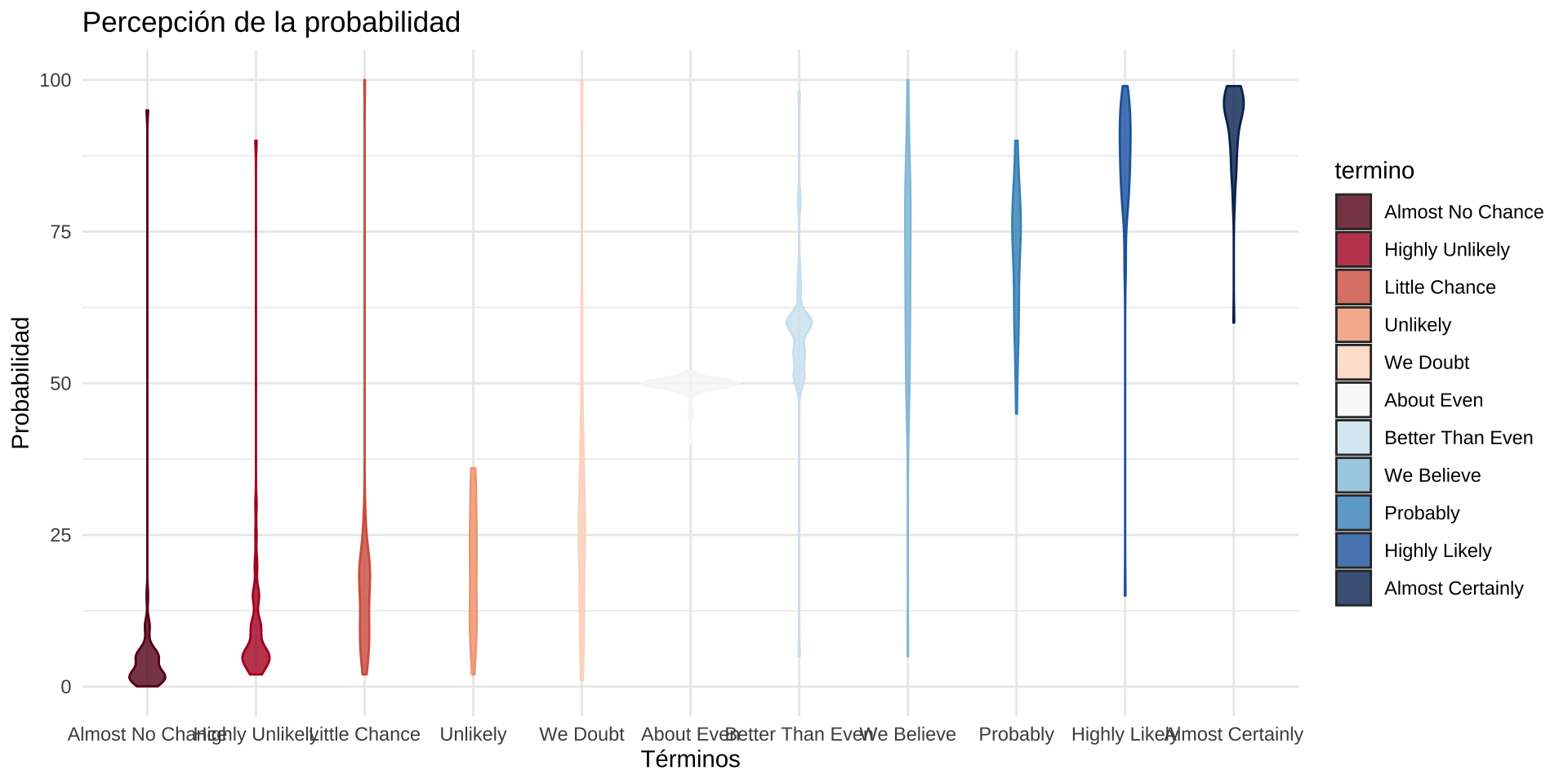
ggplot(datos_tidy |>
mutate(termino = fct_reorder(termino, prob, .fun = mean)),
aes(x = termino, y = prob, fill = termino, color = termino)) +
geom_violin(alpha = 0.8) +
scale_fill_brewer(palette = "RdBu") +
scale_color_brewer(palette = "RdBu") +
guides(color = "none") +
labs(x = "Términos", y = "Probabilidad",
title = "Percepción de la probabilidad") +
theme_minimal()Distribuciones: violin
Con el argumento scale = "count" las àreas son proporcionales al número de observaciones en cada violín (por defecto scale = "area", todos la misma área). Con bw = ... modulamos la suavidad del kernel usado (bandwidth).
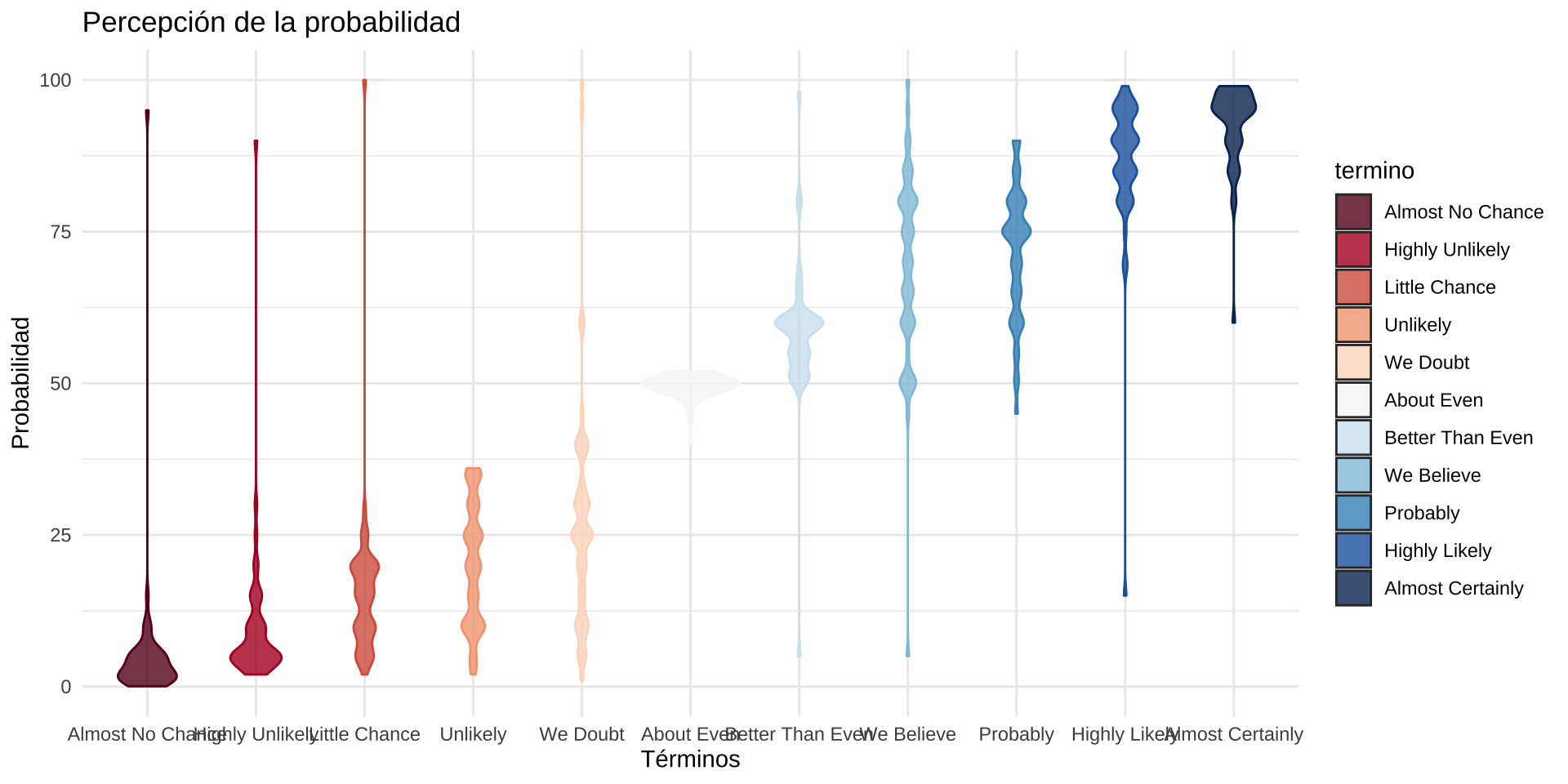
ggplot(datos_tidy |>
mutate(termino = fct_reorder(termino, prob, .fun = mean)),
aes(x = termino, y = prob, fill = termino, color = termino)) +
geom_violin(alpha = 0.8, scale = "count", bw = 1.5) +
scale_fill_brewer(palette = "RdBu") +
scale_color_brewer(palette = "RdBu") +
guides(color = "none") +
labs(x = "Términos", y = "Probabilidad",
title = "Percepción de la probabilidad") +
theme_minimal()Evolución: gráficos de líneas
Otra categoría muy común de gráficos con variables continusa son los gráficos de evolución
El más simple es el [gráfico de líneas] {.hl-yellow}, que podemos construir con geom_line(), y para el que ahora sí necesitamos un x = ... y un y = ...
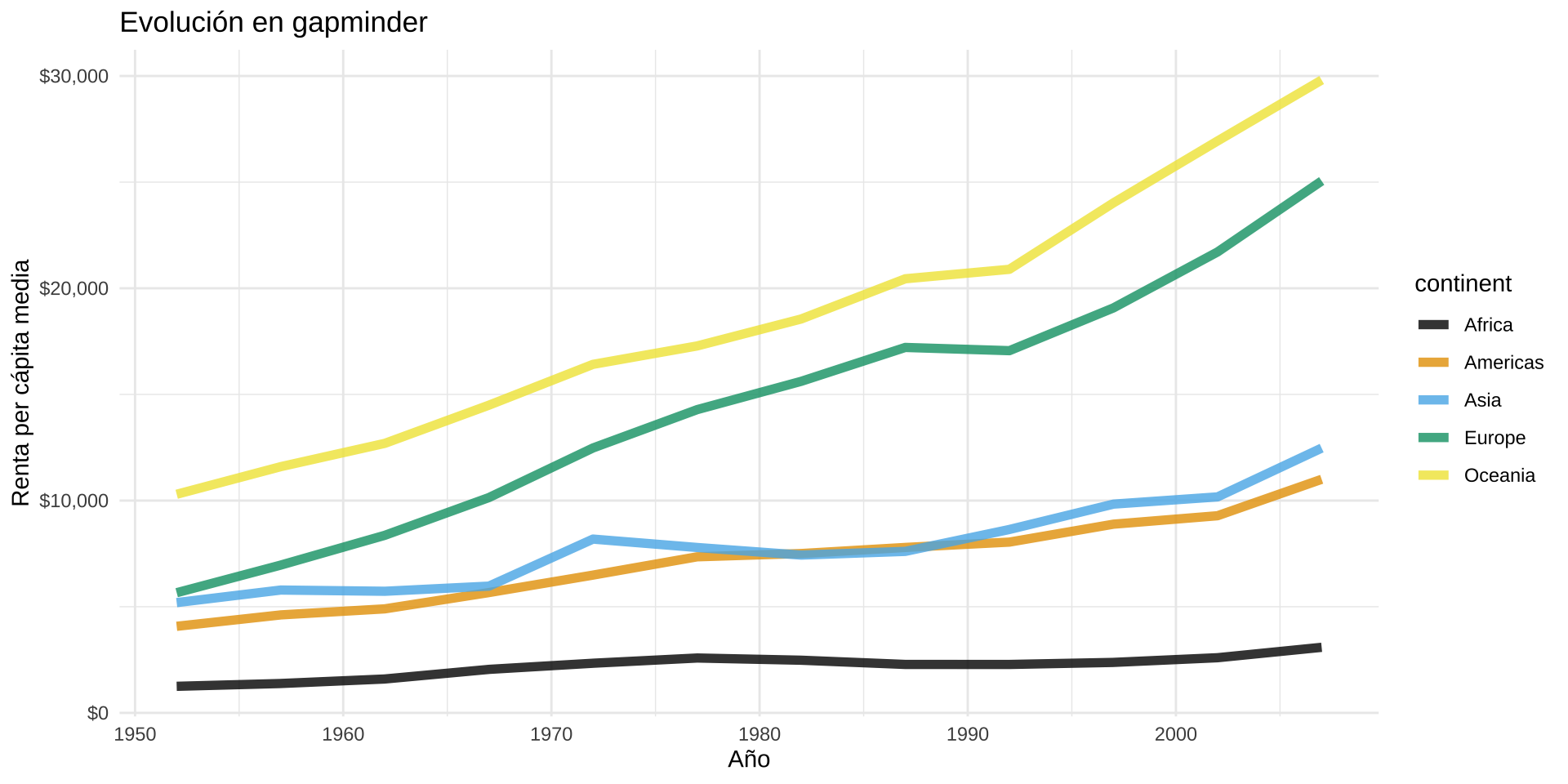
ggplot(gapminder |> summarise(mean_gdp = mean(gdpPercap), .by = c(continent, year)), aes(x = year, y = mean_gdp, color = continent)) +
geom_line(alpha = 0.8, linewidth = 2) +
scale_y_continuous(labels = scales::label_dollar()) +
scale_color_colorblind() +
labs(x = "Año", y = "Renta per cápita media",
title = "Evolución en gapminder") +
theme_minimal()Evolución: gráficos de líneas
Fíjate que dando color = ... nos hace solo una gráfica por variable de grupo. Si usamos geom_step() en su lugar obtenemos un gráfico de escalera
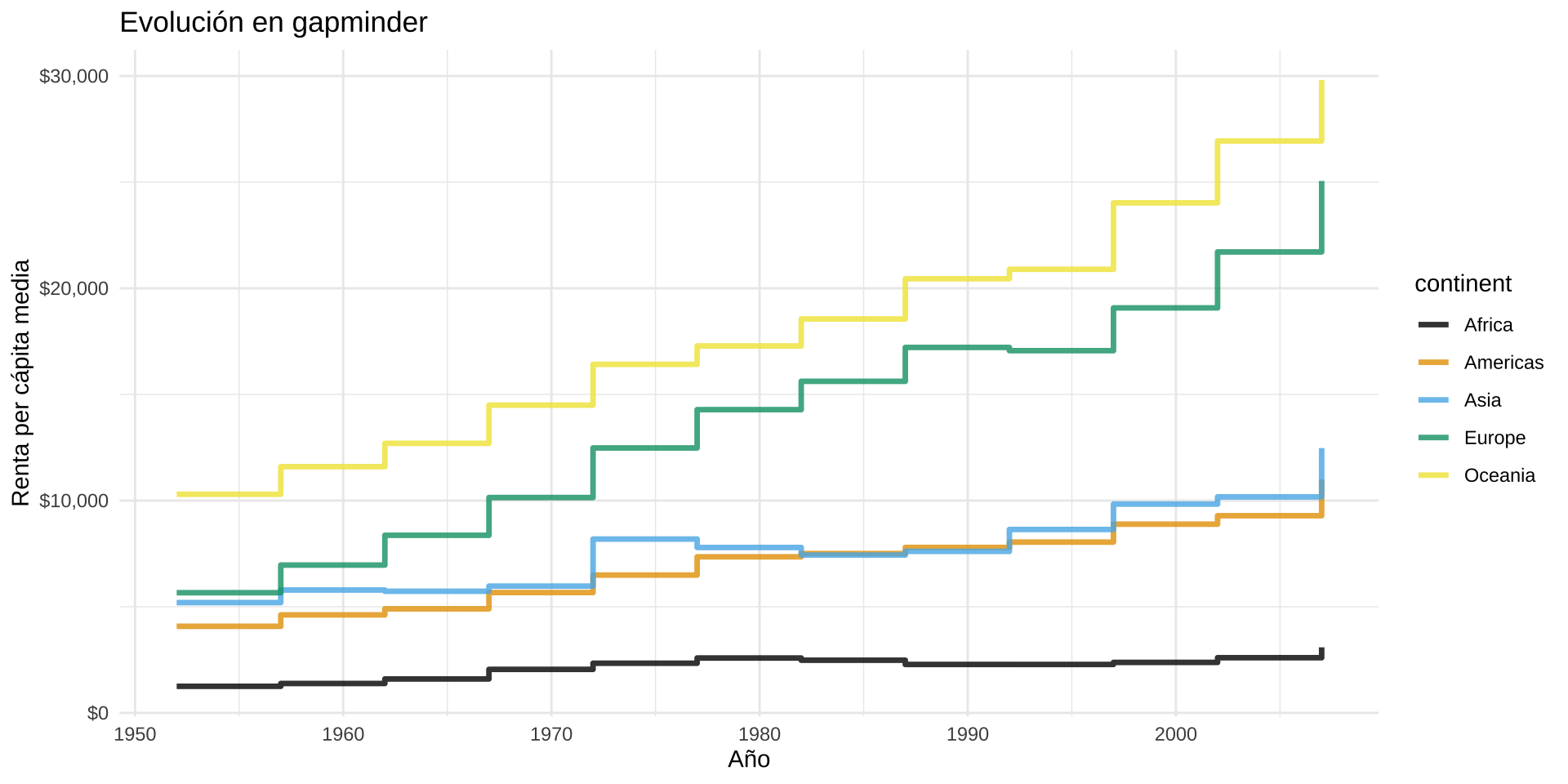
ggplot(gapminder |> summarise(mean_gdp = mean(gdpPercap), .by = c(continent, year)), aes(x = year, y = mean_gdp, color = continent)) +
geom_step(alpha = 0.8, linewidth = 1.2) +
scale_y_continuous(labels = scales::label_dollar()) +
scale_color_colorblind() +
labs(x = "Año", y = "Renta per cápita media",
title = "Evolución en gapminder") +
theme_minimal()Evolución: serie temporal
Un gráfico de línea muy particular son las series temporales, donde en el eje X hay una variable de fecha y/o hora
Por ejemplo, vamos a cargar el siguiente dataset de Github de la evolución del precio de bitcoins cuyo separado es el espacio
data <- read_table(file = "https://raw.githubusercontent.com/holtzy/data_to_viz/master/Example_dataset/3_TwoNumOrdered.csv")
data# A tibble: 1,822 × 2
date value
<date> <dbl>
1 2013-04-28 136.
2 2013-04-29 147.
3 2013-04-30 147.
4 2013-05-01 140.
5 2013-05-02 126.
6 2013-05-03 108.
7 2013-05-04 115
8 2013-05-05 119.
9 2013-05-06 125.
10 2013-05-07 113.
# ℹ 1,812 more rowsEvolución: serie temporal
La forma más sencilla es de nuevo usar geom_line(). Con scale_x_date(date_breaks = ...) podemos indicarle los saltos en las fechas de manera sencilla.
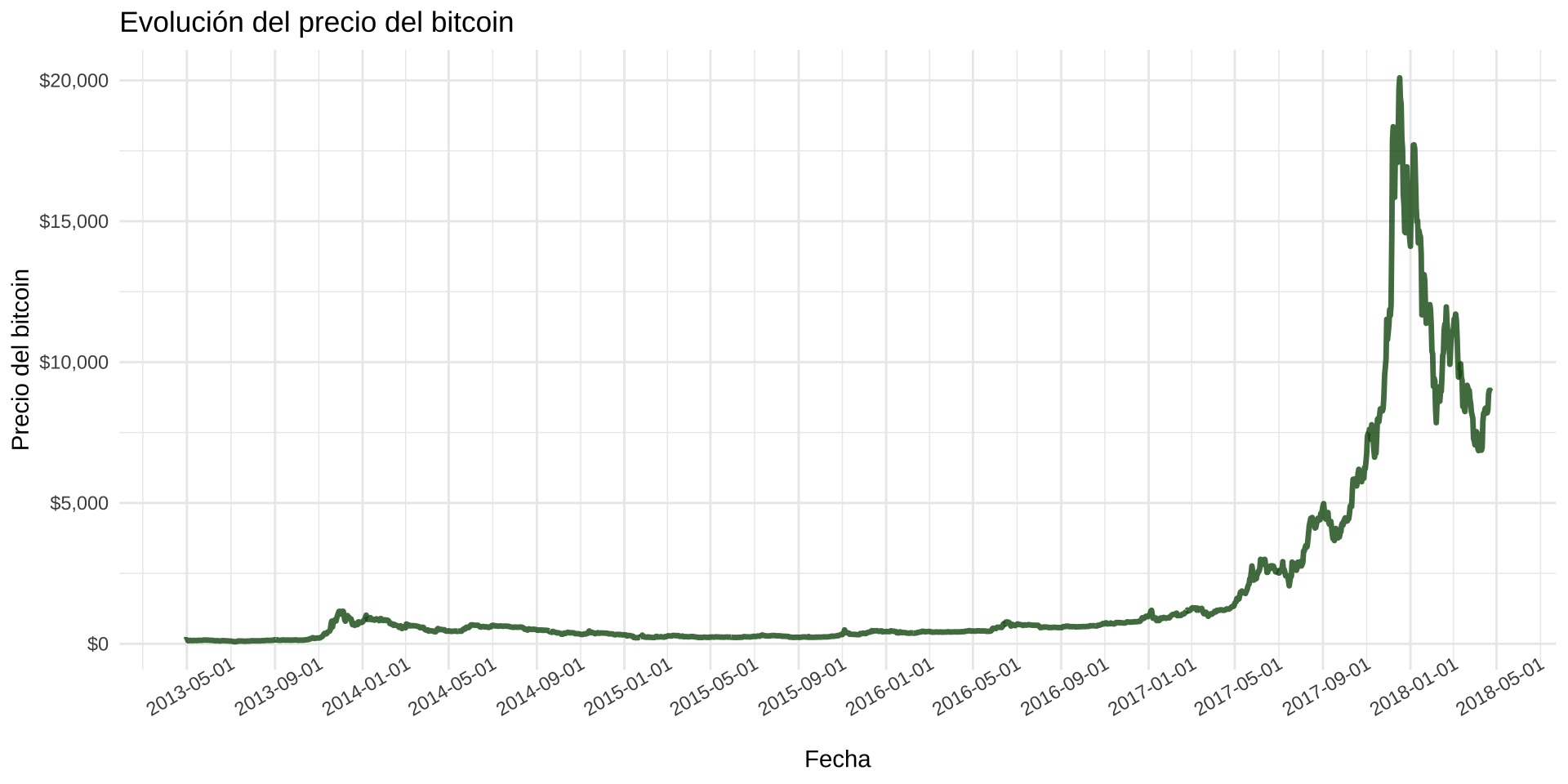
ggplot(data, aes(x = date, y = value)) +
geom_line(alpha = 0.8, color = "#145412", linewidth = 1.2) +
scale_x_date(date_breaks = "4 months") +
scale_y_continuous(labels = scales::label_dollar()) +
scale_color_colorblind() +
labs(x = "Fecha", y = "Precio del bitcoin",
title = "Evolución del precio del bitcoin") +
theme_minimal() +
theme(axis.text.x = element_text(angle = 30))Evolución: gráficos de área
Una mejora de los gráficos de línea son los gráficos de área (visualizando la curva con rellena)
La forma más sencilla es de nuevo usar geom_line() pero añadiendo la capa geom_area() (con fill en lugar de color)
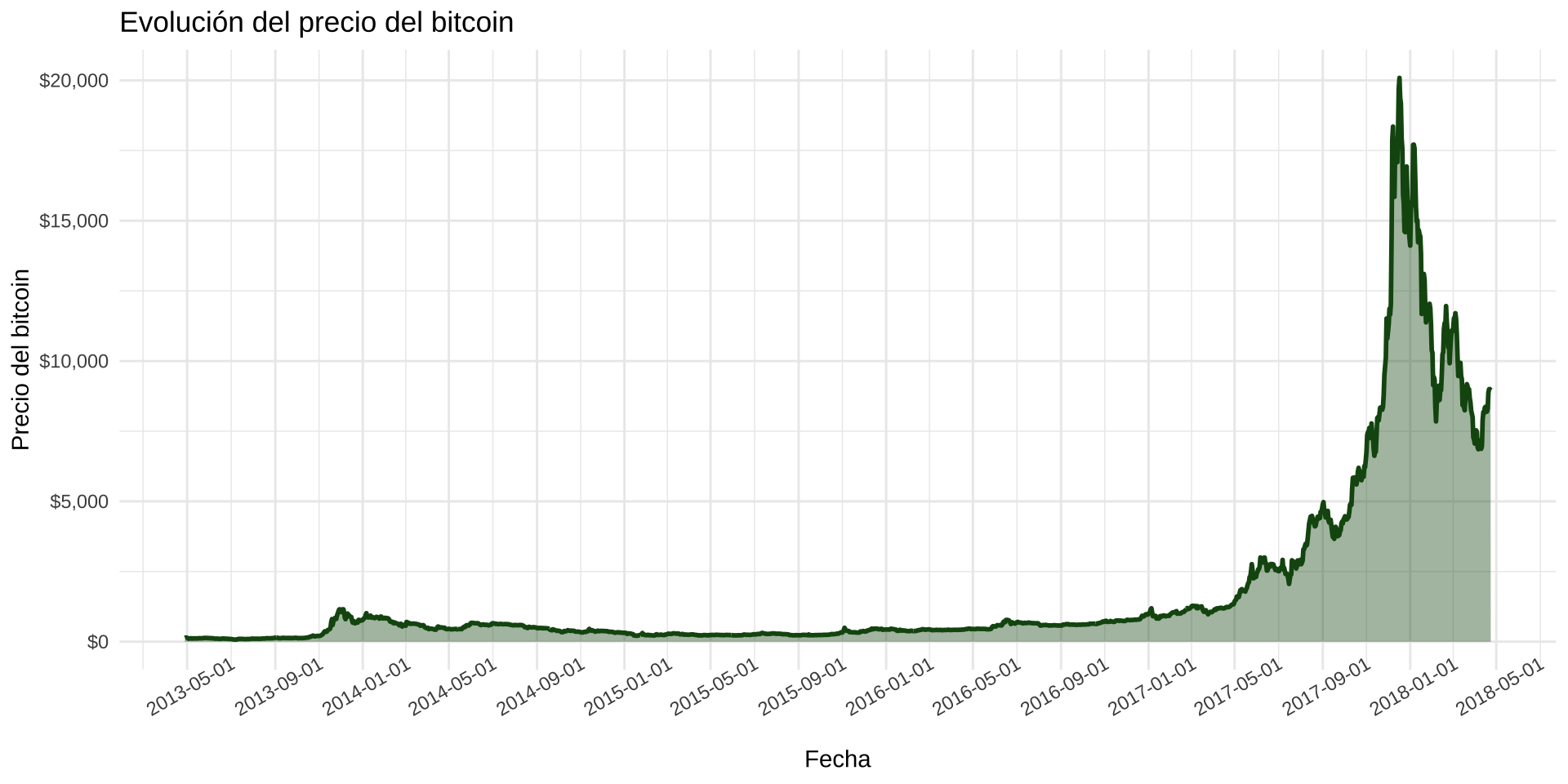
ggplot(data, aes(x = date, y = value)) +
geom_line(color = "#145412", linewidth = 1) +
geom_area(alpha = 0.4, fill = "#145412") +
scale_x_date(date_breaks = "4 months") +
scale_y_continuous(labels = scales::label_dollar()) +
labs(x = "Fecha", y = "Precio del bitcoin",
title = "Evolución del precio del bitcoin") +
theme_minimal() +
theme(axis.text.x = element_text(angle = 30))Paréntesis: interactivos
Todo gráfico ggplot podemos hacerlo interactivo guardándonos la gráfico y haciendo uso de {plotly}
gg <-
ggplot(data, aes(x = date, y = value)) +
geom_line(color = "#145412", linewidth = 1) +
geom_area(alpha = 0.4, fill = "#145412") +
scale_x_date(date_breaks = "4 months") +
scale_y_continuous(labels = scales::label_dollar()) +
labs(x = "Fecha", y = "Precio del bitcoin",
title = "Evolución del precio del bitcoin") +
theme_minimal() +
theme(axis.text.x = element_text(angle = 30))
plotly::ggplotly(gg)Evolución: áreas apiladas
Los gráficas de áreas, al igual que sucedía con los diagrmaas de barras, pueden ser de áreas apiladas, haciendo que fill() sea mapeado por aes()
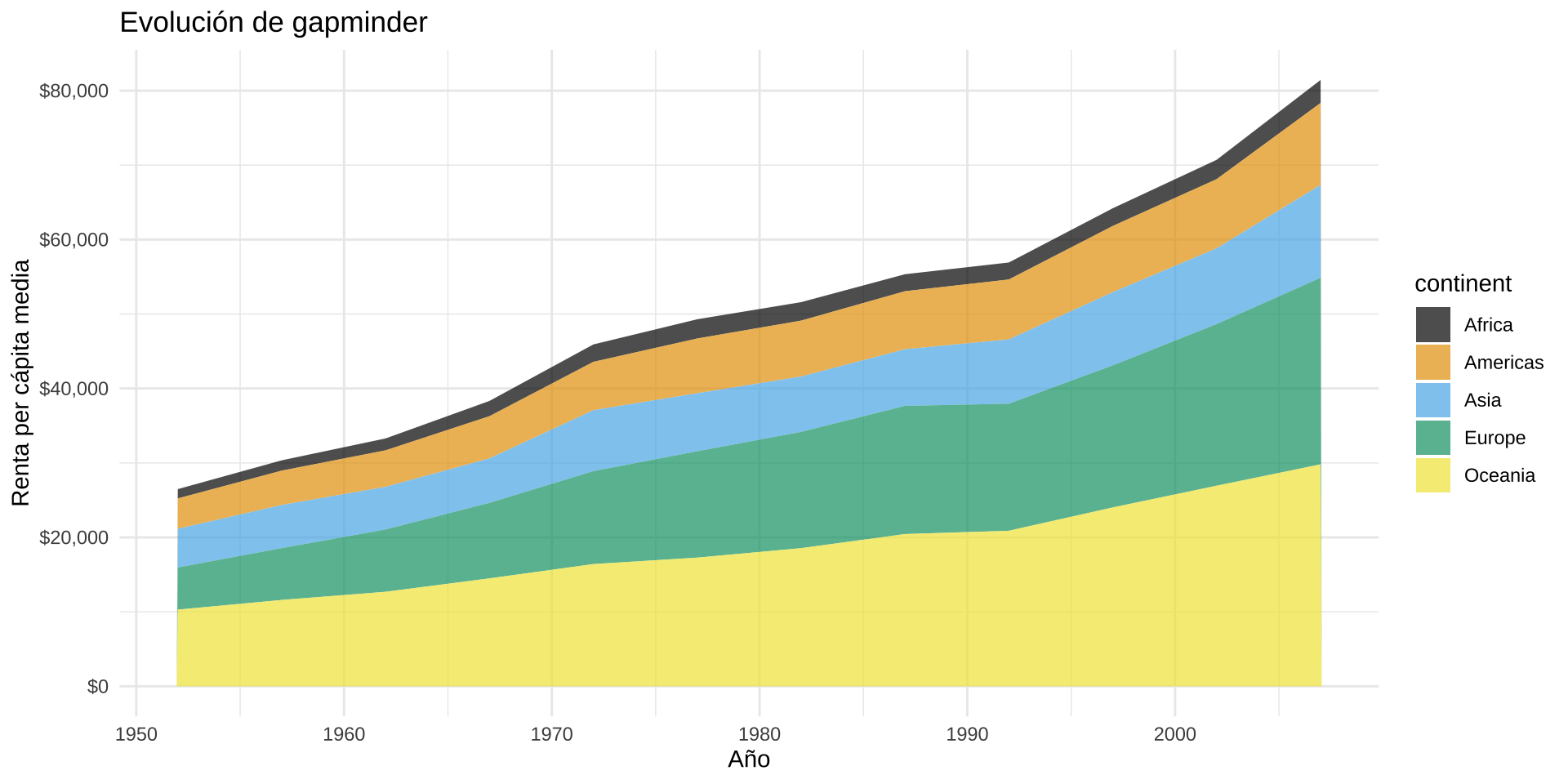
ggplot(gapminder |> summarise(mean_gdp = mean(gdpPercap), .by = c(continent, year)), aes(x = year, y = mean_gdp, fill = continent)) +
geom_area(alpha = 0.7) +
scale_y_continuous(labels = scales::label_dollar()) +
scale_fill_colorblind() +
labs(x = "Año", y = "Renta per cápita media",
title = "Evolución de gapminder") +
theme_minimal()Evolución: áreas apiladas
Los gráficas de áreas, al igual que sucedía con los diagrmaas de barras, pueden ser de áreas apiladas, haciendo que fill() sea mapeado por aes()
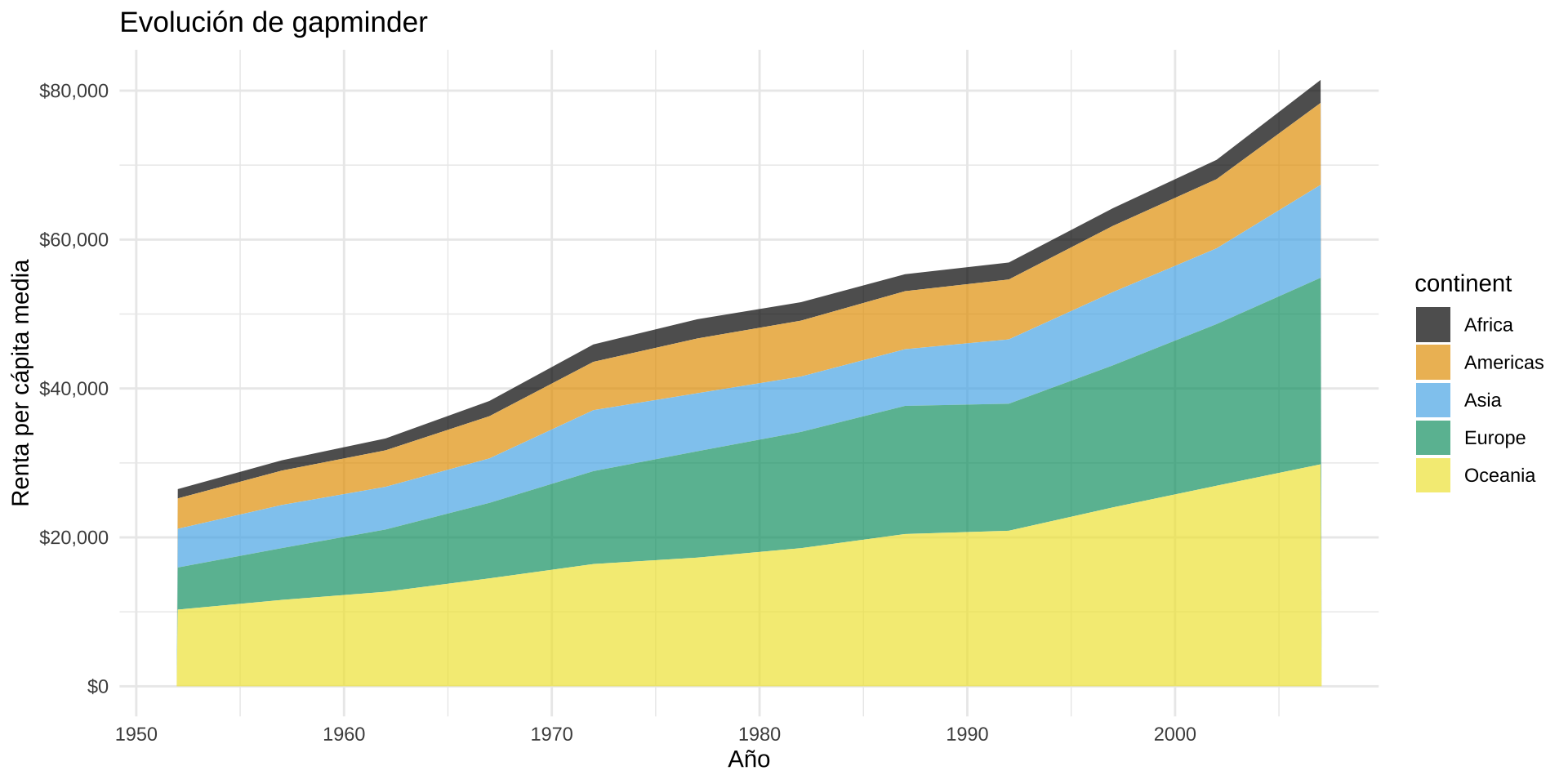
ggplot(gapminder |> summarise(mean_gdp = mean(gdpPercap), .by = c(continent, year)), aes(x = year, y = mean_gdp, fill = continent)) +
geom_area(alpha = 0.7) +
scale_y_continuous(labels = scales::label_dollar()) +
scale_fill_colorblind() +
labs(x = "Año", y = "Renta per cápita media",
title = "Evolución de gapminder") +
theme_minimal()Evolución: áreas apiladas
Haciendo una modificación en el preprocesamiento podemos hacer un gráfico de áreas apiladas en relativo
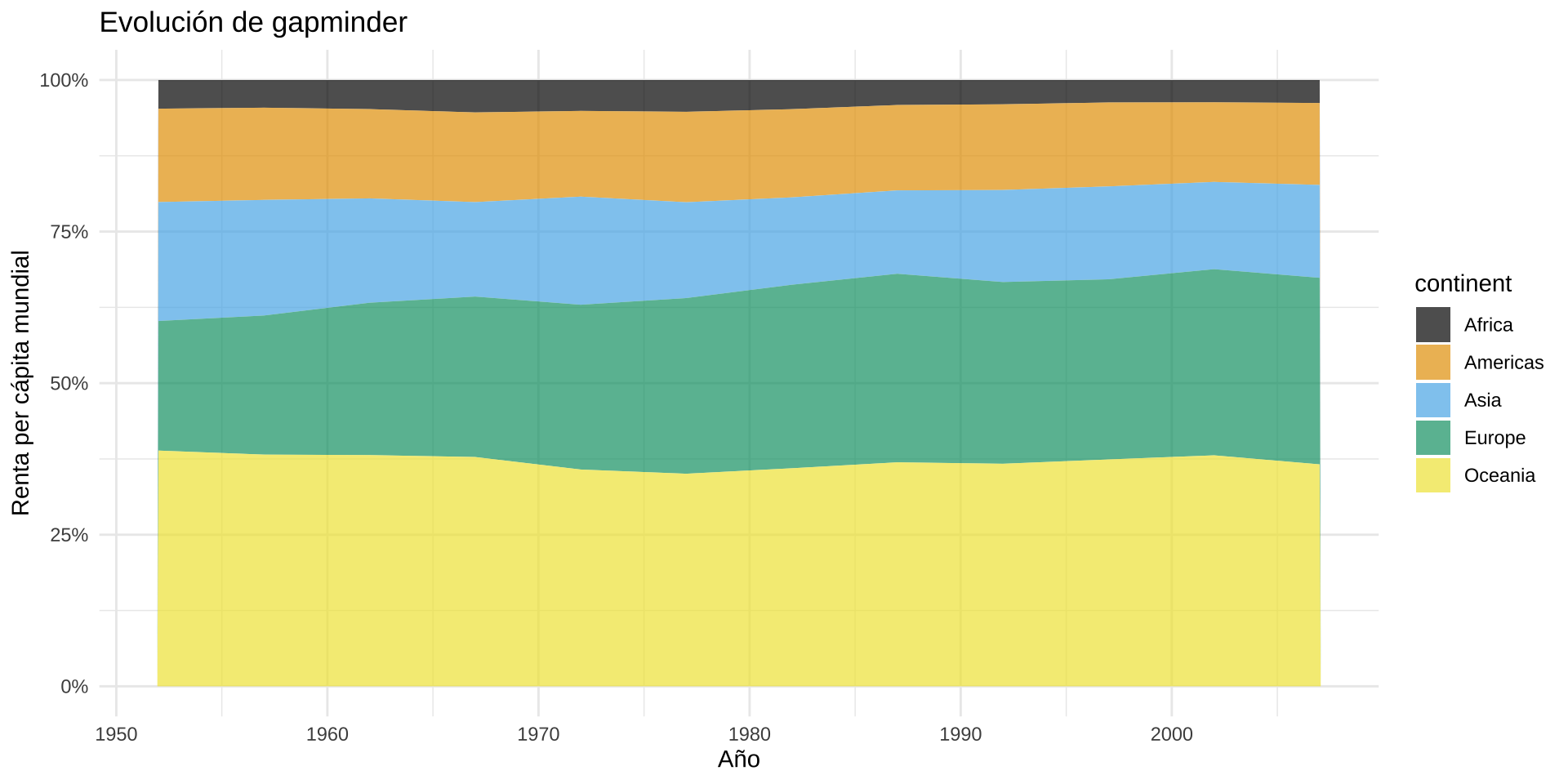
ggplot(gapminder |> summarise(mean_gdp = mean(gdpPercap), .by = c(continent, year)) |> mutate(porc = 100 * mean_gdp/sum(mean_gdp), .by = year),
aes(x = year, y = porc, fill = continent)) +
geom_area(alpha = 0.7) +
scale_y_continuous(labels = scales::label_number(suffix = "%")) +
scale_fill_colorblind() +
labs(x = "Año", y = "Renta per cápita mundial",
title = "Evolución de gapminder") +
theme_minimal()Evolución: streamcharts
Una modificación de los gráficos de áreas apiladas son los conocidos como streamcharts
En ellos las formas son más suaves que en un gráfico de área al uso, con el paquete {ggstream} (y usando geom_stream())
library(ggstream)
ggplot(gapminder |> summarise(mean_gdp = mean(gdpPercap), .by = c(continent, year)), aes(x = year, y = mean_gdp, fill = continent)) +
geom_stream(alpha = 0.7) +
scale_y_continuous(labels = scales::label_dollar()) +
scale_fill_colorblind() +
labs(x = "Año", y = "Renta per cápita mundial",
title = "Evolución de gapminder") +
theme_minimal()Evolución: streamcharts
Fíjate que por defecto lo hace en espejo, usando el eje y de manera reflejada. Con type = "ridge" lo haemos de manera apilada.
ggplot(gapminder |> summarise(mean_gdp = mean(gdpPercap), .by = c(continent, year)), aes(x = year, y = mean_gdp, fill = continent, color = continent)) +
geom_stream(alpha = 0.75, type = "ridge") +
scale_fill_colorblind() +
scale_color_colorblind() +
scale_y_continuous(labels = scales::label_dollar()) +
labs(x = "Año", y = "Renta per cápita mundial",
title = "Evolución de gapminder") +
theme_minimal()Evolución: streamcharts
Con type = "proportional" lo haemos de manera relativa
ggplot(gapminder |> summarise(mean_gdp = mean(gdpPercap), .by = c(continent, year)), aes(x = year, y = mean_gdp, fill = continent, color = continent)) +
geom_stream(alpha = 0.75, type = "proportional") +
scale_fill_colorblind() +
scale_color_colorblind() +
scale_y_continuous(labels = scales::label_dollar()) +
labs(x = "Año", y = "Renta per cápita mundial",
title = "Evolución de gapminder") +
theme_minimal()🐣 Caso práctico 16: visualizando Netflix
Visualizaremos el número de películas y series de instituto que se han estrenado en Netflix en cada año. Los datos provienen originalmente de Kaggle, y contienen las películas y series de Netflix hasta enero de 2021.
netflix <-
read_csv('https://raw.githubusercontent.com/elartedeldato/datasets/main/netflix_titles.csv')
netflix# A tibble: 7,787 × 12
show_id type title director cast country date_added release_year rating
<chr> <chr> <chr> <chr> <chr> <chr> <chr> <dbl> <chr>
1 s1 TV Show 3% <NA> João… Brazil August 14… 2020 TV-MA
2 s2 Movie 7:19 Jorge Mic… Demi… Mexico December … 2016 TV-MA
3 s3 Movie 23:59 Gilbert C… Tedd… Singap… December … 2011 R
4 s4 Movie 9 Shane Ack… Elij… United… November … 2009 PG-13
5 s5 Movie 21 Robert Lu… Jim … United… January 1… 2008 PG-13
6 s6 TV Show 46 Serdar Ak… Erda… Turkey July 1, 2… 2016 TV-MA
7 s7 Movie 122 Yasir Al … Amin… Egypt June 1, 2… 2019 TV-MA
8 s8 Movie 187 Kevin Rey… Samu… United… November … 1997 R
9 s9 Movie 706 Shravan K… Divy… India April 1, … 2019 TV-14
10 s10 Movie 1920 Vikram Bh… Rajn… India December … 2008 TV-MA
# ℹ 7,777 more rows
# ℹ 3 more variables: duration <chr>, listed_in <chr>, description <chr>🐣 Caso práctico 16: visualizando Netflix
- Paso 1: piensa como filtrar las películas y series que van sobre un instituto.
- Paso 2: tras dicho filtro, añade el año en el que se estrenó y elimina aquellas sin año conocido.
- Paso 3: obtén el número de series por año
🐣 Caso práctico 16: visualizando Netflix
- Paso 4: replica el siguiente gráfico
Código
netflix_resumen <-
netflix |>
filter(str_detect(toupper(description), "HIGH SCHOOL")) |>
mutate(year_added = year(mdy(date_added))) |>
drop_na(year_added) |>
group_by(year_added) |>
count() |> ungroup()
ggplot(netflix_resumen, aes(x = year_added, y = n)) +
geom_col(fill = "red") +
scale_x_continuous(breaks = netflix_resumen$year_added) +
labs(title = "NETFLIX",
subtitle = "Películas y series de instituto",
x = "Año de estreno", y = "Cantidad")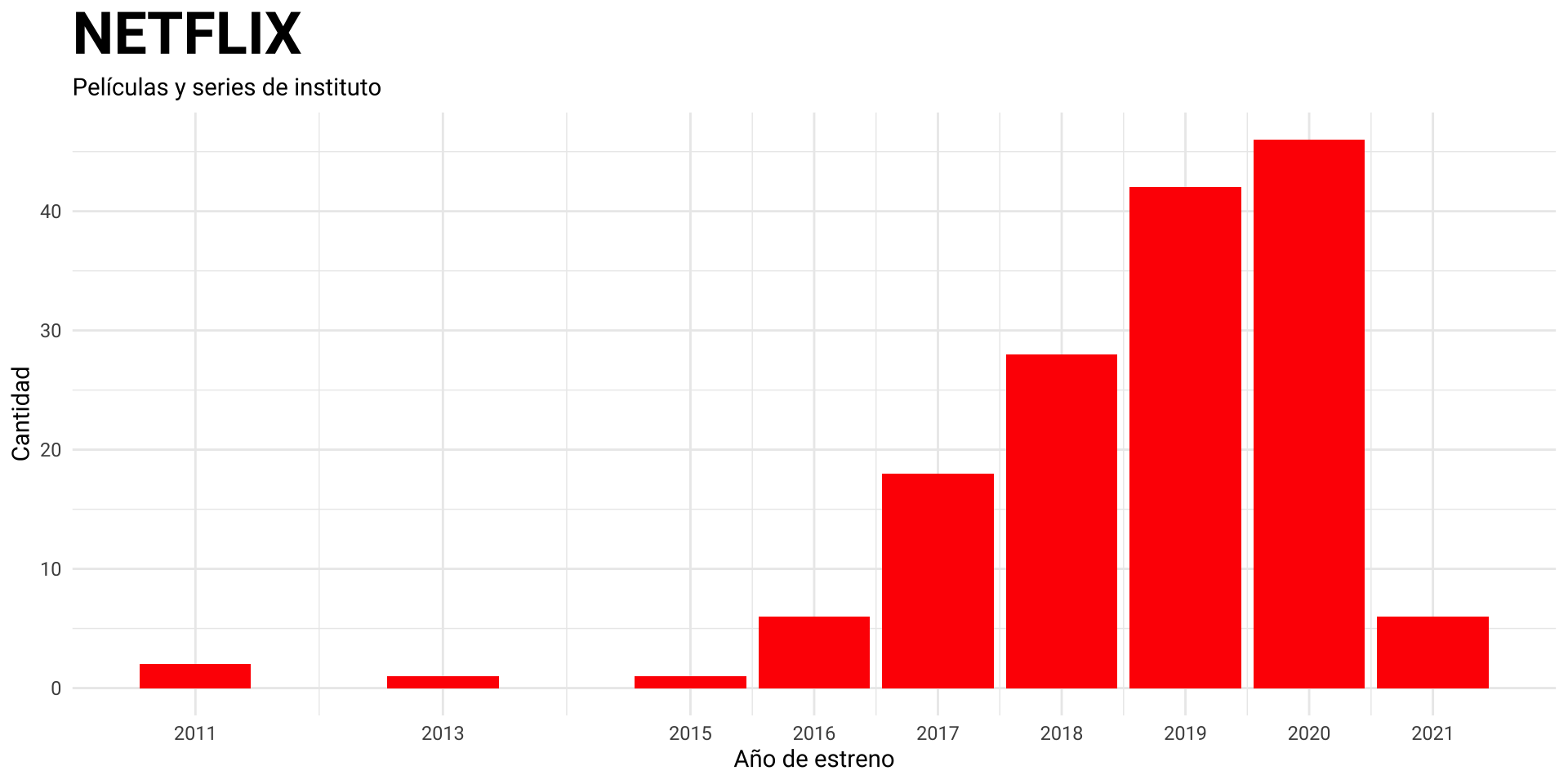
🐣 Caso práctico 16: visualizando Netflix
- Paso 5: replica el siguiente gráfico sabiendo que la fuente del título es “Bebas Neue” y la del subtítulo “Permanent Marker”
Código
library(sysfonts)
library(showtext)
font_add_google(family = "Bebas Neue",
name = "Bebas Neue")
font_add_google(family = "Permanent Marker",
name = "Permanent Marker")
showtext_auto()
ggplot(netflix_resumen, aes(x = year_added, y = n)) +
geom_col(fill = "red") +
scale_x_continuous(breaks = netflix_resumen$year_added) +
labs(title = "NETFLIX",
subtitle = "Películas y series de instituto",
x = "Año de estreno", y = "Cantidad") +
theme_minimal() +
theme(legend.position = "none",
plot.title = element_text(family = "Bebas Neue",
color = "red", size = 80),
plot.subtitle = element_text(family = "Permanent Marker",
size = 21, color = "black"))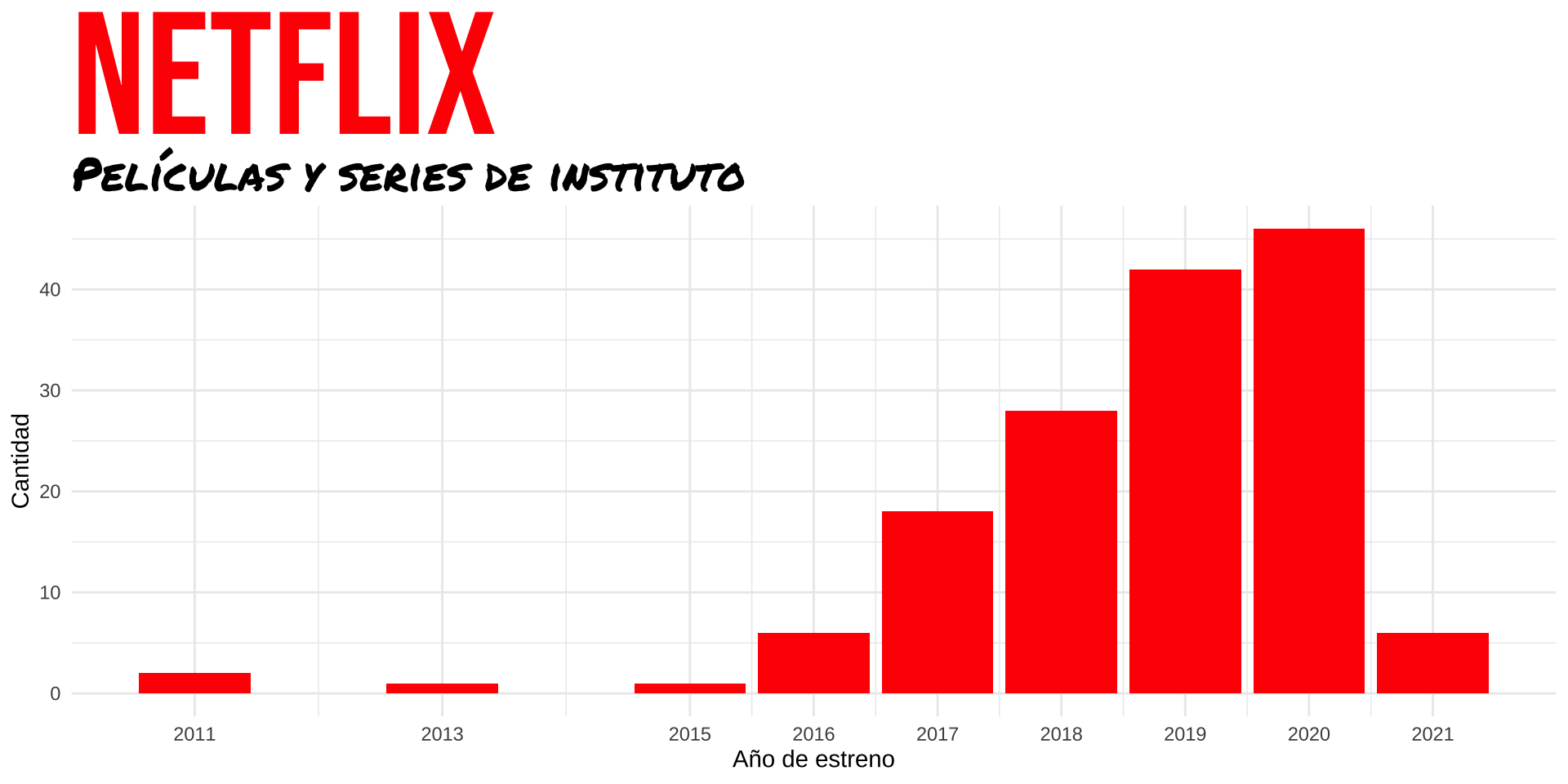
🐣 Caso práctico 16: visualizando Netflix
- Paso 6: replica el siguiente gráfico sabiendo que la fuente de los ejes es “Permanent Marker” y busca la función
annotate()
Código
ggplot(netflix_resumen, aes(x = year_added, y = n)) +
geom_col(fill = "red") +
scale_x_continuous(breaks = netflix_resumen$year_added) +
labs(title = "NETFLIX",
subtitle = "Películas y series de instituto",
x = "Año de estreno", y = "Cantidad") +
theme_void() +
theme(plot.margin = margin(t = 4, r = 4, b = 4, l = 8, "pt"),
legend.position = "none",
plot.title = element_text(family = "Bebas Neue",
color = "red", size = 80),
plot.subtitle = element_text(family = "Permanent Marker",
size = 21, color = "white"),
axis.text =
element_text(size = 15, family = "Permanent Marker",
color = "white"),
panel.background = element_rect(fill = "black"),
plot.background = element_rect(fill = "black",
color = "black"),
panel.grid.major.y =
element_line(linewidth = 0.1, color = "white")) +
annotate("text", label = "(hasta enero)",
x = 2021, y = 11, hjust = 0.3, vjust = 0, family = "Permanent Marker", size = 5, color='white', angle = 20) +
annotate("curve", x = 2021, y = 9, xend = 2021, yend = 5,
color = "white")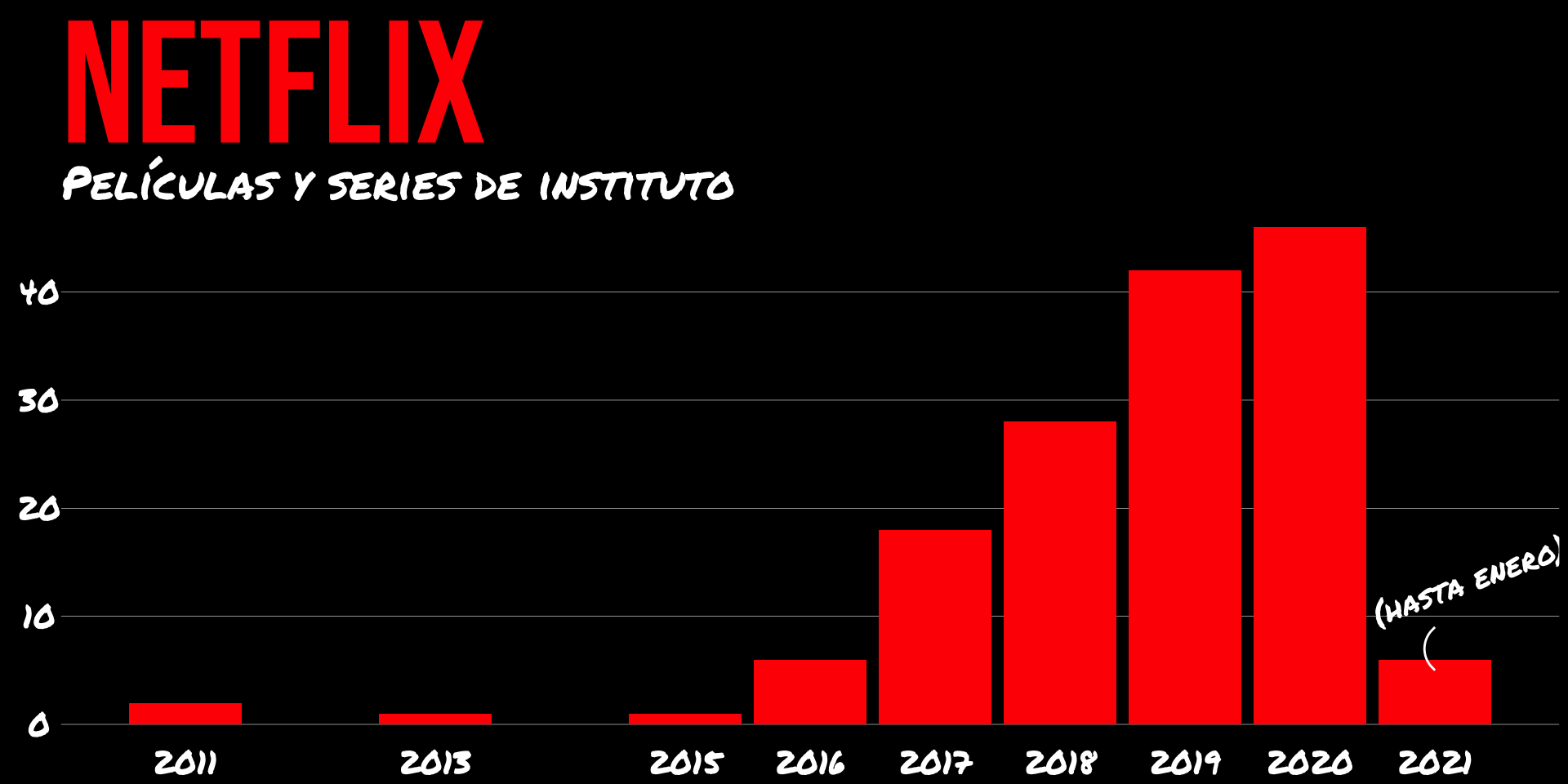
Clase 6: relacionando datos
Joins
Relacionando datos
Al trabajar con datos no siempre tendremos la información en una sola tabla y a veces nos interesará cruzar la información de distintas fuentes.
Para ello usaremos un clásico de todo lenguaje que maneja datos: los famosos join, una herramienta que nos va a permitir cruzar una o variables tablas, haciendo uso de una columna identificadora de cada una de ellas (por ejemplo, imagina que cruzamos datos de hacienda y de antecedentes penales, haciendo join por la columna DNI).
Relacionando datos
inner_join(): solo sobreviven los registros con id en ambas tablas.full_join(): mantiene todos los registros de ambas tablas.left_join(): mantiene todos los registros de la primera tabla, y busca cuales tienen id también en la segunda (en caso de no tenerlo se rellena con NA los campos de la 2ª tabla).right_join(): mantiene todos los registros de la segunda tabla, y busca cuales tienen id también en la primera.

Relacionando datos
Vamos a probar los distintos joins con un ejemplo sencillo
Left join
Imagina que queremos incorporar a tb_1 la información de la tabla_2, identificando los registros por la columna key (indicando con by = "key" la columna por la que tiene que cruzar): queremos mantener todos los registros de la primera tabla y buscar cuales tienen id (mismo valor en key) también en la segunda tabla.
Left join
# A tibble: 3 × 3
key val_x val_y
<dbl> <chr> <chr>
1 1 x1 y1
2 2 x2 y2
3 3 x3 <NA> Fíjate que los registros de la primera cuya key no ha encontrado en la segunda les ha dado el valor de ausente.
Right join
El right_join() realizará la operación contraria: vamos ahora a incorporar a tb_2 la información de la tabla_2, identificando los registros por la columna key (indicando con by = "key" la columna por la que tiene que cruzar): queremos mantener todos los registros de la segunda y buscar cuales tienen id (mismo valor en key) también en la primera tabla.
Right join
# A tibble: 3 × 3
key val_x val_y
<dbl> <chr> <chr>
1 1 x1 y1
2 2 x2 y2
3 4 <NA> y3 Fíjate que ahora los registros de la segunda cuya key no ha encontrado en la primera son los que les ha dado el valor de ausente.
Claves y sufijos
Las columnas clave que usaremos para el cruce no siempre se llamarán igual.
by = c("key_2" = "key_2"): le indicaremos en qué columna de cada tabla están las claves por las que vamos a cruzar.
Claves y sufijos
Además podemos cruzar por varias columnas a la vez (interpretará como igual registro aquel que tenga el conjunto de claves igual), con by = c("var1_t1" = "var1_t2", "var2_t1" = "var2_t2", ...). Modifiquemos el ejemplo anterior
Claves y sufijos
También podría suceder que al cruzar dos tablas, haya columnas de valores que se llamen igual
Claves y sufijos
Dicho sufijo podemos especificárselo en el argumento opcional suffix = ..., que nos permita distinguir las variables de una tabla y de otra.
Full join
Los dos anteriores casos forman lo que se conoce como outer joins: cruces donde se mantienen observaciones que salgan en al menos una tabla. El tercer outer join es el conocido como full_join() que nos mantendrá las observaciones de ambas tablas, añadiendo las filas que no casen con la otra tabla.
Inner join
Frente a los outer join está lo que se conoce como inner join, con inner_join(): un cruce en el que solo se mantienen las observaciones que salgan en ambas tablas, solo mantiene aquellos registros matcheados.
Inner join
Fíjate que en términos de registros, inner_join si es conmutativa, nos da igual el orden de las tablas: lo único que cambia es el orden de las columnas que añade.
Anti/semi join
Por último tenemos dos herramientas interesantes para filtrar (no cruzar) registros: semi_join() y anti_join(). El semi join nos deja en la primera tabla los registros que cuya clave está también en la segunda (como un inner join pero sin añadir la info de la segunda tabla). Y el segundo, los anti join, hace justo lo contrario (aquellos que no están).
💻 Tu turno
Intenta realizar los siguientes ejercicios sin mirar las soluciones
Para los ejercicios usaremos las tablas disponibles en el paquete {nycflights13} (echa un vistazo antes)
- airlines: nombre de aerolíneas (con su abreviatura).
- airports: datos de aeropuertos (nombres, longitud, latitud, altitud, etc).
- flights: datos de vuelos.
- planes: datos de los aviones.
- weather: datos meteorológicos horarios de las estaciones LGA, JFK y EWR.
💻 Tu turno
Intenta realizar los siguientes ejercicios sin mirar las soluciones
📝 Del paquete {nycflights13} cruza la tabla flights con airlines. Queremos mantener todos los registros de vuelos, añadiendo la información de las aerolíneas a la tabla de aviones.
📝 A la tabla obtenida del cruce del apartado anterior, cruza después con los datos de los aviones en planes, pero incluyendo solo aquellos vuelos de los que tengamos información de sus aviones (y viceversa).
📝 Repite el ejercicio anterior pero conservando ambas variables year (en una es el año del vuelo, en la otra es el año de construcción del avión), y distinguiéndolas entre sí
📝 Al cruce obtenido del ejercicio anterior incluye la longitud y latitud de los aeropuertos en airports, distinguiendo entre la latitud/longitud del aeropuerto en destino y en origen.
📝 Filtra de airports solo aquellos aeropuertos de los que salgan vuelos. Repite el proceso filtrado solo aquellos a los que lleguen vuelos
¡Gracias!
Pendiente de añadir: manejo de listas, mapas, introducción a la modelización
Javier Álvarez Liébana • Curso para el Ayuntamiento de Madrid • curso 2023-2024