estaturas <- c(150, 160, 170, 180)
pesos <- c(63, 70, 85, 95)
datos_matriz <- cbind(estaturas, pesos)
datos_matriz estaturas pesos
[1,] 150 63
[2,] 160 70
[3,] 170 85
[4,] 180 95Seminario de tidyverse y Quarto para docencia e investigación
Dejad vuestros bucles a un lado
.png)
Entender los conceptos básicos de tidyverse desde cero → aprender programar funcional (sin bucles)
Utilidad de tidyverse → flujos de trabajo reproducibles, legibles, transparentes y mantenibles
Introducción a Quarto → elaboración de material docente
Introducción a Github en RStudio
En el menú de las diapositivas (abajo a la izquierda) tienes una opción para descargarlas en pdf en Tools (consejo: no lo hagas hasta el final del curso ya que irán modificándose)
Código de las diapositivas: repositorio Github
Resúmenes de paquetes: chuletas de los paquetes en formato .pdf
De la misma manera que en el ordenador solemos trabajar de manera ordenada por carpetas, en RStudio podemos hacer lo mismo para trabajar de manera eficaz creando proyectos.
Un proyecto será una «carpeta» dentro de RStudio, de manera que nuestro directorio raíz automáticamente será la propia carpeta de proyecto (pudiendo pasar de un proyecto a otro con el menu superior derecho).
Podemos crear uno en una carpeta nueva o en una carpeta ya existente.
Matrices y data.frames
Cuando analizamos datos solemos tener varias variables de cada individuo: necesitamos una «tabla» que las recopile. La opción más inmediata son las matrices: concatenación de variables del mismo tipo e igual longitud.
También podemos realizar operaciones por columnas/filas sin recurrir a bucles con la función apply(), y le indicaremos como argumentos
Para poder trabajar con variables de distinto tipo tenemos lo que se conoce como data.frame: concatenación de variables de igual longitud pero pueden ser de tipo distinto.
Dado que un data.frame es ya una «base de datos» las variables no son meros vectores matemáticos: tienen un significado y podemos (debemos) ponerles nombres
library(lubridate)
tabla <- data.frame("edad" = 25:27, "estado" = c(TRUE, NA, FALSE),
"nombre" = c("javi", "sandra", "carlos"),
"f_nacimiento" = as_date(c("1989-09-10", "1996-06-12", "1980-11-27")))
tabla edad estado nombre f_nacimiento
1 25 TRUE javi 1989-09-10
2 26 NA sandra 1996-06-12
3 27 FALSE carlos 1980-11-27Si queremos acceder a sus elementos, podemos como en las matrices (aunque no es recomendable): ahora tenemos dos índices (filas y columnas, dejando libre la que no usemos)
edad estado nombre f_nacimiento
2 26 NA sandra 1996-06-12[1] "javi" "sandra" "carlos"[1] 26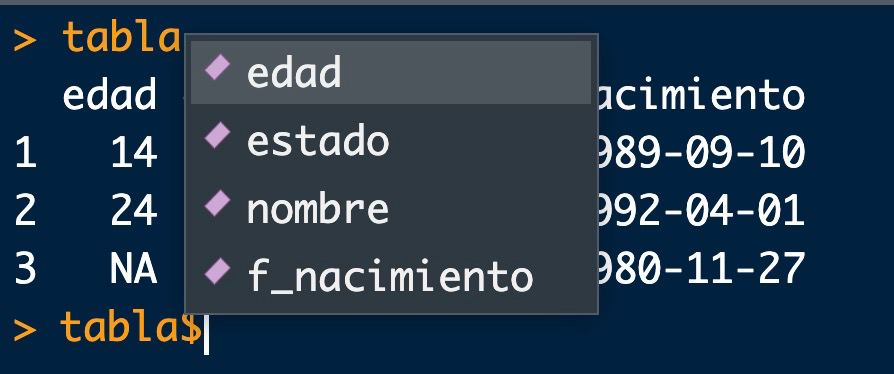
También tiene ventajas de una «base» de datos : podemos aceder a las variables por su nombre (recomendable ya que las variables pueden cambiar de posición), poniendo el nombre de la tabla seguido del símbolo $ (con el tabulador, nos aparecerá un menú de columnas a elegir)
Las tablas en formato data.frame tienen algunas limitaciones
La principal es que no permite la definición “al vuelo”: imagina que definimos una base de datos con estaturas y pesos, y queremos una tercera variable con el IMC
Error in data.frame(estatura = c(1.7, 1.8, 1.6), peso = c(80, 75, 70), : objeto 'peso' no encontradotabla <- tibble("estatura" = c(1.7, 1.8, 1.6), "peso" = c(80, 75, 70),
"IMC" = peso / (estatura^2))
tabla# A tibble: 3 × 3
estatura peso IMC
<dbl> <dbl> <dbl>
1 1.7 80 27.7
2 1.8 75 23.1
3 1.6 70 27.3Las tablas en formato tibble nos permitirá una gestión más ágil, eficiente y coherente de los datos, con 4 ventajas principales:
tribble()Consejo
El paquete {datapasta} nos permite copiar y pegar tablas de páginas web y documentos sencillos
Nuestra base de datos: tibble. Tidydata: un multiverso de datos limpios
Si conoces algún otro lenguaje de programación (o tienes gente cercana que programa) te extrañará que no vayamos a usar conceptos habituales como
Bucles for: repetir un código un número fijo de iteraciones.
Bucles while: repetir un código hasta que se cumpla una condición
Estructuras if-else: estructuras de control para decidir por donde camina el código en función del valor de las variables.
Y es que con tidyverse, en la mayoría de ocasiones vamos a poder evitarlas (en especial los bucles)

{tidyverse} es un «universo» de paquetes para garanatizar un flujo de trabajo (de inicio a fin) eficiente, coherente y lexicográficamente sencillo de entender, basado en la idea de que nuestros datos están limpios y ordenados (tidy)

{tibble}: optimizando data.frame{tidyr}: limpieza de datos{readr}: carga datos rectangulares (.csv){dplyr}: gramática para depurar{stringr}: manejo de textos{ggplot2}: visualización de datos{tidymodels}: modelización/predicciónTambién tenemos los paquetes {purrr} para el manejo de listas, {forcast} para cualitativas, {lubridate} para fechas, {readxl} para importar archivos .xls y .xlsx, {rvest} para web scraping y {rmarkdown} para comunicar resultados.

{tibble}: optimizando data.frame{tidyr}: limpieza de datos{readr}: carga datos rectangulares (.csv){dplyr}: gramática para depurar{stringr}: manejo de textos{ggplot2}: visualización de datos{tidymodels}: modelización/predicciónTambién tenemos los paquetes {purrr} para el manejo de listas, {forcast} para cualitativas, {lubridate} para fechas, {readxl} para importar archivos .xls y .xlsx, {rvest} para web scraping y {rmarkdown} para comunicar resultados.
Tidy datasets are all alike, but every messy dataset is messy in its own way (Hadley Wickham, Chief Scientist en RStudio)
TIDYVERSE
El universo de paquetes {tidyverse} se basa en la idea introducido por Hadley Wickham (el Dios al que rezo) de estandarizar el formato los datos para
Lo primero por tanto será entender qué son los conjuntos tidydata ya que todo {tidyverse} se basa en que los datos están estandarizados.

En {tidyverse} será clave el operador pipe (tubería) definido como |> (ctrl+shift+M): será una tubería que recorre los datos y los transforma.
En R base, si queremos aplicar tres funciones first(), second() y third() en orden, sería
Apunte importante
Desde la versión 4.1.0 de R disponemos de |>, un pipe nativo disponible fuera de tidyverse, sustituyendo al antiguo pipe %>% que dependía del paquete {magrittr} (bastante problemático).
La principal ventaja es que el código sea muy legible (casi literal) pudiendo hacer grandes operaciones con los datos con apenas código.
¿Pero qué aspecto tienen los datos no tidy? Vamos a cargar la tabla table4a del paquete {tidyr} (ya lo tenemos cargado del entorno tidyverse).
# A tibble: 3 × 3
country `1999` `2000`
<chr> <dbl> <dbl>
1 Afghanistan 745 2666
2 Brazil 37737 80488
3 China 212258 213766
¿Qué puede estar fallando?
❎ Cada fila representa dos observaciones (1999 y 2000) → las columnas 1999 y 2000 en realidad deberían ser en sí valores de una variable y no nombres de columnas.
Incluiremos una nueva columna que nos guarde el año y otra que guarde el valor de la variable de interés en cada uno de esos años. Y lo haremos con la función pivot_longer(): pivotaremos la tabla a formato long:
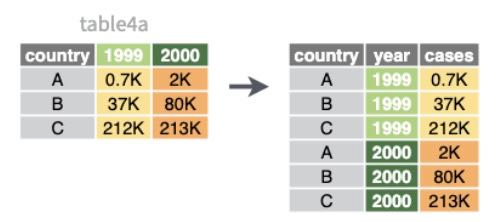
cols: nombre de las variables a pivotarnames_to: nombre de la nueva variable a la quemandamos la cabecera de la tabla (los nombres).values_to: nombre de la nueva variable a la que vamos a mandar los datos.Veamos otro ejemplo con la tabla table2
# A tibble: 12 × 4
country year type count
<chr> <dbl> <chr> <dbl>
1 Afghanistan 1999 cases 745
2 Afghanistan 1999 population 19987071
3 Afghanistan 2000 cases 2666
4 Afghanistan 2000 population 20595360
5 Brazil 1999 cases 37737
6 Brazil 1999 population 172006362
7 Brazil 2000 cases 80488
8 Brazil 2000 population 174504898
9 China 1999 cases 212258
10 China 1999 population 1272915272
11 China 2000 cases 213766
12 China 2000 population 1280428583
¿Qué puede estar fallando?
# A tibble: 12 × 4
country year type count
<chr> <dbl> <chr> <dbl>
1 Afghanistan 1999 cases 745
2 Afghanistan 1999 population 19987071
3 Afghanistan 2000 cases 2666
4 Afghanistan 2000 population 20595360
5 Brazil 1999 cases 37737
6 Brazil 1999 population 172006362
7 Brazil 2000 cases 80488
8 Brazil 2000 population 174504898
9 China 1999 cases 212258
10 China 1999 population 1272915272
11 China 2000 cases 213766
12 China 2000 population 1280428583❎ Cada observación está dividido en dos filas → los registros con el mismo año deberían ser el mismo
Lo que haremos será lo opuesto: con pivot_wider() ensancharemos la tabla
# A tibble: 6 × 4
country year cases population
<chr> <dbl> <dbl> <dbl>
1 Afghanistan 1999 745 19987071
2 Afghanistan 2000 2666 20595360
3 Brazil 1999 37737 172006362
4 Brazil 2000 80488 174504898
5 China 1999 212258 1272915272
6 China 2000 213766 1280428583Veamos otro ejemplo con la tabla table3
# A tibble: 6 × 3
country year rate
<chr> <dbl> <chr>
1 Afghanistan 1999 745/19987071
2 Afghanistan 2000 2666/20595360
3 Brazil 1999 37737/172006362
4 Brazil 2000 80488/174504898
5 China 1999 212258/1272915272
6 China 2000 213766/1280428583¿Qué puede estar fallando?
❎ Cada celda contiene varios valores
Lo que haremos será hacer uso de la función separate() para mandar separar cada valor a una columna diferente.
# A tibble: 6 × 4
country year cases pop
<chr> <dbl> <chr> <chr>
1 Afghanistan 1999 745 19987071
2 Afghanistan 2000 2666 20595360
3 Brazil 1999 37737 172006362
4 Brazil 2000 80488 174504898
5 China 1999 212258 1272915272
6 China 2000 213766 1280428583# A tibble: 6 × 4
country year cases pop
<chr> <dbl> <chr> <chr>
1 Afghanistan 1999 745 19987071
2 Afghanistan 2000 2666 20595360
3 Brazil 1999 37737 172006362
4 Brazil 2000 80488 174504898
5 China 1999 212258 1272915272
6 China 2000 213766 1280428583Fíjate que los datos, aunque los ha separado, los ha mantenido como texto cuando en realidad deberían ser variables numéricas. Para ello podemos añadir el argumento opcional convert = TRUE
# A tibble: 6 × 4
country year cases pop
<chr> <dbl> <int> <int>
1 Afghanistan 1999 745 19987071
2 Afghanistan 2000 2666 20595360
3 Brazil 1999 37737 172006362
4 Brazil 2000 80488 174504898
5 China 1999 212258 1272915272
6 China 2000 213766 1280428583Veamos el último ejemplo con la tabla table5
# A tibble: 6 × 4
country century year rate
<chr> <chr> <chr> <chr>
1 Afghanistan 19 99 745/19987071
2 Afghanistan 20 00 2666/20595360
3 Brazil 19 99 37737/172006362
4 Brazil 20 00 80488/174504898
5 China 19 99 212258/1272915272
6 China 20 00 213766/1280428583¿Qué puede estar fallando?
❎ Tenemos mismos valores divididos en dos columnas
Usaremos unite() para unir los valores de siglo y año en una misma columna
# A tibble: 6 × 3
country year_completo rate
<chr> <chr> <chr>
1 Afghanistan 1999 745/19987071
2 Afghanistan 2000 2666/20595360
3 Brazil 1999 37737/172006362
4 Brazil 2000 80488/174504898
5 China 1999 212258/1272915272
6 China 2000 213766/1280428583Intenta realizar los siguientes ejercicios sin mirar las soluciones
📝 Echa un vistazo a la tabla table4b del paquete {tidyr}. ¿Es tidydata? En caso negativo, ¿qué falla? ¿Cómo convertirla a tidy data en caso de que no lo sea ya?
📝 Echa un vistazo a la tabla relig_income del paquete {tidyr}. ¿Es tidydata? En caso negativo, ¿qué falla? ¿Cómo convertirla a tidy data en caso de que no lo sea ya?
📝 Echa un vistazo a la tabla billboard del paquete {tidyr}. ¿Es tidydata? En caso negativo, ¿qué falla? ¿Cómo convertirla a tidy data en caso de que no lo sea ya?
Operaciones con filas en tidyverse

{tibble}: optimizando data.frame{tidyr}: limpieza de datos{readr}: carga datos rectangulares (.csv){dplyr}: gramática para depurar{stringr}: manejo de textos{ggplot2}: visualización de datos{tidymodels}: modelización/predicciónTambién tenemos los paquetes {purrr} para el manejo de listas, {forcast} para cualitativas, {lubridate} para fechas, {readxl} para importar archivos .xls y .xlsx, {rvest} para web scraping y {rmarkdown} para comunicar resultados.
Dentro de {tidyverse} usaremos el paquete {dplyr} para el preprocesamiento y depuración de datos de datos.
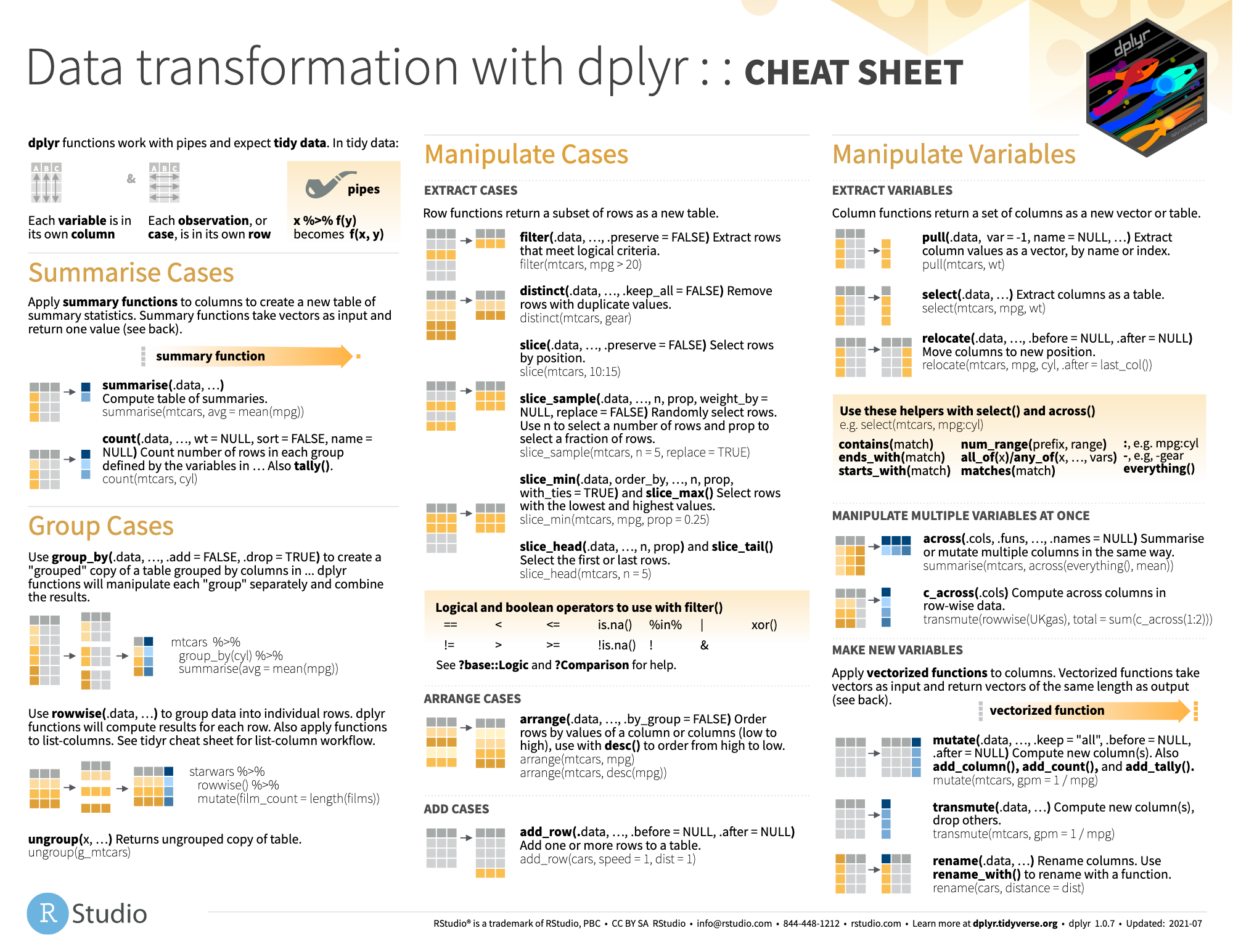
La idea es que el código sea legible, como si fuese una lista de instrucciones que al leerla nos diga de manera muy evidente lo que está haciendo.
Toda la depuración que vamos a realizar es sobre la hipótesis de que nuestros datos están en tidydata

Recuerda que en {tidyverse} será clave el operador pipe (tubería) definido como |> (ctrl+shift+M): será una tubería que recorre los datos y los transforma.
El más simple es cuando filtramos registros en base a alguna condición lógica: con filter() se seleccionarán solo individuos que cumplan ciertas condiciones (muestreo no aleatorio por condiciones)
==, !=: igual o distinto que (|> filter(variable == "a"))>, <: mayor o menor que (|> filter(variable < 3))>=, <=: mayor o igual o menor o igual que (|> filter(variable >= 5))%in%: valores pertenencen a un listado de opciones (|> filter(variable %in% c("azul", "verde")))between(variable, val1, val2): si los valores (continuos) caen dentro de un rango de valores (|> filter(between(variable, 160, 180)))¿Cómo harías para… filtrar los personajes de ojos marrones?
¿Qué tipo de variable es? –> La variable eye_color es cualitativa así que está representada por textos
# A tibble: 21 × 14
name height mass hair_color skin_color eye_color birth_year sex gender
<chr> <int> <dbl> <chr> <chr> <chr> <dbl> <chr> <chr>
1 Leia Or… 150 49 brown light brown 19 fema… femin…
2 Biggs D… 183 84 black light brown 24 male mascu…
3 Han Solo 180 80 brown fair brown 29 male mascu…
4 Yoda 66 17 white green brown 896 male mascu…
5 Boba Fe… 183 78.2 black fair brown 31.5 male mascu…
6 Lando C… 177 79 black dark brown 31 male mascu…
7 Arvel C… NA NA brown fair brown NA male mascu…
8 Wicket … 88 20 brown brown brown 8 male mascu…
9 Quarsh … 183 NA black dark brown 62 <NA> <NA>
10 Shmi Sk… 163 NA black fair brown 72 fema… femin…
# ℹ 11 more rows
# ℹ 5 more variables: homeworld <chr>, species <chr>, films <list>,
# vehicles <list>, starships <list>¿Cómo harías para… filtrar los personajes que no tienen ojos marrones?
# A tibble: 66 × 14
name height mass hair_color skin_color eye_color birth_year sex gender
<chr> <int> <dbl> <chr> <chr> <chr> <dbl> <chr> <chr>
1 Luke Sk… 172 77 blond fair blue 19 male mascu…
2 C-3PO 167 75 <NA> gold yellow 112 none mascu…
3 R2-D2 96 32 <NA> white, bl… red 33 none mascu…
4 Darth V… 202 136 none white yellow 41.9 male mascu…
5 Owen La… 178 120 brown, gr… light blue 52 male mascu…
6 Beru Wh… 165 75 brown light blue 47 fema… femin…
7 R5-D4 97 32 <NA> white, red red NA none mascu…
8 Obi-Wan… 182 77 auburn, w… fair blue-gray 57 male mascu…
9 Anakin … 188 84 blond fair blue 41.9 male mascu…
10 Wilhuff… 180 NA auburn, g… fair blue 64 male mascu…
# ℹ 56 more rows
# ℹ 5 more variables: homeworld <chr>, species <chr>, films <list>,
# vehicles <list>, starships <list>¿Cómo harías para … filtrar los personajes que tengan los ojos marrones o azules?
# A tibble: 40 × 14
name height mass hair_color skin_color eye_color birth_year sex gender
<chr> <int> <dbl> <chr> <chr> <chr> <dbl> <chr> <chr>
1 Luke Sk… 172 77 blond fair blue 19 male mascu…
2 Leia Or… 150 49 brown light brown 19 fema… femin…
3 Owen La… 178 120 brown, gr… light blue 52 male mascu…
4 Beru Wh… 165 75 brown light blue 47 fema… femin…
5 Biggs D… 183 84 black light brown 24 male mascu…
6 Anakin … 188 84 blond fair blue 41.9 male mascu…
7 Wilhuff… 180 NA auburn, g… fair blue 64 male mascu…
8 Chewbac… 228 112 brown unknown blue 200 male mascu…
9 Han Solo 180 80 brown fair brown 29 male mascu…
10 Jek Ton… 180 110 brown fair blue NA male mascu…
# ℹ 30 more rows
# ℹ 5 more variables: homeworld <chr>, species <chr>, films <list>,
# vehicles <list>, starships <list>Fíjate que %in% es equivalente a concatenar varios == con una conjunción o (|)
# A tibble: 40 × 14
name height mass hair_color skin_color eye_color birth_year sex gender
<chr> <int> <dbl> <chr> <chr> <chr> <dbl> <chr> <chr>
1 Luke Sk… 172 77 blond fair blue 19 male mascu…
2 Leia Or… 150 49 brown light brown 19 fema… femin…
3 Owen La… 178 120 brown, gr… light blue 52 male mascu…
4 Beru Wh… 165 75 brown light blue 47 fema… femin…
5 Biggs D… 183 84 black light brown 24 male mascu…
6 Anakin … 188 84 blond fair blue 41.9 male mascu…
7 Wilhuff… 180 NA auburn, g… fair blue 64 male mascu…
8 Chewbac… 228 112 brown unknown blue 200 male mascu…
9 Han Solo 180 80 brown fair brown 29 male mascu…
10 Jek Ton… 180 110 brown fair blue NA male mascu…
# ℹ 30 more rows
# ℹ 5 more variables: homeworld <chr>, species <chr>, films <list>,
# vehicles <list>, starships <list>¿Cómo harías para … filtrar los personajes que midan entre 120 y 160 cm?
¿Qué tipo de variable es? –> La variable height es cuantitativa continua así que deberemos filtrar por rangos de valores (intervalos) –> usaremos between()
# A tibble: 6 × 14
name height mass hair_color skin_color eye_color birth_year sex gender
<chr> <int> <dbl> <chr> <chr> <chr> <dbl> <chr> <chr>
1 Leia Org… 150 49 brown light brown 19 fema… femin…
2 Mon Moth… 150 NA auburn fair blue 48 fema… femin…
3 Nien Nunb 160 68 none grey black NA male mascu…
4 Watto 137 NA black blue, grey yellow NA male mascu…
5 Gasgano 122 NA none white, bl… black NA male mascu…
6 Cordé 157 NA brown light brown NA fema… femin…
# ℹ 5 more variables: homeworld <chr>, species <chr>, films <list>,
# vehicles <list>, starships <list>¿Cómo harías… filtrar los personajes que tengan ojos y no sean humanos?
# A tibble: 3 × 14
name height mass hair_color skin_color eye_color birth_year sex gender
<chr> <int> <dbl> <chr> <chr> <chr> <dbl> <chr> <chr>
1 Yoda 66 17 white green brown 896 male mascu…
2 Wicket S… 88 20 brown brown brown 8 male mascu…
3 Eeth Koth 171 NA black brown brown NA male mascu…
# ℹ 5 more variables: homeworld <chr>, species <chr>, films <list>,
# vehicles <list>, starships <list>Hay un filtro especial para una de las operaciones más habituales en depuración: retirar los ausentes. Para ello podemos usar dentro de un filtro is.na(), que nos devuelve TRUE/FALSE en función de si es ausente, o bien …
Usar drop_na(): si no indicamos variable, elimina registros con ausente en cualquier variable. Más adelante veremos como imputar esos ausentes
# A tibble: 7 × 4
name mass height hair_color
<chr> <dbl> <int> <chr>
1 Luke Skywalker 77 172 blond
2 C-3PO 75 167 <NA>
3 R2-D2 32 96 <NA>
4 Darth Vader 136 202 none
5 Leia Organa 49 150 brown
6 Owen Lars 120 178 brown, grey
7 Beru Whitesun lars 75 165 brown # A tibble: 7 × 4
name mass height hair_color
<chr> <dbl> <int> <chr>
1 Luke Skywalker 77 172 blond
2 Darth Vader 136 202 none
3 Leia Organa 49 150 brown
4 Owen Lars 120 178 brown, grey
5 Beru Whitesun lars 75 165 brown
6 Biggs Darklighter 84 183 black
7 Obi-Wan Kenobi 77 182 auburn, whiteIntenta realizar los siguientes ejercicios sin mirar las soluciones
📝 Selecciona del conjunto de starwars solo los personajes que sean androides o cuyo valor en species sea desconocido
📝 Selecciona del conjunto de starwars solo los personajes cuyo peso esté entre 65 y 90 kg.
📝 Tras limpiar de ausentes en todas las variables, selecciona del conjunto de starwars solo los personajes que sean humanos y que vengan de Tatooine
📝 Selecciona del conjunto original de starwars los personajes no humanos, male en el sexo y que midan entre 120 y 170 cm, o los personajes con ojos marrones o rojos.
📝 Busca información en la ayuda de la función str_detect() del paquete {stringr} (cargado en {tidyverse}). Consejo: prueba antes las funciones que vayas a usar con algún vector de prueba para poder comprobar su funcionamiento. Tras saber lo que hace, filtra solo aquellos personajes con apellido Skywalker
A veces nos puede interesar realizar un muestreo no aleatorio discreccional, o lo que es lo mismo, filtrar por posición: con slice(posiciones) podremos seleccionar filas concretas pasando como argumento un vector de índices
# A tibble: 1 × 4
name height mass hair_color
<chr> <int> <dbl> <chr>
1 Luke Skywalker 172 77 blond # A tibble: 4 × 8
name height mass hair_color skin_color eye_color birth_year sex
<chr> <int> <dbl> <chr> <chr> <chr> <dbl> <chr>
1 C-3PO 167 75 <NA> gold yellow 112 none
2 Beru Whitesun l… 165 75 brown light blue 47 fema…
3 Obi-Wan Kenobi 182 77 auburn, w… fair blue-gray 57 male
4 Qui-Gon Jinn 193 89 brown fair blue 92 male Disponemos de opciones por defecto:
slice_head(n = ...) y slice_tail(n = ...) podemos obtener la cabecera y cola de la tabla# A tibble: 2 × 4
name height mass hair_color
<chr> <int> <dbl> <chr>
1 Luke Skywalker 172 77 blond
2 C-3PO 167 75 <NA> Disponemos de opciones por defecto:
slice_max() y slice_min() obtenemos la filas con menor/mayor valor de una variable (si empate, todas salvo que with_ties = FALSE) que indicamos en order_by = ...# A tibble: 2 × 4
name height mass hair_color
<chr> <int> <dbl> <chr>
1 Ratts Tyerell 79 15 none
2 Yoda 66 17 white El conocido como muestreo aleatorio simple se basa en seleccionar individuos aleatoriamente, de forma que cada uno tenga ciertas probabilidades de ser seleccionado. Con slice_sample(n = ...) podemos extraer n registros aleatoriamente (a priori equiprobables).
# A tibble: 2 × 14
name height mass hair_color skin_color eye_color birth_year sex gender
<chr> <int> <dbl> <chr> <chr> <chr> <dbl> <chr> <chr>
1 Mace Win… 188 84 none dark brown 72 male mascu…
2 Cliegg L… 183 NA brown fair blue 82 male mascu…
# ℹ 5 more variables: homeworld <chr>, species <chr>, films <list>,
# vehicles <list>, starships <list>También podremos indicarle la proporción de datos a samplear (en lugar del número) y si queremos que sea con reemplazamiento (que se puedan repetir).
# 5% de registros aleatorios con reemplazamiento
starwars |>
slice_sample(prop = 0.05, replace = TRUE)# A tibble: 4 × 14
name height mass hair_color skin_color eye_color birth_year sex gender
<chr> <int> <dbl> <chr> <chr> <chr> <dbl> <chr> <chr>
1 R5-D4 97 32 <NA> white, red red NA none mascu…
2 Watto 137 NA black blue, grey yellow NA male mascu…
3 Jabba De… 175 1358 <NA> green-tan… orange 600 herm… mascu…
4 R5-D4 97 32 <NA> white, red red NA none mascu…
# ℹ 5 more variables: homeworld <chr>, species <chr>, films <list>,
# vehicles <list>, starships <list>Como decíamos, «aleatorio» no es igual que «equiprobable», así que podemos pasarle un vector de probabilidades. Por ejemplo, vamos a forzar que sea muy improbable sacar una fila que no sean las dos primeras
# A tibble: 2 × 14
name height mass hair_color skin_color eye_color birth_year sex gender
<chr> <int> <dbl> <chr> <chr> <chr> <dbl> <chr> <chr>
1 C-3PO 167 75 <NA> gold yellow 112 none mascu…
2 Luke Sky… 172 77 blond fair blue 19 male mascu…
# ℹ 5 more variables: homeworld <chr>, species <chr>, films <list>,
# vehicles <list>, starships <list># A tibble: 2 × 14
name height mass hair_color skin_color eye_color birth_year sex gender
<chr> <int> <dbl> <chr> <chr> <chr> <dbl> <chr> <chr>
1 Luke Sky… 172 77 blond fair blue 19 male mascu…
2 C-3PO 167 75 <NA> gold yellow 112 none mascu…
# ℹ 5 more variables: homeworld <chr>, species <chr>, films <list>,
# vehicles <list>, starships <list>La función slice_sample() es simplemente una integración de {tidyverse} de la función básica de R conocida como sample() que nos permite muestrear elementos
Por ejemplo, vamos a muestrear 10 tiradas de un dado, indicándole
x)size)TRUE entonces pueden salir repetidas, como en el caso del dado)Intenta realizar los siguientes ejercicios sin mirar las soluciones
📝 Selecciona solo los personajes que sean humanos y de ojos marrones, para después ordernarlos en altura descendente y peso ascendente.
📝 Extrae aleatoriamente 10 personajes pero de forma que la probabilidad de que salga cada uno sea proporcional a su peso (más pesados, más probable)
Operaciones con columnas en tidyverse
También podemos ordenar filas en función de alguna variable con arrange()
# A tibble: 5 × 6
name height mass hair_color skin_color eye_color
<chr> <int> <dbl> <chr> <chr> <chr>
1 Ratts Tyerell 79 15 none grey, blue unknown
2 Yoda 66 17 white green brown
3 Wicket Systri Warrick 88 20 brown brown brown
4 R2-D2 96 32 <NA> white, blue red
5 R5-D4 97 32 <NA> white, red red Por defecto de menor a mayor pero podemos invertir el orden con desc()
# A tibble: 5 × 3
name height mass
<chr> <int> <dbl>
1 Yarael Poof 264 NA
2 Tarfful 234 136
3 Lama Su 229 88
4 Chewbacca 228 112
5 Roos Tarpals 224 82Muchas veces necesitaremos asegurarnos que no hay duplicados en alguna variable (DNI) y podemos eliminar filas duplicadas con distinct().
# A tibble: 5 × 1
sex
<chr>
1 male
2 none
3 female
4 hermaphroditic
5 <NA> Para mantener todas las columnas de la tabla usaremos .keep_all = TRUE.
# A tibble: 3 × 14
name height mass hair_color skin_color eye_color birth_year sex gender
<chr> <int> <dbl> <chr> <chr> <chr> <dbl> <chr> <chr>
1 Luke Sky… 172 77 blond fair blue 19 male mascu…
2 C-3PO 167 75 <NA> gold yellow 112 none mascu…
3 Leia Org… 150 49 brown light brown 19 fema… femin…
# ℹ 5 more variables: homeworld <chr>, species <chr>, films <list>,
# vehicles <list>, starships <list>Por último, podemos concatenar nuevas filas con bind_rows() con las nuevas observaciones en tabla (si no cuadran columnas rellena con ausentes)
# A tibble: 2 × 2
nombre edad
<chr> <dbl>
1 javi 33
2 laura 50Intenta realizar los siguientes ejercicios sin mirar las soluciones
📝 Para saber que valores únicos hay en el color de pelo, elimina duplicados de la variable hair_color, eliminando antes los ausentes de dicha variable.
📝 De los personajes que son humanos y miden más de 160 cm, elimina duplicados en color de ojos, elimina ausentes en peso, selecciona los 3 más altos, y orden de mayor a menor peso. Devuelve la tabla.
La clave de {tidyverse} es la legibilidad: es importantísimo que el código se entienda, por nuestro yo el futuro pero también por la transparencia algorítmica hacia los demás
Por ejemplo: quitaremos ausentes de la variable peso, filtraremos los personajes humanos y altura superior a 140cm, sin duplicados en el color de pelo, extrayendo los 5 más altos y obteniendo 2 personajes aleatorios finalmente.
La clave de {tidyverse} es la legibilidad: es importantísimo que el código se entienda, por nuestro yo el futuro pero también por la transparencia algorítmica hacia los demás
Por ejemplo: quitaremos ausentes de la variable peso, filtraremos los personajes humanos y altura superior a 140cm, sin duplicados en el color de pelo, extrayendo los 5 más altos y obteniendo 2 personajes aleatorios finalmente.
La clave de {tidyverse} es la legibilidad: es importantísimo que el código se entienda, por nuestro yo el futuro pero también por la transparencia algorítmica hacia los demás
Por ejemplo: quitaremos ausentes de la variable peso, filtraremos los personajes humanos y altura superior a 140cm, sin duplicados en el color de pelo, extrayendo los 5 más altos y obteniendo 2 personajes aleatorios finalmente.
La clave de {tidyverse} es la legibilidad: es importantísimo que el código se entienda, por nuestro yo el futuro pero también por la transparencia algorítmica hacia los demás
Por ejemplo: quitaremos ausentes de la variable peso, filtraremos los personajes humanos y altura superior a 140cm, sin duplicados en el color de pelo, extrayendo los 5 más altos y obteniendo 2 personajes aleatorios finalmente.
La clave de {tidyverse} es la legibilidad: es importantísimo que el código se entienda, por nuestro yo el futuro pero también por la transparencia algorítmica hacia los demás
Por ejemplo: quitaremos ausentes de la variable peso, filtraremos los personajes humanos y altura superior a 140cm, sin duplicados en el color de pelo, extrayendo los 5 más altos y obteniendo 2 personajes aleatorios finalmente.
La opción más sencilla para seleccionar variables por nombre es select(), dando como argumentos los nombres de columnas sin comillas.
# A tibble: 87 × 2
name hair_color
<chr> <chr>
1 Luke Skywalker blond
2 C-3PO <NA>
3 R2-D2 <NA>
4 Darth Vader none
5 Leia Organa brown
6 Owen Lars brown, grey
7 Beru Whitesun lars brown
8 R5-D4 <NA>
9 Biggs Darklighter black
10 Obi-Wan Kenobi auburn, white
# ℹ 77 more rowsLa función select() nos permite seleccionar varias variables a la vez, incluso concatenando sus nombres como si fuesen índices numéricos
# A tibble: 4 × 6
name height mass hair_color skin_color eye_color
<chr> <int> <dbl> <chr> <chr> <chr>
1 Luke Skywalker 172 77 blond fair blue
2 C-3PO 167 75 <NA> gold yellow
3 R2-D2 96 32 <NA> white, blue red
4 Darth Vader 202 136 none white yellow Tenemos además palabras reservadas: everything() todas las variables…
# A tibble: 4 × 14
mass homeworld name height hair_color skin_color eye_color birth_year sex
<dbl> <chr> <chr> <int> <chr> <chr> <chr> <dbl> <chr>
1 77 Tatooine Luke … 172 blond fair blue 19 male
2 75 Tatooine C-3PO 167 <NA> gold yellow 112 none
3 32 Naboo R2-D2 96 <NA> white, bl… red 33 none
4 136 Tatooine Darth… 202 none white yellow 41.9 male
# ℹ 5 more variables: gender <chr>, species <chr>, films <list>,
# vehicles <list>, starships <list>…y last_col() para referirnos a la última columna.
# A tibble: 4 × 5
name height mass homeworld starships
<chr> <int> <dbl> <chr> <list>
1 Luke Skywalker 172 77 Tatooine <chr [2]>
2 C-3PO 167 75 Tatooine <chr [0]>
3 R2-D2 96 32 Naboo <chr [0]>
4 Darth Vader 202 136 Tatooine <chr [1]>También podemos jugar con patrones en el nombre, aquellas que comiencen por un prefijo (starts_with()), terminen con un sufijo (ends_with()), contengan un texto (contains()) o cumplan una expresión regular (matches()).
# variables cuyo nombre acaba en "color" y contengan sexo o género
starwars |> select(ends_with("color"), matches("sex|gender"))# A tibble: 87 × 5
hair_color skin_color eye_color sex gender
<chr> <chr> <chr> <chr> <chr>
1 blond fair blue male masculine
2 <NA> gold yellow none masculine
3 <NA> white, blue red none masculine
4 none white yellow male masculine
5 brown light brown female feminine
6 brown, grey light blue male masculine
7 brown light blue female feminine
8 <NA> white, red red none masculine
9 black light brown male masculine
10 auburn, white fair blue-gray male masculine
# ℹ 77 more rowsIncluso podemos seleccionar por rango numérico si tenemos variables con un prefijo y números.
Por último, podemos seleccionar columnas por tipo de dato haciendo uso de where() y dentro una función que devuelva un valor lógico de tipo de dato.
# A tibble: 87 × 11
height mass birth_year name hair_color skin_color eye_color sex gender
<int> <dbl> <dbl> <chr> <chr> <chr> <chr> <chr> <chr>
1 172 77 19 Luke Sk… blond fair blue male mascu…
2 167 75 112 C-3PO <NA> gold yellow none mascu…
3 96 32 33 R2-D2 <NA> white, bl… red none mascu…
4 202 136 41.9 Darth V… none white yellow male mascu…
5 150 49 19 Leia Or… brown light brown fema… femin…
6 178 120 52 Owen La… brown, gr… light blue male mascu…
7 165 75 47 Beru Wh… brown light blue fema… femin…
8 97 32 NA R5-D4 <NA> white, red red none mascu…
9 183 84 24 Biggs D… black light brown male mascu…
10 182 77 57 Obi-Wan… auburn, w… fair blue-gray male mascu…
# ℹ 77 more rows
# ℹ 2 more variables: homeworld <chr>, species <chr>Para facilitar la recolocación de variables tenemos una función para ello, relocate(), indicándole en .after o .before detrás o delante de qué columnas queremos moverlas.
# A tibble: 87 × 14
species name height mass hair_color skin_color eye_color birth_year sex
<chr> <chr> <int> <dbl> <chr> <chr> <chr> <dbl> <chr>
1 Human Luke S… 172 77 blond fair blue 19 male
2 Droid C-3PO 167 75 <NA> gold yellow 112 none
3 Droid R2-D2 96 32 <NA> white, bl… red 33 none
4 Human Darth … 202 136 none white yellow 41.9 male
5 Human Leia O… 150 49 brown light brown 19 fema…
6 Human Owen L… 178 120 brown, gr… light blue 52 male
7 Human Beru W… 165 75 brown light blue 47 fema…
8 Droid R5-D4 97 32 <NA> white, red red NA none
9 Human Biggs … 183 84 black light brown 24 male
10 Human Obi-Wa… 182 77 auburn, w… fair blue-gray 57 male
# ℹ 77 more rows
# ℹ 5 more variables: gender <chr>, homeworld <chr>, films <list>,
# vehicles <list>, starships <list>A veces también podemos querer modificar la «metainformación» de los datos, renombrando columnas. Para ello usaremos de rename() poniendo primero el nombre nuevo y luego el antiguo.
# A tibble: 87 × 14
nombre altura peso hair_color skin_color eye_color birth_year sex gender
<chr> <int> <dbl> <chr> <chr> <chr> <dbl> <chr> <chr>
1 Luke Sk… 172 77 blond fair blue 19 male mascu…
2 C-3PO 167 75 <NA> gold yellow 112 none mascu…
3 R2-D2 96 32 <NA> white, bl… red 33 none mascu…
4 Darth V… 202 136 none white yellow 41.9 male mascu…
5 Leia Or… 150 49 brown light brown 19 fema… femin…
6 Owen La… 178 120 brown, gr… light blue 52 male mascu…
7 Beru Wh… 165 75 brown light blue 47 fema… femin…
8 R5-D4 97 32 <NA> white, red red NA none mascu…
9 Biggs D… 183 84 black light brown 24 male mascu…
10 Obi-Wan… 182 77 auburn, w… fair blue-gray 57 male mascu…
# ℹ 77 more rows
# ℹ 5 more variables: homeworld <chr>, species <chr>, films <list>,
# vehicles <list>, starships <list>Si observas la salida de los select() sigue siendo una tabla tibble, ya que nos preserva la naturaleza de nuestros datos.
A veces no querremos dicha estructura sino extraer literalmente la columna en un vector, algo que podemos hacer con pull()
[1] "Luke Skywalker" "C-3PO" "R2-D2"
[4] "Darth Vader" "Leia Organa" "Owen Lars"
[7] "Beru Whitesun lars" "R5-D4" "Biggs Darklighter"
[10] "Obi-Wan Kenobi" "Anakin Skywalker" "Wilhuff Tarkin"
[13] "Chewbacca" "Han Solo" "Greedo"
[16] "Jabba Desilijic Tiure" "Wedge Antilles" "Jek Tono Porkins"
[19] "Yoda" "Palpatine" "Boba Fett"
[22] "IG-88" "Bossk" "Lando Calrissian"
[25] "Lobot" "Ackbar" "Mon Mothma"
[28] "Arvel Crynyd" "Wicket Systri Warrick" "Nien Nunb"
[31] "Qui-Gon Jinn" "Nute Gunray" "Finis Valorum"
[34] "Jar Jar Binks" "Roos Tarpals" "Rugor Nass"
[37] "Ric Olié" "Watto" "Sebulba"
[40] "Quarsh Panaka" "Shmi Skywalker" "Darth Maul"
[43] "Bib Fortuna" "Ayla Secura" "Dud Bolt"
[46] "Gasgano" "Ben Quadinaros" "Mace Windu"
[49] "Ki-Adi-Mundi" "Kit Fisto" "Eeth Koth"
[52] "Adi Gallia" "Saesee Tiin" "Yarael Poof"
[55] "Plo Koon" "Mas Amedda" "Gregar Typho"
[58] "Cordé" "Cliegg Lars" "Poggle the Lesser"
[61] "Luminara Unduli" "Barriss Offee" "Dormé"
[64] "Dooku" "Bail Prestor Organa" "Jango Fett"
[67] "Zam Wesell" "Dexter Jettster" "Lama Su"
[70] "Taun We" "Jocasta Nu" "Ratts Tyerell"
[73] "R4-P17" "Wat Tambor" "San Hill"
[76] "Shaak Ti" "Grievous" "Tarfful"
[79] "Raymus Antilles" "Sly Moore" "Tion Medon"
[82] "Finn" "Rey" "Poe Dameron"
[85] "BB8" "Captain Phasma" "Padmé Amidala" Intenta realizar los siguientes ejercicios sin mirar las soluciones
📝 Filtra el conjunto de personajes y quédate solo con aquellos que en la variable height no tengan un dato ausente. Con los datos obtenidos del filtro anterior, selecciona solo las variables name, height, así como todas aquellas variables que CONTENGAN la palabra color en su nombre.
📝 Con los datos obtenidos del ejercicio anterior, traduce el nombre de las columnas a castellano
📝 Con los datos obtenidos del ejercicio anterior, coloca la variable de color de pelo justo detrás de la variable de nombres.
📝 Con los datos obtenidos del ejercicio anterior, comprueba cuántas modalidades únicas hay en la variable de color de pelo (sin usar unique()).
📝 Del conjunto de datos originales, elimina las columnas de tipo lista, y tras ello elimina duplicados en la variable eye_color. Tras eliminar duplicados extrae dicha columna en un vector.
En muchas ocasiones querremos modificar o crear variables con mutate().
Vamos a crear por ejemplo una nueva variable height_m con la altura en metros.
# A tibble: 87 × 15
name height mass hair_color skin_color eye_color birth_year sex gender
<chr> <int> <dbl> <chr> <chr> <chr> <dbl> <chr> <chr>
1 Luke Sk… 172 77 blond fair blue 19 male mascu…
2 C-3PO 167 75 <NA> gold yellow 112 none mascu…
3 R2-D2 96 32 <NA> white, bl… red 33 none mascu…
4 Darth V… 202 136 none white yellow 41.9 male mascu…
5 Leia Or… 150 49 brown light brown 19 fema… femin…
6 Owen La… 178 120 brown, gr… light blue 52 male mascu…
7 Beru Wh… 165 75 brown light blue 47 fema… femin…
8 R5-D4 97 32 <NA> white, red red NA none mascu…
9 Biggs D… 183 84 black light brown 24 male mascu…
10 Obi-Wan… 182 77 auburn, w… fair blue-gray 57 male mascu…
# ℹ 77 more rows
# ℹ 6 more variables: homeworld <chr>, species <chr>, films <list>,
# vehicles <list>, starships <list>, height_m <dbl>Además con los argumentos opcionales podemos recolocar la columna modificada
# A tibble: 87 × 16
height_m IMC name height mass hair_color skin_color eye_color birth_year
<dbl> <dbl> <chr> <int> <dbl> <chr> <chr> <chr> <dbl>
1 1.72 26.0 Luke … 172 77 blond fair blue 19
2 1.67 26.9 C-3PO 167 75 <NA> gold yellow 112
3 0.96 34.7 R2-D2 96 32 <NA> white, bl… red 33
4 2.02 33.3 Darth… 202 136 none white yellow 41.9
5 1.5 21.8 Leia … 150 49 brown light brown 19
6 1.78 37.9 Owen … 178 120 brown, gr… light blue 52
7 1.65 27.5 Beru … 165 75 brown light blue 47
8 0.97 34.0 R5-D4 97 32 <NA> white, red red NA
9 1.83 25.1 Biggs… 183 84 black light brown 24
10 1.82 23.2 Obi-W… 182 77 auburn, w… fair blue-gray 57
# ℹ 77 more rows
# ℹ 7 more variables: sex <chr>, gender <chr>, homeworld <chr>, species <chr>,
# films <list>, vehicles <list>, starships <list>Importante…
Cuando aplicamos mutate(), debemos de acordarnos que las operaciones se realizan de manera vectorial, elemento a elemento, por lo que la función que usemos dentro debe devolver un vector de igual longitud. En caso contrario, devolverá una constante
# A tibble: 87 × 15
constante name height mass hair_color skin_color eye_color birth_year sex
<dbl> <chr> <int> <dbl> <chr> <chr> <chr> <dbl> <chr>
1 97.3 Luke… 172 77 blond fair blue 19 male
2 97.3 C-3PO 167 75 <NA> gold yellow 112 none
3 97.3 R2-D2 96 32 <NA> white, bl… red 33 none
4 97.3 Dart… 202 136 none white yellow 41.9 male
5 97.3 Leia… 150 49 brown light brown 19 fema…
6 97.3 Owen… 178 120 brown, gr… light blue 52 male
7 97.3 Beru… 165 75 brown light blue 47 fema…
8 97.3 R5-D4 97 32 <NA> white, red red NA none
9 97.3 Bigg… 183 84 black light brown 24 male
10 97.3 Obi-… 182 77 auburn, w… fair blue-gray 57 male
# ℹ 77 more rows
# ℹ 6 more variables: gender <chr>, homeworld <chr>, species <chr>,
# films <list>, vehicles <list>, starships <list>También podemos combinar mutate() con la expresión de control if_else() para recategorizar la variable: si se cumple una condición, hace una cosa, en caso contrario otra.
starwars |>
mutate(human = if_else(species == "Human", "Human", "Not Human"),
.after = name) |>
select(name:mass)# A tibble: 87 × 4
name human height mass
<chr> <chr> <int> <dbl>
1 Luke Skywalker Human 172 77
2 C-3PO Not Human 167 75
3 R2-D2 Not Human 96 32
4 Darth Vader Human 202 136
5 Leia Organa Human 150 49
6 Owen Lars Human 178 120
7 Beru Whitesun lars Human 165 75
8 R5-D4 Not Human 97 32
9 Biggs Darklighter Human 183 84
10 Obi-Wan Kenobi Human 182 77
# ℹ 77 more rowsPara recategorizaciones más complejas tenemos case_when(), por ejemplo, para crear una categoría de los personajes en función de su altura.
starwars |>
drop_na(height) |>
mutate(altura = case_when(height < 120 ~ "enanos",
height < 160 ~ "bajito",
height < 180 ~ "normal",
height < 200 ~ "alto",
TRUE ~ "gigante"), .before = name)# A tibble: 81 × 15
altura name height mass hair_color skin_color eye_color birth_year sex
<chr> <chr> <int> <dbl> <chr> <chr> <chr> <dbl> <chr>
1 normal Luke S… 172 77 blond fair blue 19 male
2 normal C-3PO 167 75 <NA> gold yellow 112 none
3 enanos R2-D2 96 32 <NA> white, bl… red 33 none
4 gigante Darth … 202 136 none white yellow 41.9 male
5 bajito Leia O… 150 49 brown light brown 19 fema…
6 normal Owen L… 178 120 brown, gr… light blue 52 male
7 normal Beru W… 165 75 brown light blue 47 fema…
8 enanos R5-D4 97 32 <NA> white, red red NA none
9 alto Biggs … 183 84 black light brown 24 male
10 alto Obi-Wa… 182 77 auburn, w… fair blue-gray 57 male
# ℹ 71 more rows
# ℹ 6 more variables: gender <chr>, homeworld <chr>, species <chr>,
# films <list>, vehicles <list>, starships <list>Intenta realizar los siguientes ejercicios sin mirar las soluciones
📝 Selecciona solo las variables nombre, altura y así como todas aquellas variables relacionadas con el color, a la vez que te quedas solo con aquellos que no tengan ausente en la altura.
📝 Con los datos obtenidos del ejercicio anterior, traduce el nombre de las columnas a castellano.
📝 Con los datos obtenidos del ejercicio anterior, coloca la variable de color de pelo justo detrás de la variable de nombres.
📝 Con los datos originales, comprueba cuántas modalidades únicas hay en la variable de color de pelo.
📝 Del dataset original, selecciona solo las variables numéricas y de tipo texto. Tras ello define una nueva variable llamada under_18 que nos recategorice la variable de edad: TRUE si es menor de edad y FALSE en caso contrario
📝 Del dataset original, crea una nueva columna llamada auburn (cobrizo/caoba) que nos diga TRUE si el color de pelo contiene dicha palabra y FALSE en caso contrario (reminder str_detect()).
📝 Del dataset original, incluye una columna que calcule el IMC. Tras ello, crea una nueva variable que valga NA si no es humano, delgadez por debajo de 18, normal entre 18 y 30, sobrepeso por encima de 30.
Haciendo uso de todo lo aprendido, vamos a proceder a crear una tabla con datos de bebés de tamaño n = 20 en donde simulemos el sexo de los bebés y su peso
Crea un tibble con dos columnas, una llamada id_bebe y otra llamada sexo. En el primer caso debe ir de 1 a 20. En el segundo caso, simula su sexo de manera que haya un 0.5 de probabilidad de chico y 0.5 de chica.
Conocido el sexo, crea una tercera columna llamada peso en la que simules dicho valor. Supondremos que para los chicos el peso sigue una distribución \(N(\mu = 3.266kg, \sigma = 0.514)\) y que para las chicas sigue una distribución \(N(\mu = 3.155kg, \sigma = 0.495)\).
tb <-
tibble("id_bebe" = 1:20,
"sexo" = sample(c("chico", "chica"), size = 20, replace = TRUE))
microbenchmark::microbenchmark(tb |>
mutate(peso =
rnorm(n = 20, mean = if_else(sexo == "chico", 3.266, 3.155),
sd = if_else(sexo == "chico", 0.514, 0.495))), for (i in 1:20) { tb[i, 3] = ifelse(tb$sexo[i] == "chico", rnorm(n = 1, mean = 3.266, sd = 0.514), rnorm(n = 1, mean = 3.155, sd = 0.495)) }, times = 1e3)Unit: microseconds
expr
mutate(tb, peso = rnorm(n = 20, mean = if_else(sexo == "chico", 3.266, 3.155), sd = if_else(sexo == "chico", 0.514, 0.495)))
for (i in 1:20) { tb[i, 3] = ifelse(tb$sexo[i] == "chico", rnorm(n = 1, mean = 3.266, sd = 0.514), rnorm(n = 1, mean = 3.155, sd = 0.495)) }
min lq mean median uq max neval cld
594.9 648.75 849.7468 701.70 816.15 59327.7 1000 a
2373.6 2522.30 2870.1816 2594.45 2867.25 8377.3 1000 bSummarise y group_by(). Contar y resumir: estadísticas desagregadas por factores/grupos.
Hasta ahora solo hemos transformado o consultado los datos pero no hemos generado estadísticas. Empecemos por lo sencillo: ¿cómo contar (frecuencias)?
Cuando lo usamos en solitario count() nos devolverá simplemente el número de registros , pero cuando lo usamos con variables count() calcula lo que se conoce como frecuencias: número de elementos de cada modalidad.
Además si pasamos varias variables nos calcula lo que se conoce como una tabla de contigencia. Con sort = TRUE nos devolverá el conteo ordenado (más frecuentes primero).
Una de las funciones más potentes a combinar con las acciones vistas es group_by(), que nos permitirá agrupar nuestros registros previamente
Cuando apliquemos group_by() es importante entender que NO MODIFICA los datos, sino que nos crea una variable de grupo (subtablas por cada grupo) que modificará las acciones futuras: las operaciones se aplicarán a cada subtabla por separado
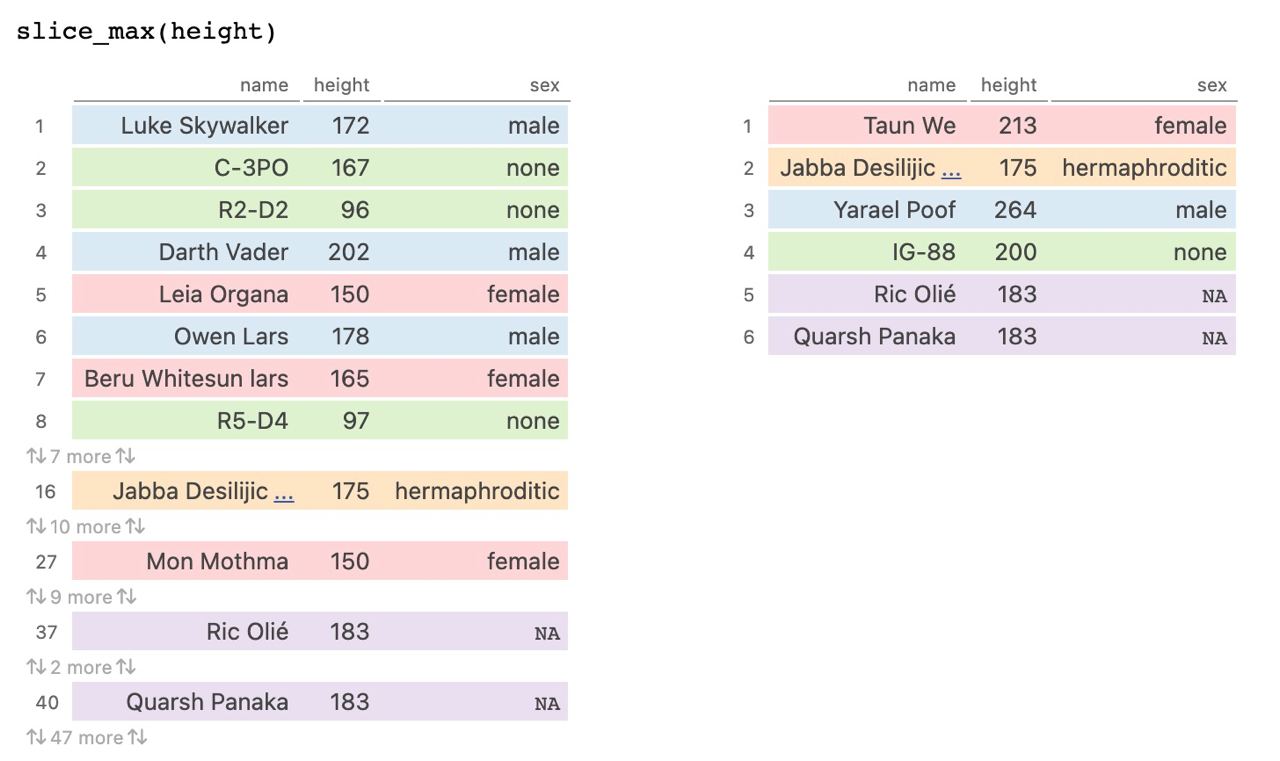
Por ejemplo, imaginemos que queremos extraer el personaje más alto con slice_max().
# A tibble: 1 × 14
name height mass hair_color skin_color eye_color birth_year sex gender
<chr> <int> <dbl> <chr> <chr> <chr> <dbl> <chr> <chr>
1 Yarael P… 264 NA none white yellow NA male mascu…
# ℹ 5 more variables: homeworld <chr>, species <chr>, films <list>,
# vehicles <list>, starships <list>¿Y si queremos extraer el personaje más alto pero…de cada uno de los sexos?
# A tibble: 6 × 14
name height mass hair_color skin_color eye_color birth_year sex gender
<chr> <int> <dbl> <chr> <chr> <chr> <dbl> <chr> <chr>
1 Taun We 213 NA none grey black NA fema… femin…
2 Jabba De… 175 1358 <NA> green-tan… orange 600 herm… mascu…
3 Yarael P… 264 NA none white yellow NA male mascu…
4 IG-88 200 140 none metal red 15 none mascu…
5 Ric Olié 183 NA brown fair blue NA <NA> <NA>
6 Quarsh P… 183 NA black dark brown 62 <NA> <NA>
# ℹ 5 more variables: homeworld <chr>, species <chr>, films <list>,
# vehicles <list>, starships <list>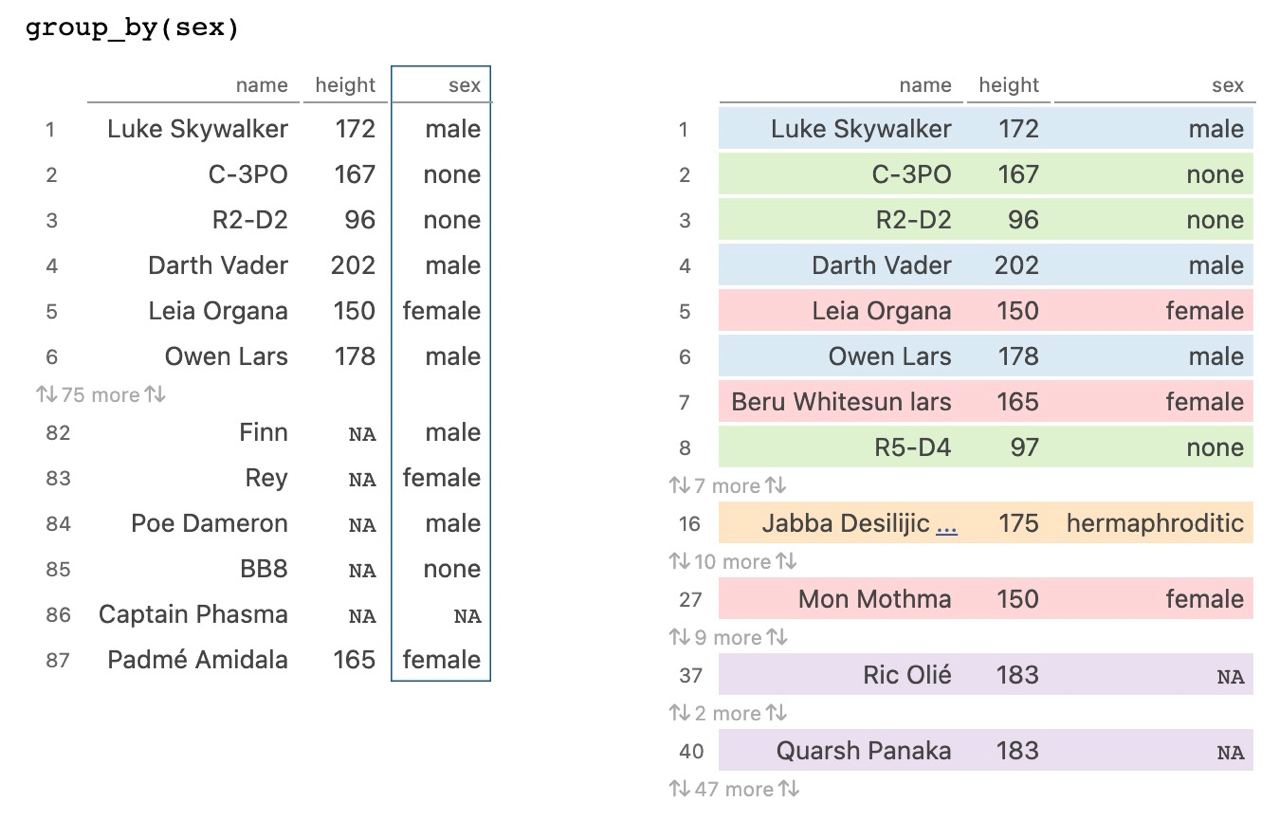
La web https://tidydatatutor.com/ permite visualizar las operaciones de {tidyverse} (con el pipe antiguo)
Importante
Recuerda siempre hacer ungroup para eliminar la variable de grupo creada
En la nueva versión de {dplyr} ahora se permite incluir la variable de grupo en la llamada a muchas funciones con el argumento by = ... o .by = ...
# A tibble: 6 × 6
name height mass hair_color skin_color eye_color
<chr> <int> <dbl> <chr> <chr> <chr>
1 Yarael Poof 264 NA none white yellow
2 IG-88 200 140 none metal red
3 Taun We 213 NA none grey black
4 Jabba Desilijic Tiure 175 1358 <NA> green-tan, brown orange
5 Ric Olié 183 NA brown fair blue
6 Quarsh Panaka 183 NA black dark brown Una opción muy útil usada antes de una operación también es rowwise(): toda operación que venga después se aplicará en cada fila por separado. Por ejemplo, vamos a definir un conjunto dummy de notas.
Si aplicamos la media directamente el valor será idéntico ya que nos ha hecho la media global, pero nos gustaría sacar una media por registro. Para eso usaremos rowwise()
Por último tenemos summarise(), que nos permitirá sacar resúmenes estadísticos. Por ejemplo, vamos a calcular la media de las alturas.
# A tibble: 1 × 1
media_altura
<dbl>
1 174.Cuidado
Fíjate que mutate() devuelve tantas filas como registros originales, mientras que con summarise() calcula un nuevo dataset de resumen, solo incluyendo aquello que esté indicado.
Si además esto lo combinamos con la agrupación de group_by() o .by = ..., en pocas líneas de código puedes obtener estadísticas desagreagadas
En el nuevo {dplyr} han incluido reframe() para evitar problemas de summarise() cuando devolvemos más de un valor por variable.
Warning: Returning more (or less) than 1 row per `summarise()` group was deprecated in
dplyr 1.1.0.
ℹ Please use `reframe()` instead.
ℹ When switching from `summarise()` to `reframe()`, remember that `reframe()`
always returns an ungrouped data frame and adjust accordingly.# A tibble: 5 × 1
`quantile(mass)`
<dbl>
1 15
2 55.6
3 79
4 84.5
5 1358 Un truco es hacer uso de selectores across() y where(). El primero nos permite actuar sobre varias columnas por nombre (con mutate() o summarise())
# A tibble: 5 × 2
sex medias$height $mass
<chr> <dbl> <dbl>
1 male 179. 81.0
2 none 131. 69.8
3 female 169. 54.7
4 hermaphroditic 175 1358
5 <NA> 181. 48 El segundo, where(), nos permite hacer lo mismo pero seleccionando por tipo.
# A tibble: 6 × 5
sex gender height mass birth_year
<chr> <chr> <dbl> <dbl> <dbl>
1 male masculine 179. 81.0 85.5
2 none masculine 140 69.8 53.3
3 female feminine 169. 54.7 47.2
4 hermaphroditic masculine 175 1358 600
5 <NA> <NA> 181. 48 62
6 none feminine 96 NaN NaN Intenta realizar los siguientes ejercicios sin mirar las soluciones
📝 Calcula cuántos personajes hay de cada especie, ordenados de más a menor frecuencia.
📝 Tras eliminar ausentes en las variables de peso y estatura, añade una nueva variable que nos calcule el IMC de cada personaje, y determina el IMC medio de nuestros personajes desagregada por sexo
📝 Obtén la edad del personaje más joven y más viejo de cada sexo.
Vamos antes a hacer un repaso de lo aprendido en {tidyverse}
{tidyr}.Comunicar resultados y material docente: rmd y Quarto
Una de las principales fortalezas de R es la facilidad para generar informes, libros, webs, apuntes y hasta diapositivas (este mismo material por ejemplo). Para ello instalaremos antes
{rmarkdown} (para generar archivos .rmd).rmd ahora como .qmd)
Los archivos de extensión .qmd (o .rmd) nos permitirán fácilmente combinar:
R), con cajitas de código llamadas chunks.La principal ventaja de realizar este tipo de material en Quarto/Rmarkdown es que, al hacerlo desde RStudio, puedes generar un informe o una presentación sin salirte del entorno de programación en el que estás trabajando
De esta forma podrás analizar los datos, resumirlos y a la vez comunicarlos con la misma herramienta.
Recientemente el equipo de RStudio desarrolló Quarto, una versión mejorada de Rmarkdown (archivos .qmd), con un formato un poco más estético y simple. Tienes toda la documentación y ejemplos en https://quarto.org/
Vamos a crear el primer fichero rmarkdown con Quarto con extensión .qmd. Para ello solo necesitaremos hacer click en
File << New File << Quarto Document
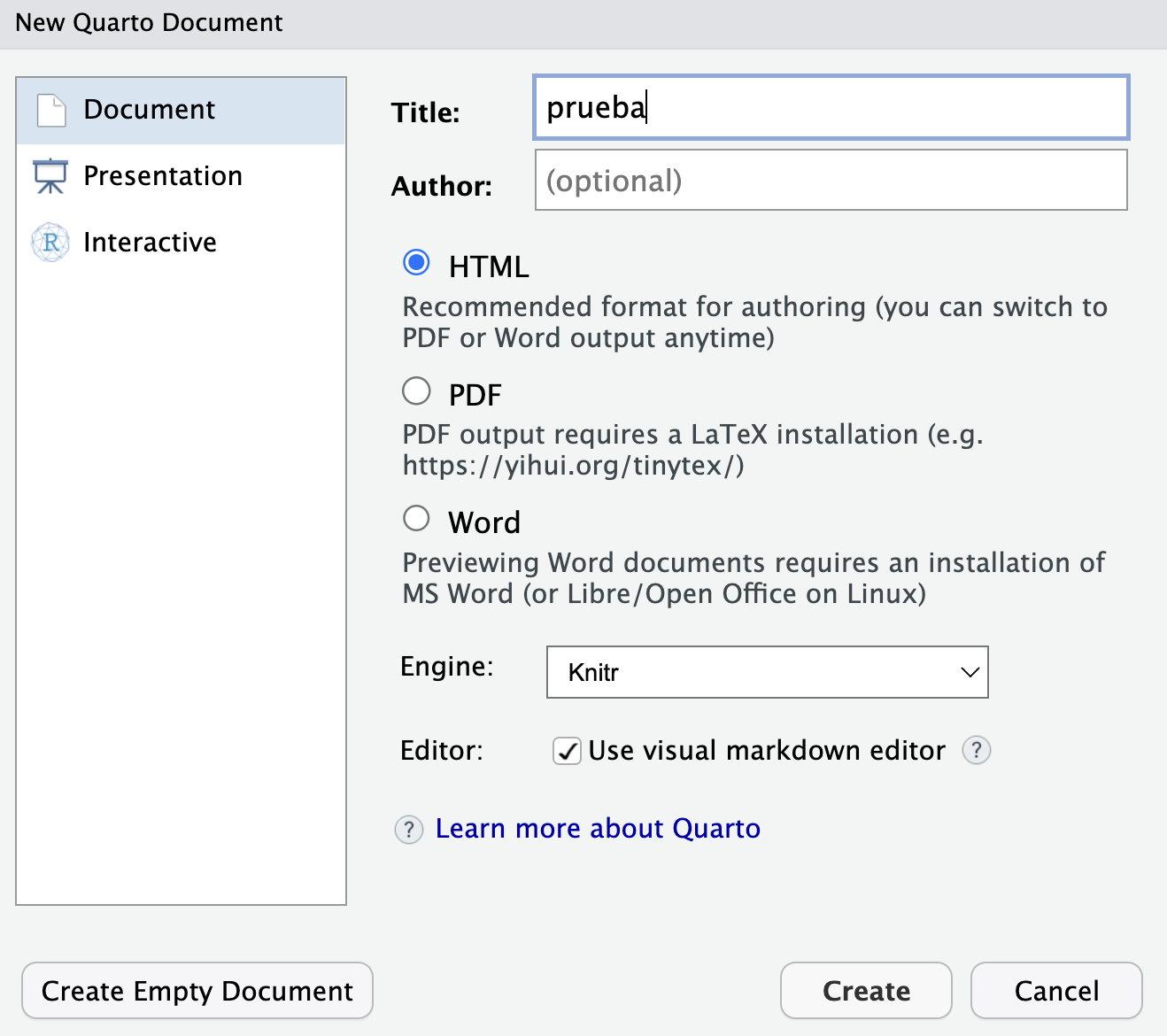
Tras hacerlo nos aparecerán varias opciones de formatos de salida:
.pdf.html (recomendable): documento dinámico, permite la interacción con el usuario, como una «página web»..doc (nada recomendable)De momento dejaremos marcado el formato HTML que viene por defecto, y escribiremos el título de nuestro documento. Tras ello tendremos nuestro archivo .qmd (ya no es un script .R como los que hemos abierto hasta ahora).
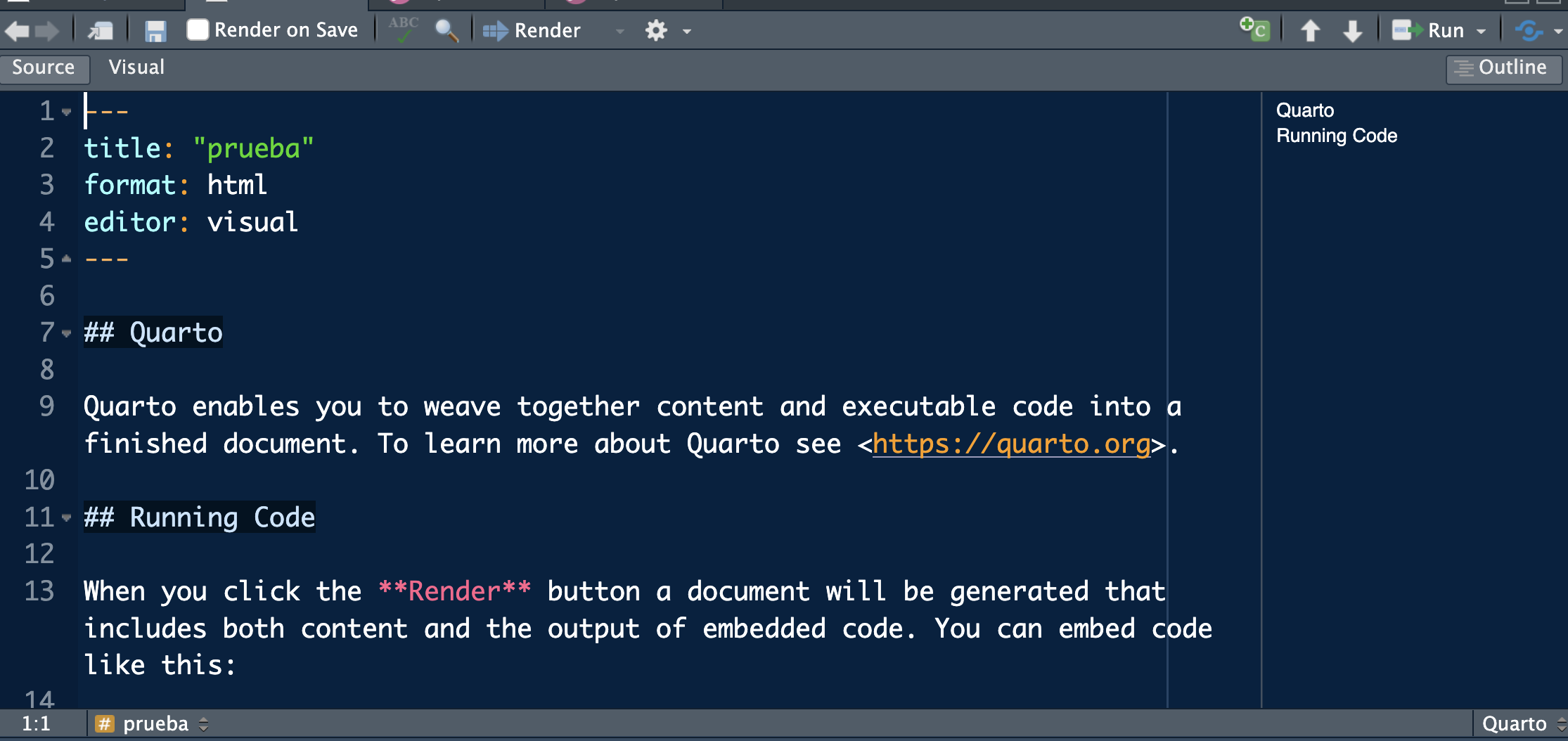
Deberías tener algo similar a la captura de la imagen con dos modos de edición: Source (con código, la opción recomendada hasta que lo domines) y Visual (más parecido a un blog)
Para ejecutar TODO el documento debes clickar Render on Save y darle a guardar.
Deberías haber obtenido una salida en html similar a esta (y se te ha generado en tu ordenador un archivo html)

Un fichero .qmd se divide básicamente en tres partes:
Cabecera: la parte que tienes al inicio entre ---.
Texto: que podremos formatear y mejorar con negritas (escrito como negritas, con doble astérisco al inicio y final), cursivas (cursivas, con barra baja al inicio y final) o destacar nombres de funciones o variables de R. Puedes añadir ecuaciones como \(x^2\) (he escrito $x^2$, entre dólares).
Código R
La cabecera están en formato YAML y contiene los metadatos del documento:
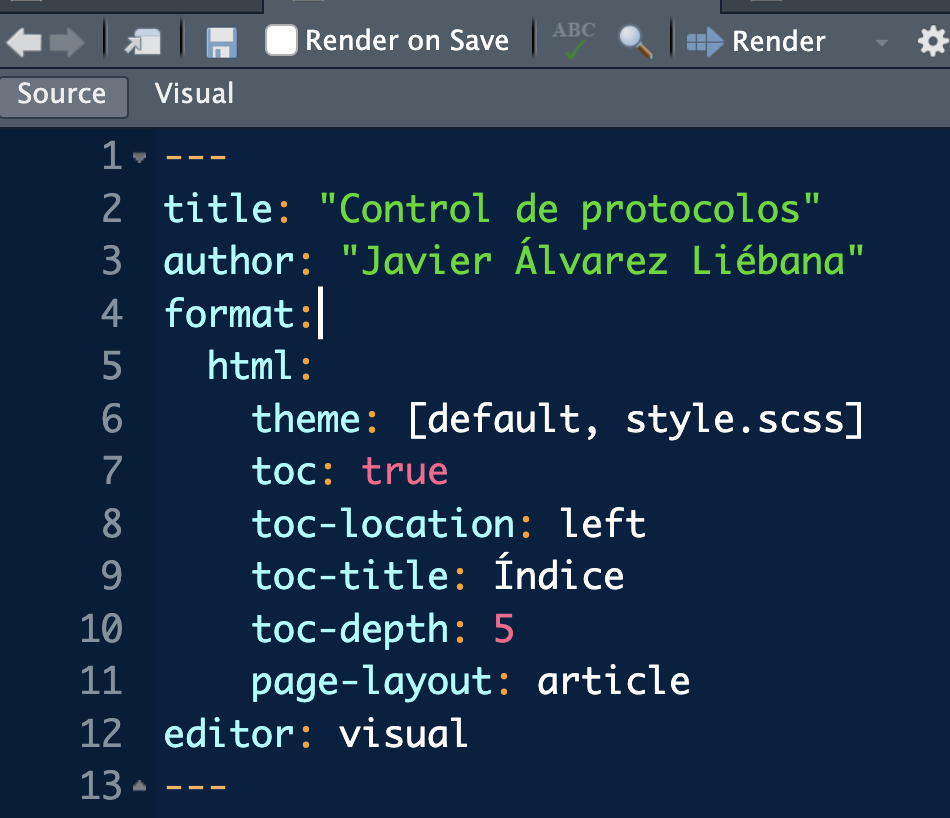
title y subtitle: el título/subtítulo del documentoauthor: autor del mismodate: fechaformat: formato de salida (podremos personalizar)
theme: si tienes algún archivo de estilostoc: si quieres índice o notoc-location: posición del índicetoc-title: título del índicetoc-depth: profundidad del índiceeditor: si estás en modo visual o source.Respecto a la escritura solo hay una cosa importante: salvo que indiquemos lo contrario, TODO lo que vamos a escribir es texto (normal). No código R.
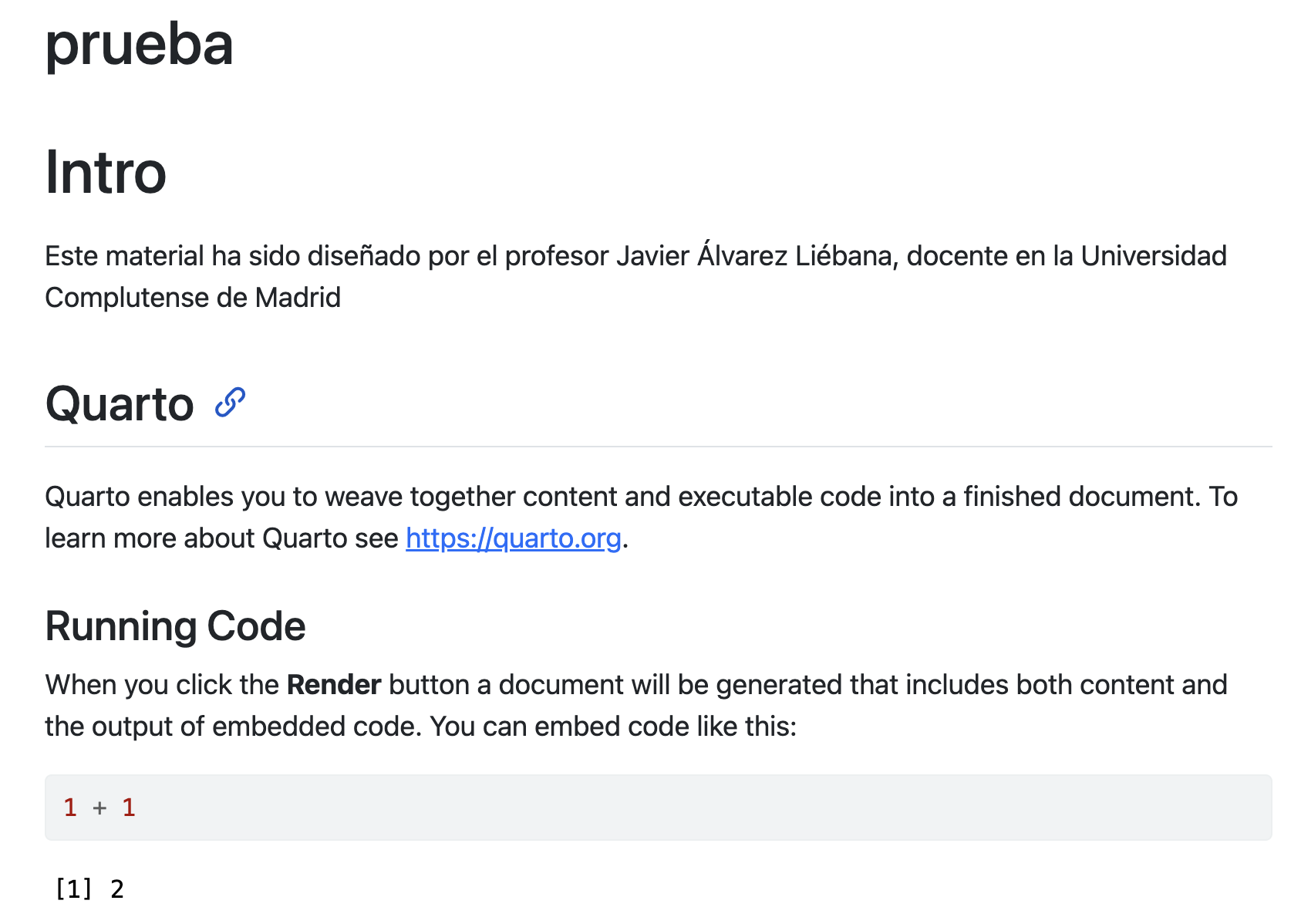
Vamos a empezar escribiendo una sección al inicio (# Intro y detrás por ej. la frase
Este material ha sido diseñado por el profesor Javier Álvarez Liébana, docente en la Universidad Complutense de Madrid
Además al Running Code le añadiremos una almohadilla #: las almohadillas FUERA DE CHUNKS nos servirán para crear epígrafes (secciones) en el documento
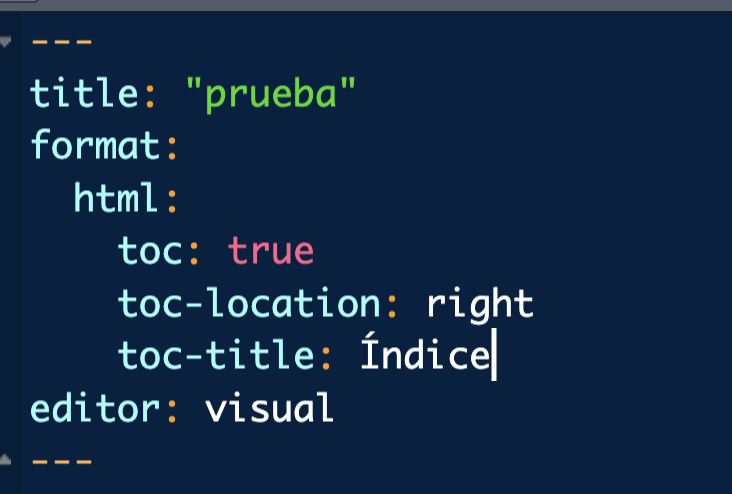

Para que el índice capture dichas secciones modificaremos la cabecera del archivo como se observa en la imagen (puedes cambiar la localización del índice y el título si quieres para probar).
Vamos a personalizar un poco el texto haciendo lo siguiente:
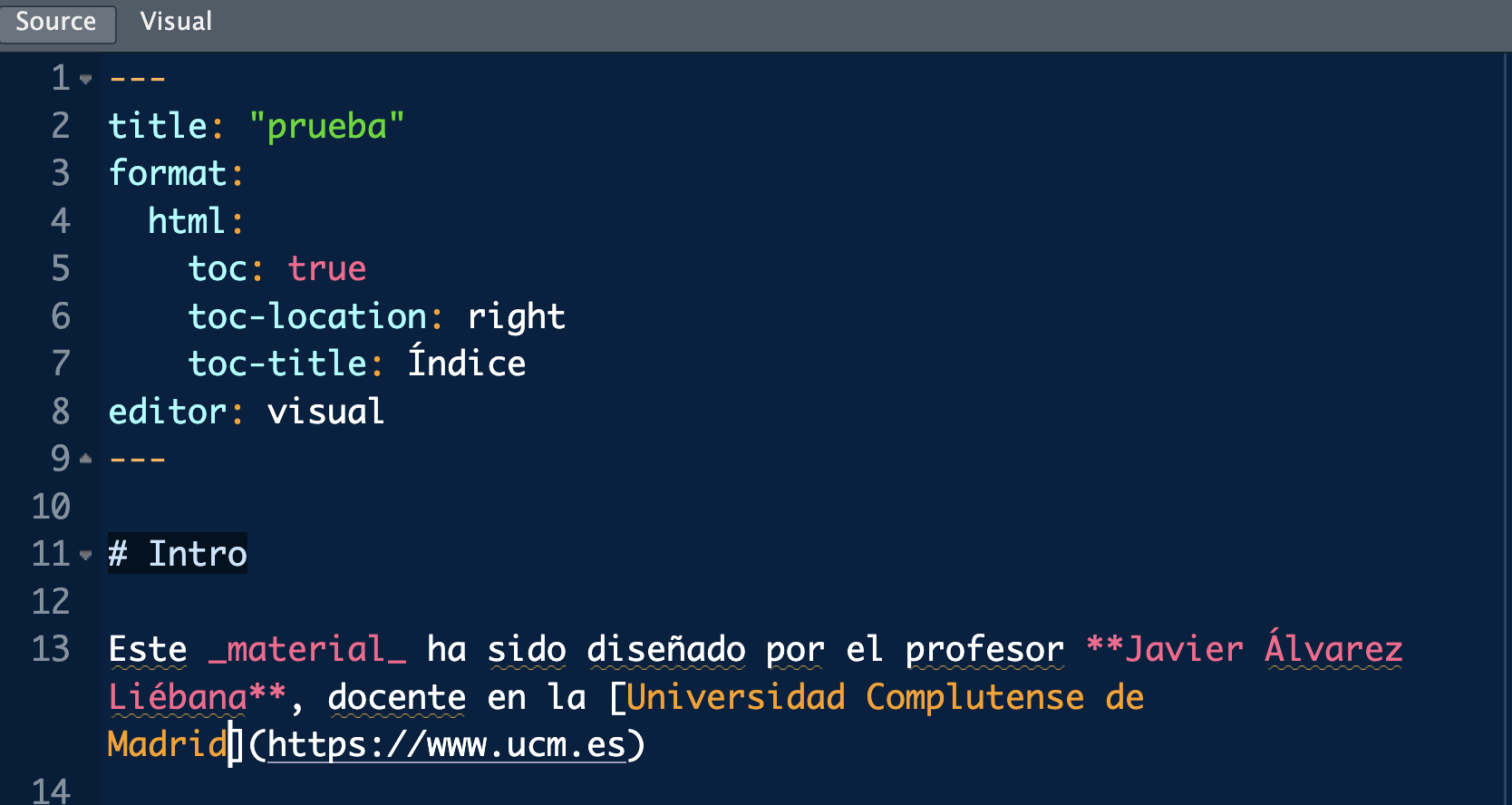

Vamos a añadir negrita al nombre (poniendo ** al inicio y al final).
Vamos añadir cursiva a la palabra material (poniendo _ al inicio y al final).
Vamos añadir un enlace https://www.ucm.es, asociándolo al nombre de la Universidad. Para ello el título lo ponemos entre corchetes y justo detrás el enlace entre paréntesis [«Universidad Complutense de Madrid»](https://www.ucm.es)
Para añadir código R debemos crear nuestras cajas de código llamadas chunks: altos en el camino en nuestro texto markdown donde podremos incluir código de casi cualquier lenguaje (y sus salidas).
Para incluir uno deberá de ir encabezado de la siguiente forma tienes un atajo Command + Option + I (Mac) o Ctrl + Shift + I (Windows)
Dentro de dicha cajita (que tiene ahora otro color en el documento) escribiremos código R como lo veníamos haciendo hasta ahora en los scripts.
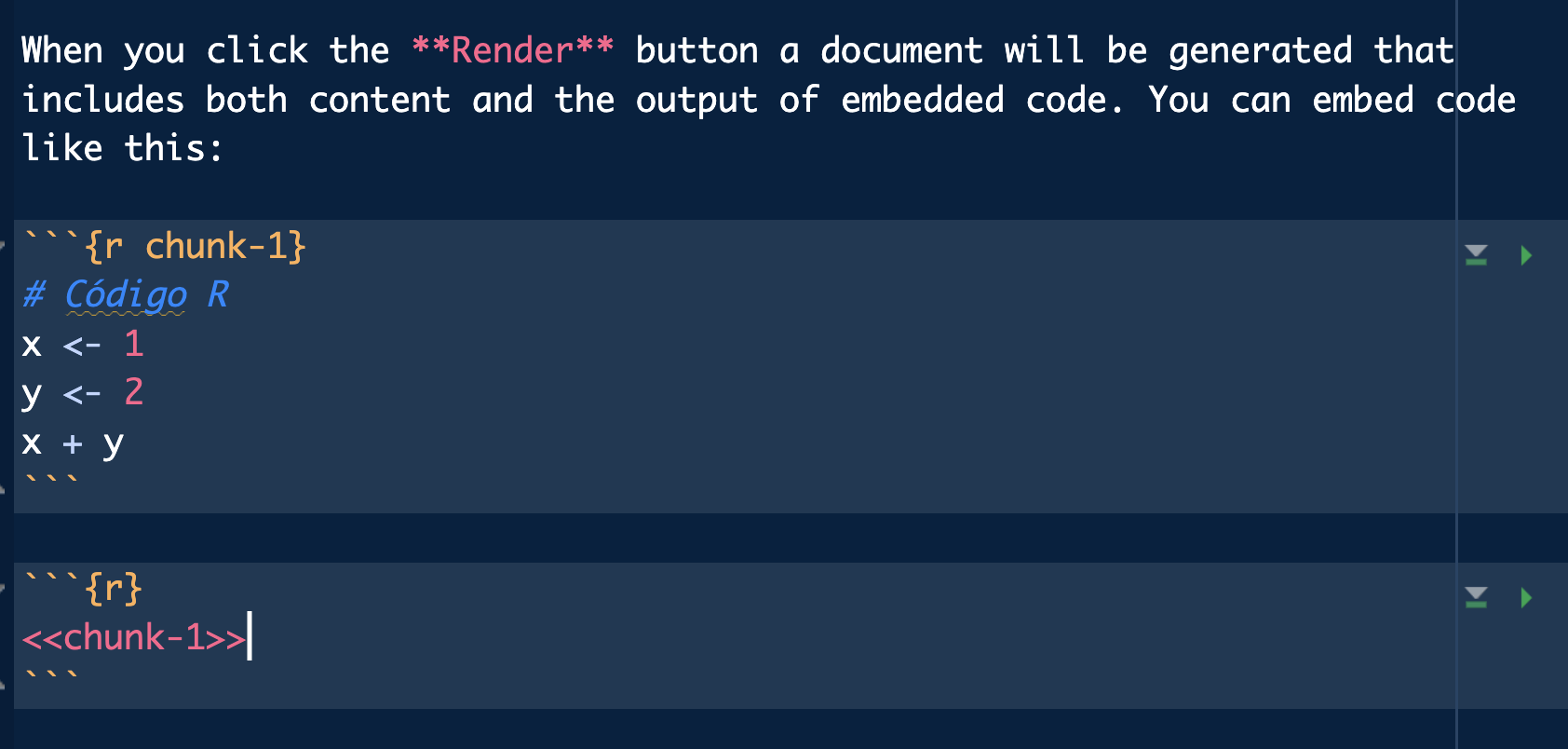
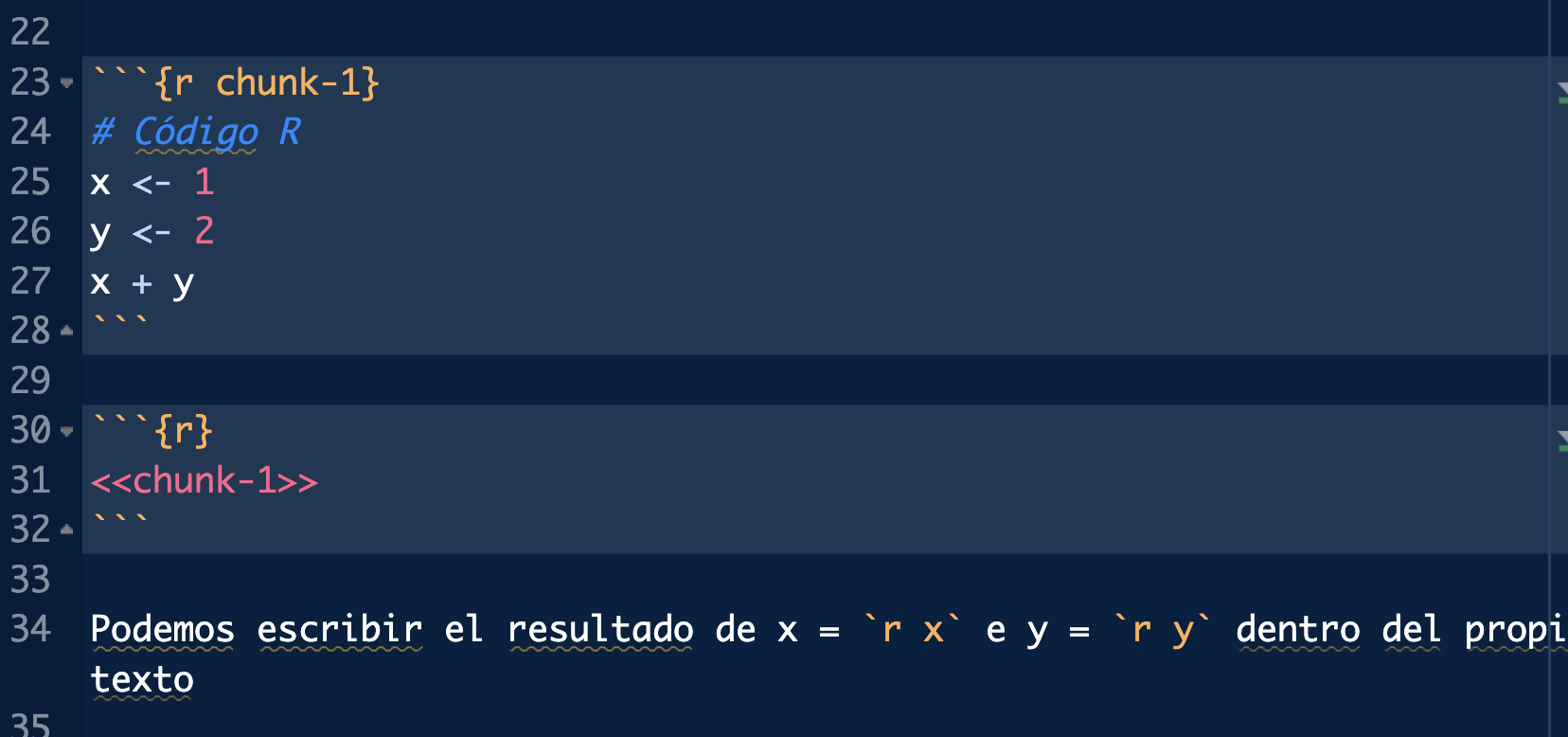
Los chunks pueden tener un nombre o etiqueta, de forma que podamos referenciarlos de nuevo para no repetir código.
En cada chunk aparecen dos botones:
botón de play: activa la ejecución y salida de ese chunk particular (lo puedes visualizar dentro de tu propio RStudio)
botón de rebobinar: activa la ejecución y salida de todos los chunk hasta ese (sin llegar a él)
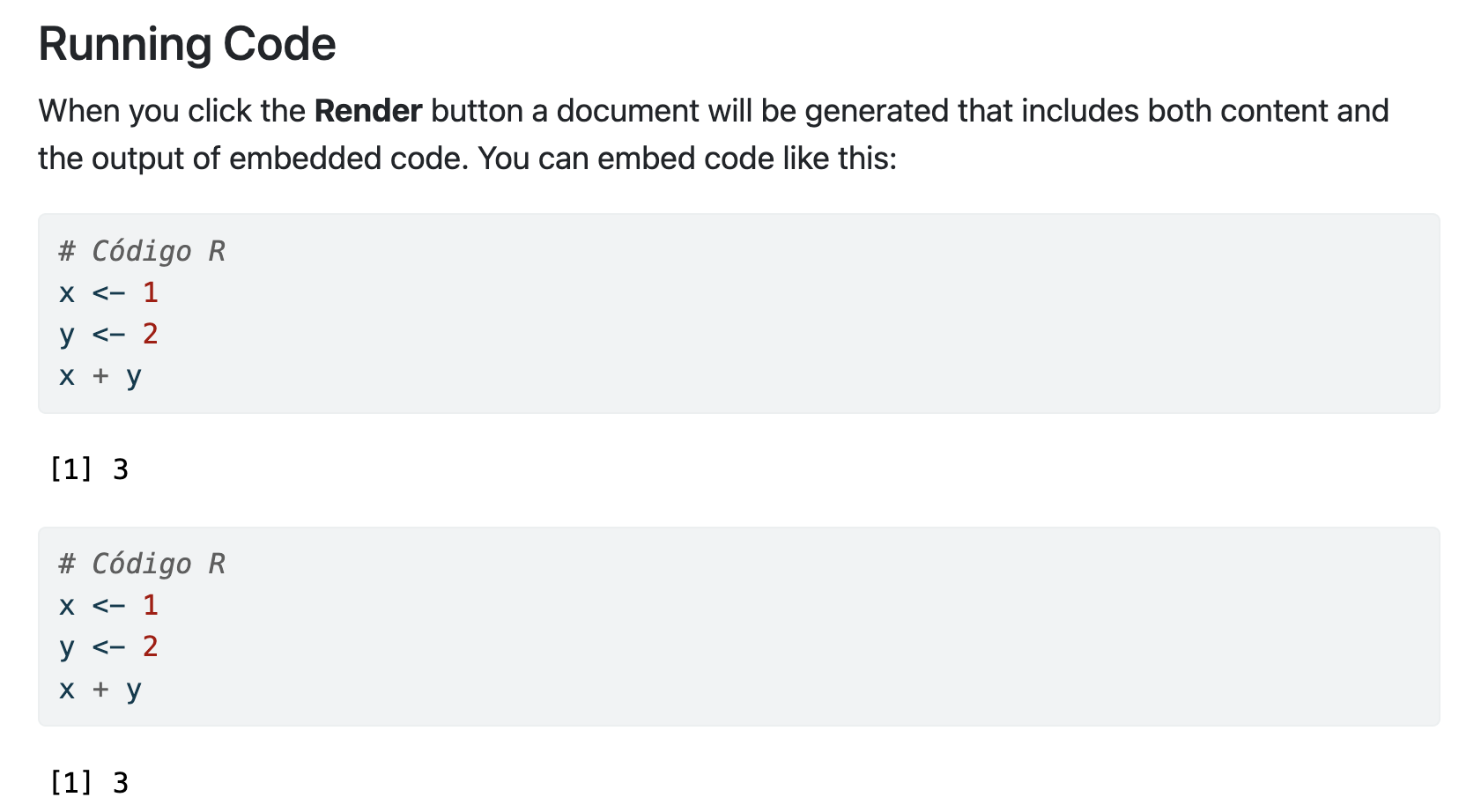
Además podemos incluir código R dentro de la línea de texto (en lugar de mostrar el texto x ejecuta el código R mostrando la variable).
Los chunks podemos personalizarlos con opciones al inicio del chunk precedido de #|:
#| echo: false: ejecuta código y se muestra resultado pero no visualiza código en la salida.
#| include: false: ejecuta código pero no muestra resultado y no visualiza código en la salida.
#| eval: false: no ejecuta código, no muestra resultado pero sí visualiza código en la salida.
#| message: false: ejecuta código pero no muestra mensajes de salida.
#| warning: false: ejecuta código pero no muestra mensajes de warning.
#| error: true: ejecuta código y permite que haya errores mostrando el mensaje de error en la salida.
Estas opciones podemos aplicarlas chunk a chunk o fijar los parámetros de forma global con knitr::opts_chunk$set() al inicio del documento (dentro de un chunk).
Además de texto y código podemos introducir lo siguiente:
Ecuaciones: puedes añadir además ecuaciones como \(x^2\) (he escrito $x^2$, la ecuación entre dólares).
Listas: puedes itemizar elementos poniendo *
* Paso 1: ...
* Paso 2: ...
{#nombre-seccion}) y llamarlas luego con [Sección](@nombre-seccion)
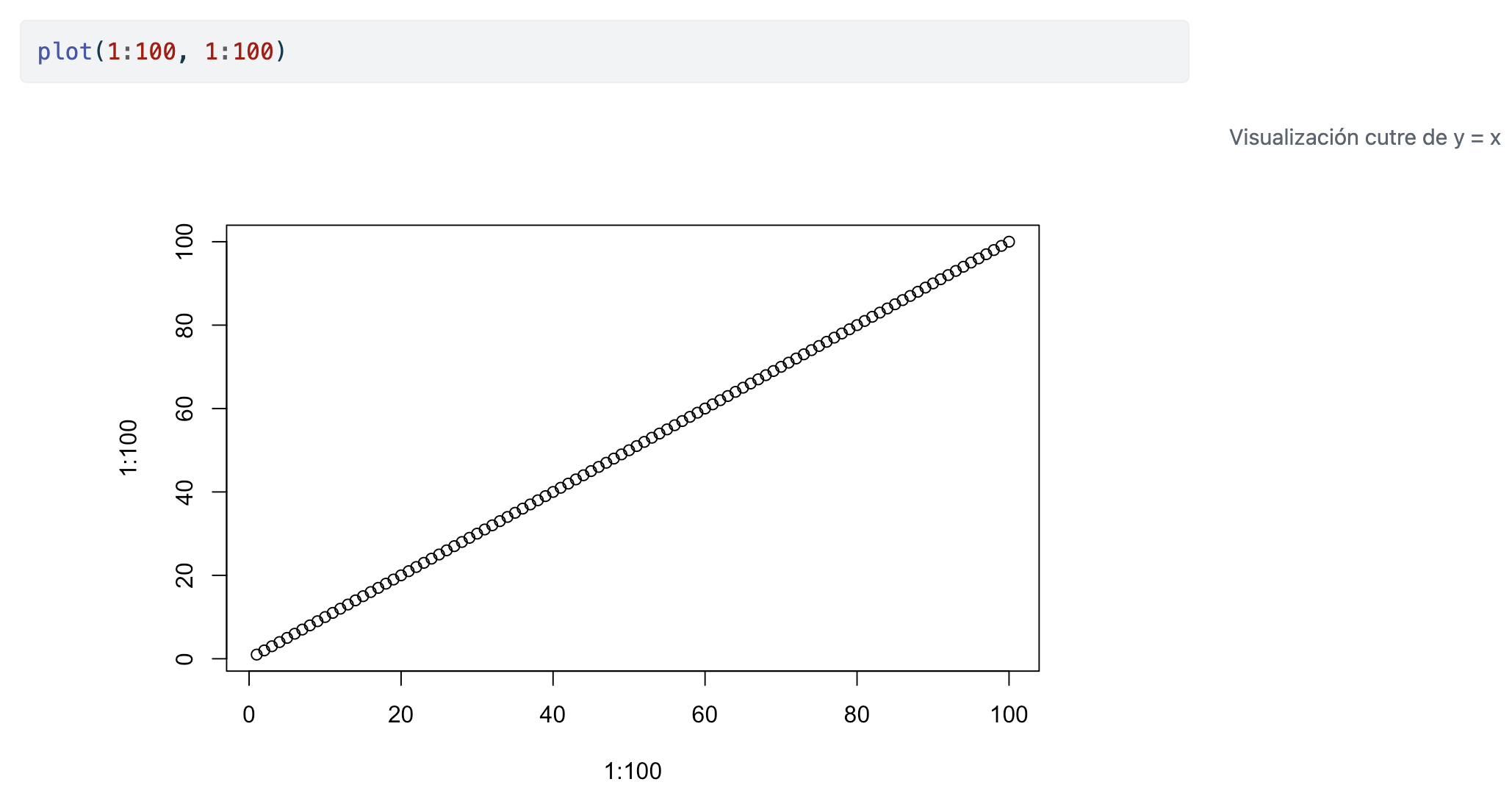
Por último, también podemos añadir pies de gráficas o imágenes añadiendo #| fig-cap: "..."
Fíjate que el caption está en el margen (por ejemplo). Puedes cambiarlo introduciendo ajustes en la cabecera (todo lo relativo a figuras empieza por fig-, y puedes ver las opciones tabulando). Tienes más información en https://quarto.org/
Por último puedes añadir un tema personalizado incluyendo un archivo de estilos (archivo en formato .scss o .css). Tenéis disponible un archivo de estilos en https://github.com/dadosdelaplace/seminario-R-biods.
Importante
El archivo de estilos debe estar en la misma carpeta que el archivo .qmd
El proceso es equivalente con tres cambios:
File > New File hay que hacer click en Quarto Presentation. La cabecera ahora será ahora parecido a esto (entre otras personalizaciones posibles)El proceso es equivalente con tres cambios:
. . . (tres puntos separados por espacios y un salto de línea)R sino revealjs)Importar/exportar datos en R
Hasta ahora solo hemos usado datos cargados ya en paquetes pero muchas veces necesitaremos importar datos de manera externa. Una de las principales fortalezas de R es que podemos importar datos de manera muy sencilla en distintos formatos:
.rda, .RData y .rds.csv y .tsv.txt.xls y .xlsx.sas7bdat, .sav y .datEl paquete {readr} dentro del entorno {tidyverse} contiene distintas funciones útiles para la carga de datos rectangulares (sin formatear).
read_csv(): archivos .csv cuyo separador sea la comaread_csv2(): punto y comaread_tsv(): tabulador.read_table(): espacio.read_delim(): función genérica para archivos delimitados por caracteres.
Todos necesitan como argumento la ruta del archivo amén de otros opcionales (saltar o no cabecera, decimales, etc). Ver más en https://readr.tidyverse.org/
La principal ventaja de {readr} es que automatiza el formateo para pasar de un archivo plano (sin formato) a un tibble (en filas y columnas, con formato).
.csv: con read_csv() cargaremos archivos separados por coma, pasando como argumento la ruta en file = .... Vamos a importar el dataset chickens.csv (sobre pollos de dibujos animados, why not). Si te fijas en la salida nos proporciona el tipo de variables.# A tibble: 5 × 4
chicken sex eggs_laid motto
<chr> <chr> <dbl> <chr>
1 Foghorn Leghorn rooster 0 That's a joke, ah say, that's a jok…
2 Chicken Little hen 3 The sky is falling!
3 Ginger hen 12 Listen. We'll either die free chick…
4 Camilla the Chicken hen 7 Bawk, buck, ba-gawk.
5 Ernie The Giant Chicken rooster 0 Put Captain Solo in the cargo hold. El formato de las variables normalmente lo hará read_csv() de forma automática, y podemos consultarlo con spec()
Aunque lo haga normalmente bien de forma automática podemos especificar el formato explícitamente en col_types = list() (en formato lista, con col_xxx() para cada tipo de variable, por ejemplo una la pondremos como cualitativa o factor).
chickens <-
read_csv(file = "./datos/chickens.csv",
col_types = list(col_character(), col_factor(), col_double(), col_character()))
chickens# A tibble: 5 × 4
chicken sex eggs_laid motto
<chr> <fct> <dbl> <chr>
1 Foghorn Leghorn rooster 0 That's a joke, ah say, that's a jok…
2 Chicken Little hen 3 The sky is falling!
3 Ginger hen 12 Listen. We'll either die free chick…
4 Camilla the Chicken hen 7 Bawk, buck, ba-gawk.
5 Ernie The Giant Chicken rooster 0 Put Captain Solo in the cargo hold. Incluso podemos indicar que variables que queremos seleccionar (sin ocupar memoria), indicándoselo en col_select = ...
Vamos a usar de nuevo read_csv() con el archivo massey-rating.txt.
# A tibble: 10 × 1
`UCC PAY LAZ KPK RT COF BIH DII ENG ACU Rank Team Conf`
<chr>
1 1 1 1 1 1 1 1 1 1 1 1 Ohio St B10
2 2 2 2 2 2 2 2 2 4 2 2 Oregon P12
3 3 4 3 4 3 4 3 4 2 3 3 Alabama SEC
4 4 3 4 3 4 3 5 3 3 4 4 TCU B12
5 6 6 6 5 5 7 6 5 6 11 5 Michigan St B10
6 7 7 7 6 7 6 11 8 7 8 6 Georgia SEC
7 5 5 5 7 6 8 4 6 5 5 7 Florida St ACC
8 8 8 9 9 10 5 7 7 10 7 8 Baylor B12
9 9 11 8 13 11 11 12 9 14 9 9 Georgia Tech ACC
10 13 10 13 11 8 9 10 11 9 10 10 Mississippi SEC Si te fijas nos interpreta todo como una sola columna: no tiene comas el archivo y no sabe por donde separar
¿Qué sucede cuando el separador no es el correcto?
Para ello tenemos
read_csv2() cuando el separador sea el punto y coma, read_tsv() cuando el sea un tabulador y read_table() cuando el sea un espacio
read_delim() en general
# A tibble: 10 × 13
UCC PAY LAZ KPK RT COF BIH DII ENG ACU Rank Team Conf
<dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <chr> <chr>
1 1 1 1 1 1 1 1 1 1 1 1 Ohio St
2 2 2 2 2 2 2 2 2 4 2 2 Oreg… P12
3 3 4 3 4 3 4 3 4 2 3 3 Alab… SEC
4 4 3 4 3 4 3 5 3 3 4 4 TCU B12
5 6 6 6 5 5 7 6 5 6 11 5 Mich… St
6 7 7 7 6 7 6 11 8 7 8 6 Geor… SEC
7 5 5 5 7 6 8 4 6 5 5 7 Flor… St
8 8 8 9 9 10 5 7 7 10 7 8 Bayl… B12
9 9 11 8 13 11 11 12 9 14 9 9 Geor… Tech
10 13 10 13 11 8 9 10 11 9 10 10 Miss… SEC Otro de los paquetes fundamentales de importación será el paquete {readxl} para importar datos desde una Excel. Tres funciones serán claves:
read_xls() específica para .xls, read_xlsx() específica para .xlsxread_excel(): para ambasVamos a importar deaths.xlsx con registros de fallecimientos de famosos
# A tibble: 8 × 6
`Lots of people` ...2 ...3 ...4 ...5 ...6
<chr> <chr> <chr> <chr> <chr> <chr>
1 simply cannot resist writing <NA> <NA> <NA> <NA> some not…
2 at the top <NA> of their sp…
3 or merging <NA> <NA> <NA> cells
4 Name Profession Age Has kids Date of birth Date of …
5 David Bowie musician 69 TRUE 17175 42379
6 Carrie Fisher actor 60 TRUE 20749 42731
7 Chuck Berry musician 90 TRUE 9788 42812
8 Bill Paxton actor 61 TRUE 20226 42791 # A tibble: 8 × 6
`Lots of people` ...2 ...3 ...4 ...5 ...6
<chr> <chr> <chr> <chr> <chr> <chr>
1 simply cannot resist writing <NA> <NA> <NA> <NA> some not…
2 at the top <NA> of their sp…
3 or merging <NA> <NA> <NA> cells
4 Name Profession Age Has kids Date of birth Date of …
5 David Bowie musician 69 TRUE 17175 42379
6 Carrie Fisher actor 60 TRUE 20749 42731
7 Chuck Berry musician 90 TRUE 9788 42812
8 Bill Paxton actor 61 TRUE 20226 42791 Algo por desgracia muy habitual es que haya algún tipo de comentario o texto al inicio del archivo, teniendo que saltarnos dichas filas.
Podemos saltarnos dichas filas directamente en la carga con skip = ... (indicando el número de filas que nos saltamos)
# A tibble: 5 × 6
Name Profession Age `Has kids` `Date of birth` `Date of death`
<chr> <chr> <chr> <chr> <dttm> <chr>
1 David Bowie musician 69 TRUE 1947-01-08 00:00:00 42379
2 Carrie Fisher actor 60 TRUE 1956-10-21 00:00:00 42731
3 Chuck Berry musician 90 TRUE 1926-10-18 00:00:00 42812
4 Bill Paxton actor 61 TRUE 1955-05-17 00:00:00 42791
5 Prince musician 57 TRUE 1958-06-07 00:00:00 42481 Además con col_names = ... podemos renombrar ya las columnas en la importación (si proporcionamos nombres asume la 1ª línea ya como un dato)
# A tibble: 7 × 6
name profession age kids birth death
<chr> <chr> <chr> <chr> <dttm> <chr>
1 David Bowie musician 69 TRUE 1947-01-08 00:00:00 42379
2 Carrie Fisher actor 60 TRUE 1956-10-21 00:00:00 42731
3 Chuck Berry musician 90 TRUE 1926-10-18 00:00:00 42812
4 Bill Paxton actor 61 TRUE 1955-05-17 00:00:00 42791
5 Prince musician 57 TRUE 1958-06-07 00:00:00 42481
6 Alan Rickman actor 69 FALSE 1946-02-21 00:00:00 42383
7 Florence Henderson actor 82 TRUE 1934-02-14 00:00:00 42698En ocasiones las fechas de Excel están mal formateadas (sorpresa): podemos hacer uso de convertToDate() del paquete {openxlsx} para convertirlo
# A tibble: 7 × 6
name profession age kids birth death
<chr> <chr> <chr> <chr> <dttm> <date>
1 David Bowie musician 69 TRUE 1947-01-08 00:00:00 2016-01-10
2 Carrie Fisher actor 60 TRUE 1956-10-21 00:00:00 2016-12-27
3 Chuck Berry musician 90 TRUE 1926-10-18 00:00:00 2017-03-18
4 Bill Paxton actor 61 TRUE 1955-05-17 00:00:00 2017-02-25
5 Prince musician 57 TRUE 1958-06-07 00:00:00 2016-04-21
6 Alan Rickman actor 69 FALSE 1946-02-21 00:00:00 2016-01-14
7 Florence Henderson actor 82 TRUE 1934-02-14 00:00:00 2016-11-24También podemos cargar un Excel con varias hojas: para indicarle la hoja (bien por su nombre bien por su número) usaremos el argumento sheet = ...
# A tibble: 5 × 11
mpg cyl disp hp drat wt qsec vs am gear carb
<dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 21 6 160 110 3.9 2.62 16.5 0 1 4 4
2 21 6 160 110 3.9 2.88 17.0 0 1 4 4
3 22.8 4 108 93 3.85 2.32 18.6 1 1 4 1
4 21.4 6 258 110 3.08 3.22 19.4 1 0 3 1
5 18.7 8 360 175 3.15 3.44 17.0 0 0 3 2El paquete {haven} dentro de la órbita tidyverse nos permitirá importar archivos de los 3 software de pago más importantes: SAS, SPSS y Stata
De la misma manera que podemos importar también podemos exportar
.RData (opción recomendada para variables guardadas en R). Recuerda que esta extensión solo se podrá usar en R. Para ello nos basta con usar save(objeto, file = ruta)De la misma manera que podemos importar también podemos exportar
.RDS (opción recomendada para variables guardadas en R). Recuerda que esta extensión solo se podrá usar en R. Para ello nos basta con usar saveRDS(objeto, file = ruta)De la misma manera que podemos importar también podemos exportar
.csv. Para ello nos basta con usar write_csv(objeto, file = ruta)De la misma manera que podemos importar también podemos exportar
.xlsx. Para ello nos basta con usar write.xlsx(objeto, file = ruta) del paquete {openxlsx}Una de las principales ventajas de R es que podemos hacer uso de todas las funciones anteriores de importar pero directamente desde una web, sin necesidad de realizar la descarga manual: en lugar de pasarle la ruta local le indicaremos el enlace. Por ejemplo, vamos a descargar los datos de covid del ISCIII (https://cnecovid.isciii.es/covid19/#documentaci%C3%B3n-y-datos)
# A tibble: 700 × 8
provincia_iso sexo grupo_edad fecha num_casos num_hosp num_uci num_def
<chr> <chr> <chr> <date> <dbl> <dbl> <dbl> <dbl>
1 A H 0-9 2020-01-01 0 0 0 0
2 A H 10-19 2020-01-01 0 0 0 0
3 A H 20-29 2020-01-01 0 0 0 0
4 A H 30-39 2020-01-01 0 0 0 0
5 A H 40-49 2020-01-01 0 0 0 0
6 A H 50-59 2020-01-01 0 0 0 0
7 A H 60-69 2020-01-01 0 0 0 0
8 A H 70-79 2020-01-01 0 0 0 0
9 A H 80+ 2020-01-01 0 0 0 0
10 A H NC 2020-01-01 0 0 0 0
# ℹ 690 more rowsOtra opción disponible (sobre todo si trabajamos con otras personas que trabajan) es importar desde una hoja de cálculo Google Drive, haciendo uso de read_sheet() del paquete {googlesheets4}
La primera vez te pedirá un permiso de tidyverse para interactuar con vuestro drive
Intenta realizar los siguientes ejercicios sin mirar las soluciones
📝 El dataset who que hemos usado en ejercicios anteriores, expórtalo a un formato nativo de R en la carpeta datos del proyecto
📝 Carga el dataset who pero desde la carpeta de datos (importa el archivo creado en el ejercicio anterior)
📝 Repite lo mismo (exportar e importar) en 4 formatos: .csv, .xlsx, .sav (spss) y .dta (stata)
# csv
library(readr)
write_csv(who, file = "./datos/who.csv")
who_data <- read_csv(file = "./datos/who.csv")
# excel
library(openxlsx)
write.xlsx(who, file = "./datos/who.xlsx")
who_data <- read_xlsx(path = "./datos/who.xlsx")
# sas y stata
library(haven)
write_sav(who, path = "./datos/who.sav")
who_data <- read_spss(path = "./datos/who.sav")
write_dta(who, path = "./datos/who.dta")
who_data <- read_dta(path = "./datos/who.dta")Uso de listas: paquete purrr
Veamos un pequeño resumen de los datos que ya conocemos:
vectores: colección de elementos de igual tipo. Pueden ser números, caracteres o valores lógicos, entre otros.
matrices: colección BIDIMENSIONAL de elementos de igual tipo e igual longitud.
data.frame / tibble: colección BIDIMENSIONAL de elementos de igual longitud pero de cualquier tipo.
Las listas serán colecciones de variables de diferente tipo y diferente longitud, con estructuras totalmente heterógeneas (incluso una lista puede tener dentro a su vez otra lista).
Una opción más flexible y versatil que la familia lapply es hacer uso del paquete {purrr} ya cargado dentro del entorno {tidyverse}
Este paquete está pensado para imitar la programa funcional de otros lenguajes orientados Big Data como Scala o Hadoop (de Google).
La función más simple de {purrr} es conocida como la función map(), encargada de aplicar una función a una lista de manera vectorial, elemento a elemento. . . .
Por ejemplo, con map() podemos “mapear” cada lista y aplicar la función raíz cuadrada a cada elemento de la lista.
$x1
[1] 1.000000 1.414214 1.732051 2.000000
$x2
[1] 3.316625 3.464102 3.605551 3.741657 3.872983 4.000000 4.123106 4.242641
[9] 4.358899 4.472136Be careful
Con R tenemos una vectorización automática cuando hablamos de vectores ya que trabaja elemento-a-elemento, pero por defecto, el output de map es una lista.
Otro ejemplo: definamos dos distribuciones normales, con diferente \(n\). ¿Cómo calcular la media de cada una de manera inmediata?
Lo mismo si queremos usar cualquier otra función que queramos definir nosotros mismos (por ejemplo, la media de los cuadrados)
Además de ser más legible y eficiente, con {purrr} podemos decidir el formato de salida de manera sencilla
map_dbl()map_int()map_chr()map_lgl()Además si incluyes como argumento un número en lugar de una función, nos devolverá el i-ésimo elemento de la lista.
También nos permite la opción de generalizarlo para funciones que necesiten dos argumentos de entrada (operaciones binarias) con map2()
El output podemos también obtenerlo en formato tibble añadiendo un list_rbind() o list_cbind() para concatenar los valores d euna lista en formato tabular.
Lo anterior se puede generalizar con pmap_xxx() lo que nos permite aplicar funciones con múltiples argumentos.
Dentro del paquete contamos con otro tipo de [iteradores que no devuelven nada] (solo side-effects) como es el caso de walk() (solo un argumento de entrada), walk2() (dos argumentos) and pwalk() (multiples argumentos), todos ellos con un return invisible
Recientemente se ha desarrollado {loopurrr}, un paquete que permite la traducción con as_loop() de lenguaje funcional en {purrr} a bucles para poder entender mejor lo que hace (ver más en https://timteafan.github.io/loopurrr/)
Cruzando datos
Al trabajar con datos no siempre tendremos la información en una sola tabla y a veces nos interesará cruzar la información de distintas fuentes.
Para ello usaremos un clásico de todo lenguaje que maneja datos: los famosos join, una herramienta que nos va a permitir cruzar una o variables tablas, haciendo uso de una columna identificadora de cada una de ellas (por ejemplo, imagina que cruzamos datos de hacienda y de antecedentes penales, haciendo join por la columna DNI).
inner_join(): solo sobreviven los registros con id en ambas tablas.
full_join(): mantiene todos los registros de ambas tablas.
left_join(): mantiene todos los registros de la primera tabla, y busca cuales tienen id también en la segunda (en caso de no tenerlo se rellena con NA los campos de la 2ª tabla).
right_join(): mantiene todos los registros de la segunda tabla, y busca cuales tienen id también en la primera.
Vamos a probar los distintos joins con un ejemplo sencillo
Imagina que queremos incorporar a tb_1 la información de la tabla_2, identificando los registros por la columna key (indicando con by = "key" la columna por la que tiene que cruzar): queremos mantener todos los registros de la primera tabla y buscar cuales tienen id (mismo valor en key) también en la segunda tabla.
# A tibble: 3 × 3
key val_x val_y
<dbl> <chr> <chr>
1 1 x1 y1
2 2 x2 y2
3 3 x3 <NA> Fíjate que los registros de la primera cuya key no ha encontrado en la segunda les ha dado el valor de ausente.
El right_join() realizará la operación contraria: vamos ahora a incorporar a tb_2 la información de la tabla_2, identificando los registros por la columna key (indicando con by = "key" la columna por la que tiene que cruzar): queremos mantener todos los registros de la segunda y buscar cuales tienen id (mismo valor en key) también en la primera tabla.
# A tibble: 3 × 3
key val_x val_y
<dbl> <chr> <chr>
1 1 x1 y1
2 2 x2 y2
3 4 <NA> y3 Fíjate que ahora los registros de la segunda cuya key no ha encontrado en la primera son los que les ha dado el valor de ausente.
Las columnas clave que usaremos para el cruce no siempre se llamarán igual.
by = c("key_2" = "key_2"): le indicaremos en qué columna de cada tabla están las claves por las que vamos a cruzar.Además podemos cruzar por varias columnas a la vez (interpretará como igual registro aquel que tenga el conjunto de claves igual), con by = c("var1_t1" = "var1_t2", "var2_t1" = "var2_t2", ...). Modifiquemos el ejemplo anterior
También podría suceder que al cruzar dos tablas, haya columnas de valores que se llamen igual
Dicho sufijo podemos especificárselo en el argumento opcional suffix = ..., que nos permita distinguir las variables de una tabla y de otra.
Los dos anteriores casos forman lo que se conoce como outer joins: cruces donde se mantienen observaciones que salgan en al menos una tabla. El tercer outer join es el conocido como full_join() que nos mantendrá las observaciones de ambas tablas, añadiendo las filas que no casen con la otra tabla.
Frente a los outer join está lo que se conoce como inner join, con inner_join(): un cruce en el que solo se mantienen las observaciones que salgan en ambas tablas, solo mantiene aquellos registros matcheados.
Fíjate que en términos de registros, inner_join si es conmutativa, nos da igual el orden de las tablas: lo único que cambia es el orden de las columnas que añade.
Por último tenemos dos herramientas interesantes para filtrar (no cruzar) registros: semi_join() y anti_join(). El semi join nos deja en la primera tabla los registros que cuya clave está también en la segunda (como un inner join pero sin añadir la info de la segunda tabla). Y el segundo, los anti join, hace justo lo contrario (aquellos que no están).
Intenta realizar los siguientes ejercicios sin mirar las soluciones
Para los ejercicios usaremos las tablas disponibles en el paquete {nycflights13} (echa un vistazo antes)
Intenta realizar los siguientes ejercicios sin mirar las soluciones
📝 Del paquete {nycflights13} cruza la tabla flights con airlines. Queremos mantener todos los registros de vuelos, añadiendo la información de las aerolíneas a la tabla de aviones.
📝 A la tabla obtenida del cruce del apartado anterior, cruza después con los datos de los aviones en planes, pero incluyendo solo aquellos vuelos de los que tengamos información de sus aviones (y viceversa).
📝 Repite el ejercicio anterior pero conservando ambas variables year (en una es el año del vuelo, en la otra es el año de construcción del avión), y distinguiéndolas entre sí
📝 Al cruce obtenido del ejercicio anterior incluye la longitud y latitud de los aeropuertos en airports, distinguiendo entre la latitud/longitud del aeropuerto en destino y en origen.
📝 Filtra de airports solo aquellos aeropuertos de los que salgan vuelos. Repite el proceso filtrado solo aquellos a los que lleguen vuelos
Trabajando con variables cualitativas
En el caso de las variables cualitativas, llamaremos niveles o modalidades a los diferentes valores que pueden tomar estos datos. Por ejemplo, en el caso de la variable sex del conjunto starwars, tenemos 4 niveles permitidos: female, hermaphroditic, male y none (amén de datos ausentes).
Este tipo de variables se conocen en R como factores. Y el paquete fundamental para tratarlos es {forcats} (del entorno {tidyverse}).
Este paquete nos permite fijar los niveles (guardados internamente como levels) que toma una determinada variable categórica, dándoles un tratamiento diferente a las cadena de texto normales.
Veamos un ejempo sencillo definiendo una variable estado que tome los valores "sano", "leve" y "grave" de la siguiente manera.
estado <-
c("leve", "grave", "sano", "sano", "leve", "sano", "sano", "grave",
"grave", "leve", "grave", "sano", "sano")
estado [1] "leve" "grave" "sano" "sano" "leve" "sano" "sano" "grave" "grave"
[10] "leve" "grave" "sano" "sano" La variable estado actualmente es de tipo texto, de tipo chr, algo que podemos comprobar con class(estado).
Desde un punto de vista estadístico y computacional, para R esta variable ahora mismo sería equivalente una variable de nombres. Pero estadísticamente no es lo mismo una variable con nombres (que identifican muchas veces el registro) que una variable categórica como estado que solo puede tomar esos 3 niveles. ¿Cómo convertir a factor?
Haciendo uso de la función as_factor() del paquete {forcats}.
No solo ha cambiado la clase de la variable sino que ahora, debajo del valor guardado, nos aparece la frase Levels: grave leve sano: son las modalidades o niveles de nuestra cualitativa.
Imagina que ese día en el hospital no tuviésemos a nadie en estado grave: aunque ese día nuestra variable no tome dicho valor, el estado grave es un nivel permitido en la base de datos, así que aunque lo eliminemos, por ser un factor, el nivel permanece (no lo tenemos ahora pero es un nivel permitido).
Con factor() podemos especificar explícitamente los nombres de las modalidades, incluso si son nominales u ordinales
Con levels = ... podemos indicarle explícitamente el orden de las modalidades
Si queremos indicarle que elimine un nivel no usado en ese momento (y que queremos excluir de la definición) podemos hacerlo con fct_drop()
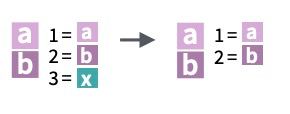
Al igual que podemos eliminar niveles podemos ampliar los niveles existentes (aunque no existan datos de ese nivel en ese momento) con fct_expand()
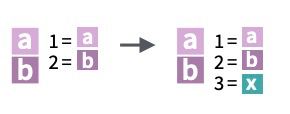
Además con fct_explicit_na() podemos asignar un nivel a los valores para que sea incluido dicho nivel en los análisis y visualizaciones.
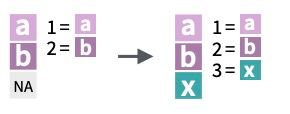
Incluso una vez definidos podemos reordenar los níveles con fct_relevel()
estado_fct_expand <-
estado_fct |>
mutate(estado = fct_expand(estado, c("UCI", "fallecido"))) |>
pull(estado)
estado_fct_expand |>
fct_relevel(c("fallecido", "leve", "sano", "grave", "UCI")) [1] leve grave sano sano leve sano sano grave grave leve grave sano
[13] sano
Levels: fallecido < leve < sano < grave < UCIEsta forma de trabajar con variables cualitativas nos permite dar una definición teórica de nuestra base de datos, pudiendo incluso contar valores que aún no existen (pero que podrían), haciendo uso de fct_count()
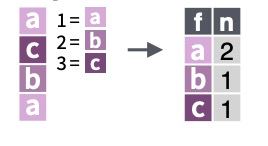
Los níveles también podemos ordenarlos por frecuencia con fct_infreq()
A veces querremos agrupar niveles, por ejemplo, no permitiendo niveles que no sucedan un mínimo de veces con fct_lump_min(.., min = ..) (las observaciones que no lo cumplan irán a un nivel genérico llamado Other, aunque se puede cambiar con el argumento other_level).
Podemos hacer algo equivalente pero en función de su frecuencia relativa con fct_lump_prop().
Esto lo podemos aplicar a nuestros conjuntos de datos para recategorizar variables de forma muy rápida.
Con fct_reorder() podemos también indicar que queremos ordenar los factores en función de una función aplicada a otra variable.
[1] Human Droid Droid Human Human Human Human Droid Human Human
[11] Human Human Otras Human Otras Otras Human Human Otras Human
[21] Human Droid Otras Human Human Otras Human Otras Otras Human
[31] Otras Human Gungan Gungan Gungan Otras Otras Human Otras Otras
[41] Otras Otras Otras Otras Human Otras Otras Otras Otras Otras
[51] Otras Otras Otras Human Human Human Otras Otras Otras Human
[61] Human Human Human Otras Otras Otras Otras Human Otras Droid
[71] Otras Otras Otras Otras Otras Human Otras Human
Levels: Droid Gungan Human Otras [1] Human Droid Droid Human Human Human Human Droid Human Human
[11] Human Human Otras Human Otras Otras Human Human Otras Human
[21] Human Droid Otras Human Human Otras Human Otras Otras Human
[31] Otras Human Gungan Gungan Gungan Otras Otras Human Otras Otras
[41] Otras Otras Otras Otras Human Otras Otras Otras Otras Otras
[51] Otras Otras Otras Human Human Human Otras Otras Otras Human
[61] Human Human Human Otras Otras Otras Otras Human Otras Droid
[71] Otras Otras Otras Otras Otras Human Otras Human
Levels: Droid Otras Human GunganIntenta realizar los siguientes ejercicios sin mirar las soluciones
📝 Dada la variable meses definida debajo (definida como un vector de caracteres), convierte dicha variable a factor (solo eso)
📝 Dada la variable meses definida debajo convierte dicha variable a factor pero indicando los niveles de forma correcta.
meses <- c(NA, "Abr", "Ene", "Oct", "Jul", "Ene", "Sep", NA, "Feb", "Dic",
"Jul", "Mar", "Ene", "Mar", "Feb", "Abr", "May", "Oct", "Sep", NA,
"Dic", "Jul", "Nov", "Feb", "Oct", "Jun", "Sep", "Oct", "Oct", "Sep")
# Orden de niveles correcto e incluimos agosto aunque no haya
meses_fct <-
factor(meses,
levels = c("Ene", "Feb", "Mar", "Abr", "May", "Jun", "Jul", "Ago", "Sep", "Oct", "Nov", "Dic"))
meses_fct📝 Cuenta cuantos valores hay de cada mes pero teniendo en cuenta que son factores (quizás haya niveles sin ser usados y de los que debería obtener un 0).
📝 Dado que hay ausentes, indica que los ausentes sea un decimotercer nivel etiquetado como “ausente”.
Trabajar ordenados, publicar resultados, replicabilidad de lo realizado
GitHub es la plataforma colaborativa más conocida basada en el sistema de control de versiones Git
Importante
Desde el 4 de junio de 2018 Github es de Microsoft (ergo el código que subas también)
Tras hacernos una cuenta en Github, arriba a la derecha tendremos un círculo, y haciendo click en Your Profile, veremos algo similar a esto
Edit profile: nos permite añadir una descripción y foto de perfil.
Overview: en ese panel de cuadrados se visualizará nuestra actividad a lo largo del tiempo.
Repositories: el códugo será subido a repositorios, el equivalente a nuestras carpetas compartidas en Dropbox.
Antes de aprender como crear repositorios, Github también nos servirá para
El código de paquetes que no tengamos subido en CRAN podremos instalarlo como código desde Github
Por ejemplo, vamos a instalar un paquete llamado {peRReo}, cuya única función es darnos paletas de colores basadas en portadas de álbumes de música urbana

Las instrucciones de instalación suelen venir detalladas en la portada del repositorio

La mayoría de veces lo que subamos no será un paquete de R como tal sino que subiremos un código más o menos organizado y comentado. En ese caso podremos descargar el repo entero haciendo click Code y luego Download ZIP.
Por ejemplo, vamos a descargarnos los scripts de dataviz que han subido desde el Centre d’Estudis d’Opinió
¿Lo ideal? Tener dos tipos de repositorios
Una colección de repositorios públicos (producción) donde hacer transparente el código y los datos (ya validados), coordinado por un nº reducido de personas.
Una colección de repositorios privados (desarrollo) donde esté todo el equipo colaborando y donde se haga el trabajo del día, con trazabilidad interna.
Vamos a crear nuestro primero repositorio que servirá además como carta de presentación de nuestro perfil en Github.
Repositories: hacemos click en las pestaña de Repositories.
New: hacemos click en el botón verde New para crear un nuevo repositorio
Repository name: el nombre del repositorio. En este caso vamos a crear un repositorio muy concreto: el nombre debe coincidir exactamente con tu nombre de usuario
Description: descripción de tu repositorio. En este caso será un repo de presentación.
Public vs private: con cada repositorio tendremos la opción de hacer el repositorio
En este caso concreto, dado que será un repositorio de presentación, lo haremos público.
{peRReo} era el archivo que contenía los detalles de instalación)De momento ignoraremos los demás campos para este primer repositorio.
Por defecto Github asume que este repositorio, con el mismo nombre que nuestro usuario será el repositorio que querremos que se presente de inicio cuando alguien entra en nuestro perfil, y será el repositorio donde [incluir en el README.md] una presentación de nosotros y un índice de tu trabajo (si quieres).
Fíjate que ahora en nuestra portada tenemos dicho README.md que podemos personalizar a nuestro gusto haciendo uso de html y markdown.
Aquí puedes ver algunos ejemplos de README.MD
Una vez que tenemos nuestro README de presentación (recuerda que puedes personalizar a tu gusto con html y markdown) vamos a crear un repositorio de código.
Si ya era importante trabajar con proyectos en RStudio, cuando lo combinamos con Github es aún más crucial que creemos un proyecto antes de subir el código, así que vamos a crear uno de prueba que se llame repo-github-1.
En dicho proyecto vamos a crear un script (en mi caso llamado codigo.R) en el que deberás hacer los siguientes pasos:
casos_hosp_uci_def_sexo_edad_provres.csv"M"), de 2020 y con sexo conocido (hombre/mujer). Tras ello quédate con las columnas fecha, sexo, grupo_edad, num_casos (ese orden). Por último obtén la suma de casos diarios por fecha y sexo.# Depuración
datos_madrid <-
datos_covid |>
# Filtrado por Madrid y fecha
filter(provincia_iso == "M" & fecha <= "2020-12-31" & sexo != "NC") |>
# Selección de columnas
select(provincia_iso:fecha, num_casos) |>
# Resumen de casos diarios por fecha y sexo
summarise(num_casos = sum(num_casos), .by = c(fecha, sexo))exportado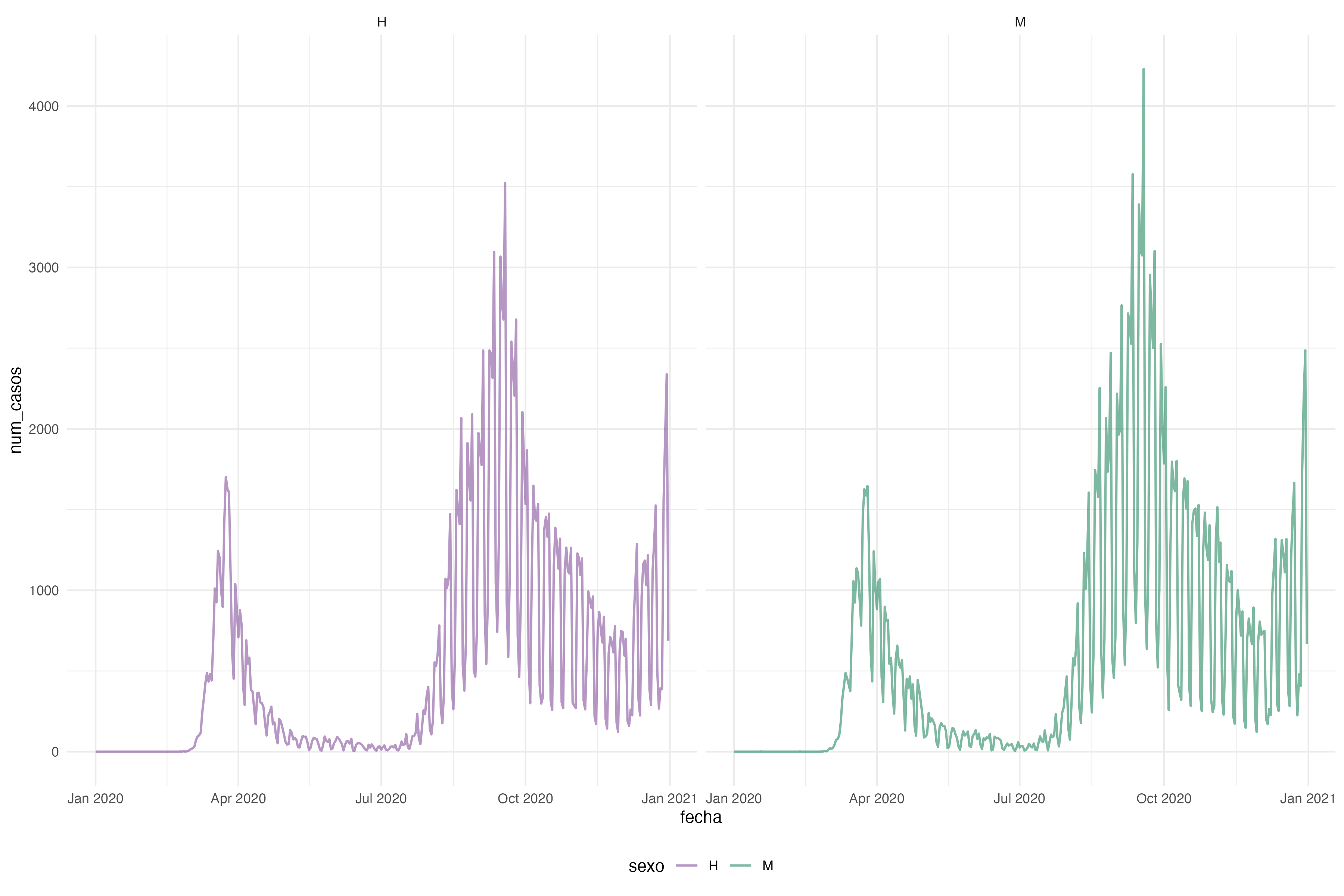
¿Cómo subimos el proyecto? Vamos de nuevo a crear un proyecto de cero. Antes no hemos hablado de dos campos importantes:
Add .gitignore nos permitirá seleccionar el lenguaje en el que estará nuestro proyecto para que Github lo entienda al sincronizar (y no actualice cosas que no deba).
Choose a license nos permitirá seleccionar la licencia que determinará las condiciones en las que otros podrán reusar tu código.
Si te fijas traer crearlo tenemos solo 3 archivos: el de licencia, el .gitignore y el readme.md (donde deberíamos escribir una guía de uso de lo que hayamos subido)
Para subir los archivos vamos a clickar en Add file < Upload File y arrastraremos TODOS los archivos de la carpeta de nuestro proyecto.
Tras la subida de archivos tendremos un cuadro llamado Commit changes
Un commit es una modificación del repositorio con algo que se añade/elimine/modifique, y dicho cuadro es recomendable usarlo para resumir en qué consiste la modificación, de manera que quede trazado el cambio.
Haciendo click en el reloj donde indica el número de commits accedemos al histórico de commits (cambios) con hora, día, autor, comentarios, etc.
Vamos a realizar un cambio en nuestro código: en tu código local (local –> tu ordenador), en lugar de filtrar por Madrid haz el filtro por Barcelona, guarda el código y sube en el repositorio el nuevo archivo (con el mismo nombre, Github hará la sobrescritura)
Si ahora consultamos el commit, al lado hay un número que lo identifica, y clickando en él nos resume los cambios: no solo almacena todas las versiones pasadas sino que además nos muestra las diferencias entre los archivos cambiados
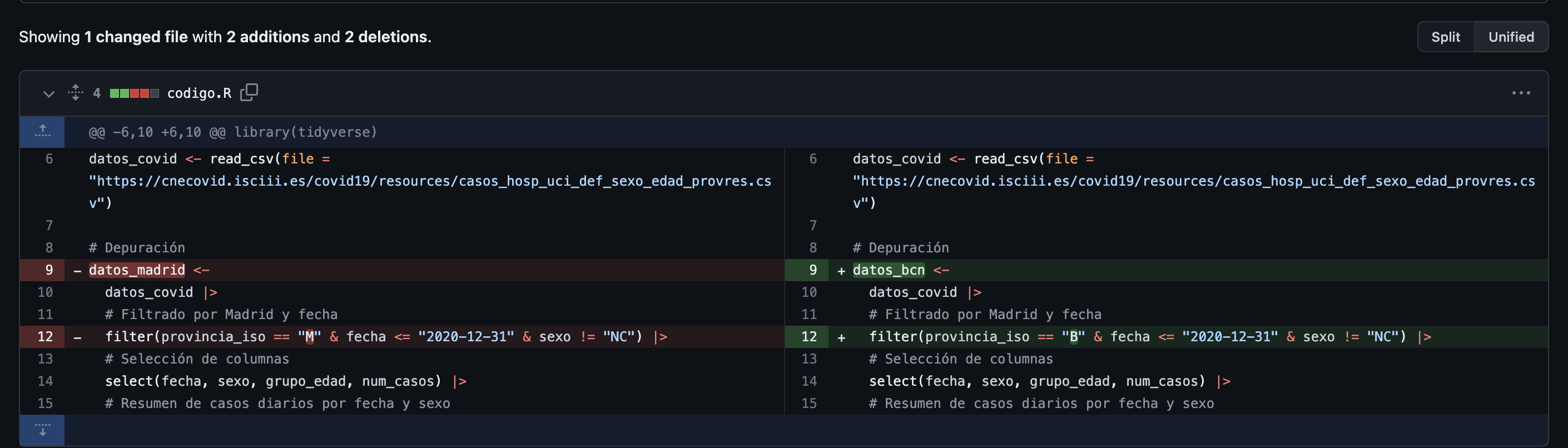
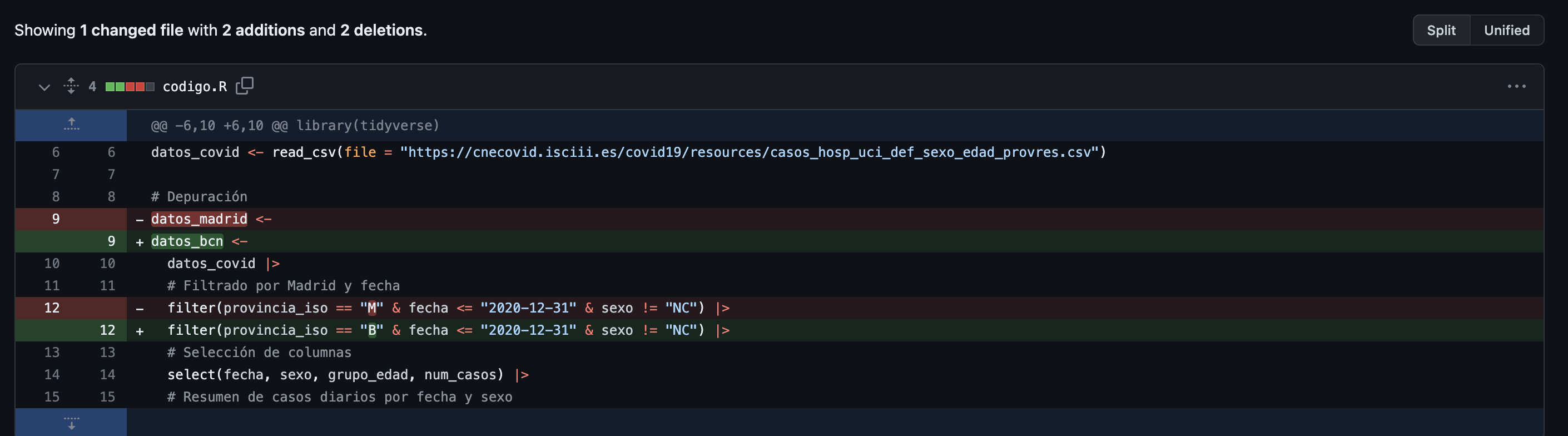
Tenemos dos modos de visualización de los cambios: el modo split nos muestra el antiguo y el nuevo, con las inclusiones en verde y lo que ya no está en rojo; y el modo unified nos muestra todo en un mismo documento.
Github nos permite incluso recuperar una versión del pasado de nuestro repositorio, haciendo click en el tercer icono del commit.
Si te fijas ahora al lado de 1 branch tenemos un menú desplegable en el que antes ponía main y ahora un número identificador del commit. Ya hablaremos de la idea de rama (branch)
Vamos a poner en práctica lo aprendido:
Crea un nuevo repositorio en Github (llamado repo-github-2) donde habrá alojado con proyecto de R.
Crea un proyecto en RStudio que se llame (por ejemplo) proyecto-qmd
Una vez dentro del proyecto en RStudio haz click en File < New File < Quarto Document

Deberás tener un documento similar a este: un quarto markdown (.qmd), un documento que nos permitirá incluir markdown + código (puede ser R o puede ser Observable, D3, etc).
Este formato es ideal para:

Si te fijas ahora nuestro repositorio tiene un archivo con formato .html…es decir…
¡Es una web!
¿Cómo convertir nuestro repositorio en una web?
SettingsPagesbranch selecciona la única rama que tenemos ahora (main).html (en web complejas estará como en cualquier web en docs, en algo simple estará en la ruta raiz del repositorio)SaveSi te fijas en la parte superior del repositorio ahora tenemos un icono naranja, que nos indica que la web está en proceso de ser desplegada (deploy)
Pasados unos segundos (dependiendo del tamaño de la web y tu conexión a internet) ese icono pasará a ser un check verde: habemus web
El link de la web por defecto será {nombre_usuario}.github.io/{nombre_repo}
¡Un momento! Ahora mismo nuestra web no nos está mostrando nuestro .qmd, sino por defecto el README.md.
Para que Github entienda que queremos visualizar ese .html que hemos generado a partir del .qmd vamos en nuestro proyecto local a borrar todo lo que no sea nuestro archivo .Rproj y nuestro archivo .qmd, y vamos a cambiar el nombre a este último llamándolo index.qmd, y volvemos a compilarlo para generar un index.html
Vamos a subir a Github ese nuevo proyecto con el cambio de nombre (llamado repo-github-3) para ver luego las diferencias entre uno y otro
Si repetimos el proceso para hacer una Page y esperamos al tick verde…
Si a tu .qmd ya le llamas de inicio index.qmd, automáticamente, al detectar Github un index.html, interpreta que ese archivo index.html es el que define la web (y puedes personalizar añadiendo un archivo css de estilos)
Habemus web simplemente clickando en Pages :)
Vamos a crear el último repositorio que se llamará repo-diapos, y crear un proyecto en RStudio del mismo nombre (por ejemplo). Una vez creado le daremos a File < New File < Quarto Presentation.
La forma de escribir será igual que un .qmd normal solo que ahora cada diapositiva la separaremos con un --- (usando archivos de estilos podemos personalizar lo que queramos)
Llama al archivo directamente index.qmd, súbelo a Github y con un click en Pages tienes una web con tus diapositivas
La forma más sencilla para trabajar de manera colaborativa en Github, y tenerlo sincronizado con nuestro local, es hacer uso de Gitkraken
Una vez dentro clickamos en el icono de la carpeta (Repo Management) y si ya tenemos el repositorio en Github seleccionamos Clone, indicando donde queremos clonar (en nuestro local) y que repositorio de Github queremos clonar.
Una vez clonado, la idea es que cada cambio que hagamos en local nos aparecerá en Gitkraken como View changes.
Cuando tengas suficientes cambios como para actualizar el repositorio (tampoco tiene sentido actualizar con cada edición), verás algo similar a esto con todos los commits realizados
Podrás decidir cuáles de los commits locales quieres incluir en remoto, bien uno a uno o en Stage all changes (para todos)
Tras incluir los commits deberás incluir un título y descripción del commit
Tras hacerlo verás que ahora tenemos dos iconos separados en una especie de árbol (¿te acuerdas de la branch o rama?):
Ordenador: la versión del repositorio que tienes en tu ordenador.
Logo: la versión del repositorio que tienes subida en remoto
Mientras eso suceda solo tendrás sincronizado tu ordenador con Gitkraken, pero no con Github. Para ello haremos click en Push (con Pull podrás forzar a tener en local lo mismo que en remoto).
Como hemos mencionado ya en varias ocasiones, hay un elefante en la habitación que aún no hemos mentado: las ramas o branchs de un repositorio.
Imagina que estáis trabajando varios en un proyecto y teneís una versión que funciona pero que queréis modificar en paralelo a partir del estado actual del repositorio.
Las ramas nos permiten partir de una versión común del repositorio y hacer cambios que no afecten a los demás
Para crear una rama a partir del estado actual de repositorio haremos click en Branch y le pondremos un nombre
Una vez creada verás dos iconos y un menú desplegable con las distintas ramas en las que quieres hacer el commit. Imagina que realizas un cambio pero no quieres añadirlo a la rama principal: puedes hacer el commit en tu rama propia en LOCAL (lo harás en la rama activa de tu menú de branchs).
La primera vez te pedirá que escribas la rama en REMOTO con la quieres sincronizar tu rama en local. Consejo: ponle el mismo nombre en remoto que en local.
Fíjate que ahora tenemos el ordenador y el logo en el mismo sitio. Esto no significa que tengas ambas ramas en tu local, solo que Gitkraken tiene ambas sincronizadas: clickando en cualquiera de ellas, tus archivos en tu ordenador cambiarán.
Lo más recomendable es que solo se incorpore de una rama secundaria a la rama principal aquello que está validado por un/a coordinador/a del repositorio, asegurándose que todo funciona correctamente.
Cuando queramos incluirlo haremos click con botón derecho en el icono de la rama secundaria y seleccionamos Start a pull request to origin from...
Una pull request será una petición al responsable de la rama principal para incluir los cambios
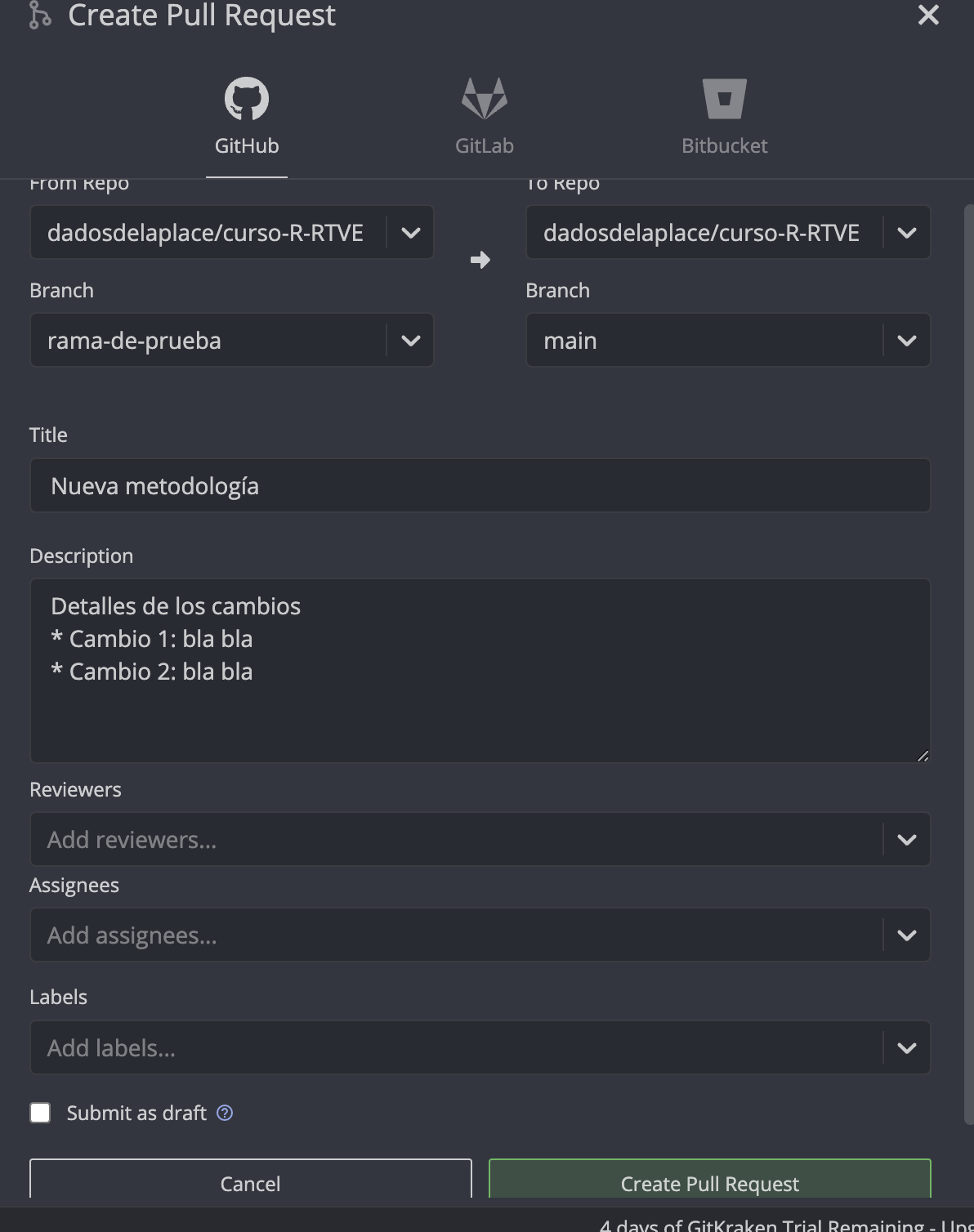
En el cuadro que no se abre deberemos escribir:
merge (normalmente la main)Mientras no se acepte aparecerá un icono de rama y un +1 en Pull Requests
Si somos al mantenedor del repositorio, haciendo click en el menú nos saldrán las ramas que nos quieren hacer hacer merge
Al hacer click se abrirá un cuadro de Pull Request para decidir si
mergemerge por si queremos solicitar algún cambio antes de ser aprobadoTras revisar todo y aprobarlo clickaremos en Confirm merge, y tras ello podremos decidir si esa rama que era paralela a la principal la queremos eliminar o dejar visible a todos (consejo: dejar visible para tene trazabilidad del proyecto de trabajo)
Javier Álvarez Liébana • Seminarios BioDS (UCM) • curso 2023-2024